Nye RNA-nanoenheder i levende celler kan fornemme og analysere flere komplekse signaler
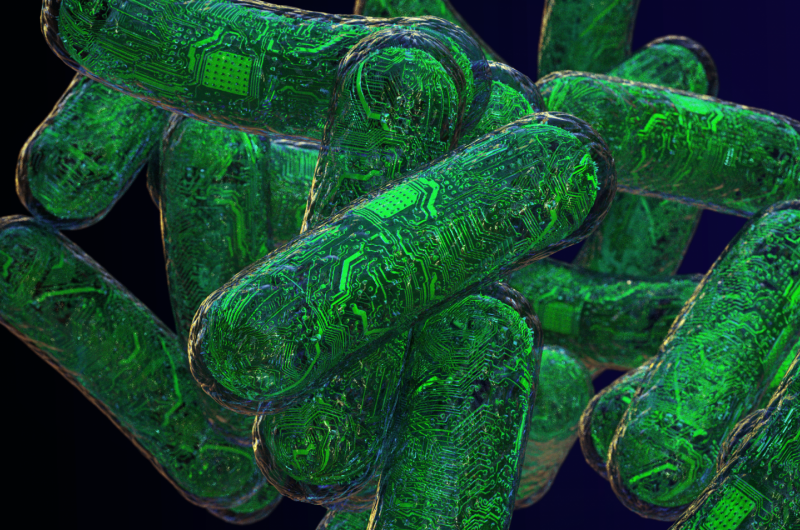
Ribonukleinsyre (RNA) bruges til at skabe logiske kredsløb, der er i stand til at udføre forskellige beregninger. I nye forsøg, Green og hans kolleger har inkorporeret RNA logiske porte i levende bakterieceller, der fungerer som små computere. Kredit:Jason Drees for Biodesign Institute
Den tværfaglige forbindelse mellem biologi og teknik, kendt som syntetisk biologi, vokser i hastigt tempo, åbner nye udsigter, som man næppe kunne forestille sig for kort tid siden.
I ny forskning, Alex Green, en professor ved ASU's Biodesign Institute, demonstrerer, hvordan levende celler kan induceres til at udføre beregninger på samme måde som små robotter eller computere.
Resultaterne af den nye undersøgelse har betydelige implikationer for intelligent lægemiddeldesign og smart lægemiddellevering, grøn energiproduktion, lavpris diagnostiske teknologier og endda udviklingen af futuristiske nanomaskiner, der er i stand til at jage kræftceller eller slukke for afvigende gener.
"Vi bruger meget forudsigelige og programmerbare RNA-RNA-interaktioner til at definere, hvad disse kredsløb kan gøre, " siger Green. "Det betyder, at vi kan bruge computersoftware til at designe RNA-sekvenser, der opfører sig, som vi vil have dem til i en celle. Det gør designprocessen meget hurtigere."
Undersøgelsen vises i den forskudte onlineudgave af tidsskriftet Natur .
Designer RNA
Den beskrevne fremgangsmåde bruger kredsløb sammensat af ribonukleinsyre eller RNA. Disse kredsløbsdesign, som ligner konventionelle elektroniske kredsløb, selvsamler i bakterieceller, giver dem mulighed for at fornemme indkommende beskeder og reagere på dem ved at producere et bestemt beregningsoutput, (I dette tilfælde, et protein).
I den nye undersøgelse, specialiserede kredsløb kendt som logiske porte blev designet i laboratoriet, derefter inkorporeret i levende celler. De små kredsløbsafbrydere udløses, når beskeder (i form af RNA-fragmenter) binder sig til deres komplementære RNA-sekvenser i det cellulære kredsløb, aktivering af den logiske port og frembringelse af et ønsket output.
RNA-switcherne kan kombineres på forskellige måder for at producere mere komplekse logiske porte, der er i stand til at evaluere og reagere på flere input, ligesom en simpel computer kan tage flere variabler og udføre sekventielle operationer som addition og subtraktion for at nå et endeligt resultat.
Den nye undersøgelse forbedrer dramatisk den lethed, hvormed cellulær databehandling kan udføres. Den eneste RNA-tilgang til fremstilling af cellulære nanoenheder er et betydeligt fremskridt, da tidligere indsats krævede brug af komplekse mellemled, som proteiner. Nu, de nødvendige ribocomputing dele kan let designes på computer. De simple baseparringsegenskaber af RNA's fire nukleotidbogstaver (A, C, G og U) sikrer den forudsigelige selvsamling og funktion af disse dele i en levende celle.
Greens arbejde på dette område begyndte på Wyss Institute på Harvard, hvor han hjalp med at udvikle den centrale komponent, der blev brugt i de cellulære kredsløb, kendt som en RNA-tåholdskontakt. Arbejdet blev udført, mens Green var post-doc og arbejdede med nanoteknologieksperten Peng Yin, sammen med de syntetiske biologer James Collins og Pamela Silver, som alle er medforfattere på det nye papir. "De første eksperimenter var i 2012, " siger Green. "Dybest set, tåholdskontakterne fungerede så godt, at vi ønskede at finde en måde at udnytte dem bedst muligt til mobilapplikationer."
Efter ankomsten til ASU, Greens første kandidatstuderende Duo Ma arbejdede på eksperimenter på Biodesign Institute, mens en anden postdoc, Jongmin Kim fortsatte lignende arbejde på Wyss Institute. Begge er også medforfattere til det nye studie.
Naturens Pentium-chip
Muligheden for at bruge DNA og RNA, livets molekyler, at udføre computerlignende beregninger blev første gang demonstreret i 1994 af Leonard Adleman fra University of Southern California. Siden da, hurtige fremskridt har fremmet området betydeligt, og for nylig, sådan molekylær beregning er blevet udført i levende celler. (Bakterieceller anvendes normalt til dette formål, da de er enklere og nemmere at manipulere.)
Teknikken beskrevet i det nye papir udnytter det faktum, at RNA, i modsætning til DNA, er enkeltstrenget, når det produceres i celler. Dette giver forskere mulighed for at designe RNA-kredsløb, der kan aktiveres, når en komplementær RNA-streng binder sig til en blotlagt RNA-sekvens i det designede kredsløb. Denne binding af komplementære strenge er regelmæssig og forudsigelig, hvor A nukleotider altid parrer med U og C parrer altid med G.
Med alle bearbejdningselementerne i kredsløbet lavet ved hjælp af RNA, som kan påtage sig et astronomisk antal potentielle sekvenser, den reelle kraft af den nyligt beskrevne metode ligger i dens evne til at udføre mange operationer på samme tid. Denne kapacitet til parallel behandling tillader hurtigere og mere sofistikeret beregning, samtidig med at cellens begrænsede ressourcer udnyttes effektivt.
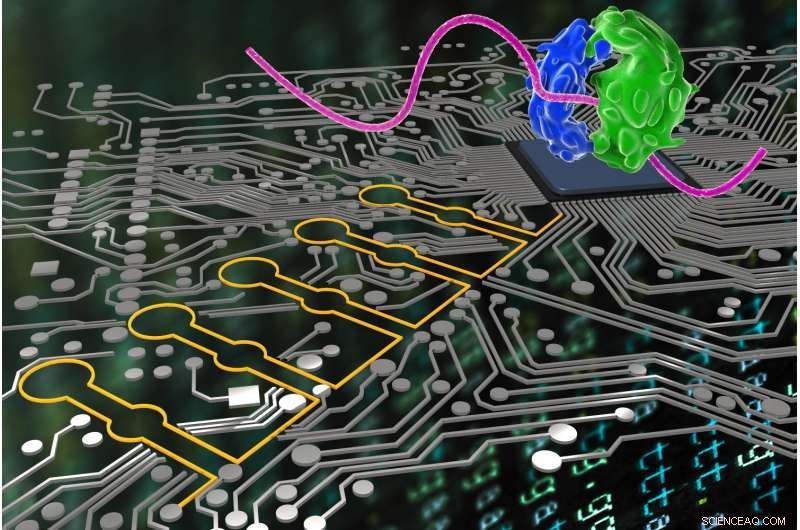
Svarende til hvordan dataloger bruger logisk sprog til at få deres programmer til at gøre nøjagtige OG, ELLER og IKKE beslutninger mod et endeligt mål, "Ribocomputing Devices" (stiliseret her i gult) udviklet af et team på Wyss Institute kan nu bruges af syntetiske biologer til at fornemme og fortolke flere signaler i celler og logisk instruere deres ribosomer (stiliseret i blåt og grønt) til at producere forskellige proteiner. Kredit:Wyss Institute ved Harvard University
Logiske resultater
I den nye undersøgelse, logiske porte kendt som OG, OR og NOT blev designet. En OG-port producerer kun et output i cellen, når to RNA-meddelelser A OG B er til stede. En ELLER-port reagerer på enten A ELLER B, mens en NOT-port vil blokere output, hvis en given RNA-input er til stede. Kombination af disse porte kan producere kompleks logik, der er i stand til at reagere på flere input.
Brug af RNA-tåholdskontakter, forskerne producerede de første ribocomputing-enheder, der var i stand til fire-input OG, seks-input OR og en 12-input enhed i stand til at udføre en kompleks kombination af OG, OR- og IKKE-logik kendt som disjunktiv normal formudtryk. Når den logiske port støder på de korrekte RNA-bindingssekvenser, der fører til aktivering, en tåholdskontakt åbnes, og oversættelsesprocessen til protein finder sted. Alle disse kredsløbsfølende og outputfunktioner kan integreres i det samme molekyle, gør systemerne kompakte og nemmere at implementere i en celle.
Forskningen repræsenterer den næste fase af det igangværende arbejde med de meget alsidige RNA-tåholdskontakter. I tidligere arbejde, Green og hans kolleger viste, at en billig, papirbaseret række af RNA-tåholdskontakter kunne fungere som en meget præcis platform til diagnosticering af Zika-virus. Påvisning af viralt RNA af arrayet aktiverede tåholdskontakterne, udløser produktion af et protein, som er registreret som en farveændring på arrayet.
Det grundlæggende princip med at bruge RNA-baserede enheder til at regulere proteinproduktionen kan anvendes på stort set alle RNA-input, indvarsler en ny generation af præcise, lavprisdiagnostik for en bred vifte af sygdomme. Den cellefri tilgang er særlig velegnet til nye trusler og under sygdomsudbrud i udviklingslandene, hvor medicinske ressourcer og personale kan være begrænsede.
Computeren indeni
Ifølge Green, den næste fase af forskningen vil fokusere på brugen af RNA-tåholdsteknologien til at producere såkaldte neurale netværk i levende celler - kredsløb, der er i stand til at analysere en række excitatoriske og hæmmende input, gennemsnittet af dem og produceret et output, når en bestemt aktivitetstærskel er nået, på samme måde som en neuron gennemsnittet indkommende signaler fra andre neuroner. Ultimativt, forskere håber at få celler til at kommunikere med hinanden via programmerbare molekylære signaler, danner en virkelig interaktiv, hjernelignende netværk.
"Fordi vi bruger RNA, et universelt livsmolekyle, vi ved, at disse interaktioner også kan fungere i andre celler, så vores metode giver en generel strategi, der kan overføres til andre organismer, Grøn siger, hentyder til en fremtid, hvor menneskelige celler bliver fuldt programmerbare enheder med omfattende biologiske evner.
 Varme artikler
Varme artikler
-
 Team udvikler verdens mest kraftfulde mikrobølgeoscillatorer i nanoskalaSkematisk repræsentation af en spin-transfer nano-oscillator (STNO) med frie og fastgjorte magnetiske lag (venstre), og et scanning-elektron-mikroskopi (SEM) billede af et tværsnit af en STNO (højre),
Team udvikler verdens mest kraftfulde mikrobølgeoscillatorer i nanoskalaSkematisk repræsentation af en spin-transfer nano-oscillator (STNO) med frie og fastgjorte magnetiske lag (venstre), og et scanning-elektron-mikroskopi (SEM) billede af et tværsnit af en STNO (højre), -
 Nanopartikler til at dræbe kræftceller med varmeJernoxidnanopartikler med en pænt stablet indre struktur (venstre) har brug for et stærkere magnetfelt end forventet for at varme op, mens dem med et mere tilfældigt arrangement opvarmes hurtigere, se
Nanopartikler til at dræbe kræftceller med varmeJernoxidnanopartikler med en pænt stablet indre struktur (venstre) har brug for et stærkere magnetfelt end forventet for at varme op, mens dem med et mere tilfældigt arrangement opvarmes hurtigere, se -
 Hurtigere fotoner kunne muliggøre total datasikkerhedElektrisk indstillelig on-demand on-chip enkelt foton kilde aktiveret af en stærk Purcell effekt. Kredit:John OHara Forskere ved University of Sheffield har løst et nøglepuslespil i kvantefysik, d
Hurtigere fotoner kunne muliggøre total datasikkerhedElektrisk indstillelig on-demand on-chip enkelt foton kilde aktiveret af en stærk Purcell effekt. Kredit:John OHara Forskere ved University of Sheffield har løst et nøglepuslespil i kvantefysik, d -
 Formkodet dynamisk samling af mobile mikromaskinerFormkodet samling af magnetiske mikroaktuatorer i form af et mikrokøretøj. Kredit:Nature Materials, doi:10.1038/s41563-019-0407-3 Feltstyret og selvkørende kolloid samling kan bruges til at bygge
Formkodet dynamisk samling af mobile mikromaskinerFormkodet samling af magnetiske mikroaktuatorer i form af et mikrokøretøj. Kredit:Nature Materials, doi:10.1038/s41563-019-0407-3 Feltstyret og selvkørende kolloid samling kan bruges til at bygge
- Virkningerne af industrialisering på dyr
- Hvordan kreolske sprog udvikler sig kan afhænge af, hvordan folk begyndte at bruge dem
- Selvisolering er umuligt for tusindvis af newzealændere, medmindre vi hjælper dem med at faste
- Ny usynlighedskappe skjuler genstande fra menneskesyn
- Lavinestatistikker tyder på, at Tabbys stjerne er tæt på en kontinuerlig faseovergang
- Portugisiske brandmænd får overtaget mod skovbrande (Opdatering)


