Binder knude:Nye DNA -nanostrukturer
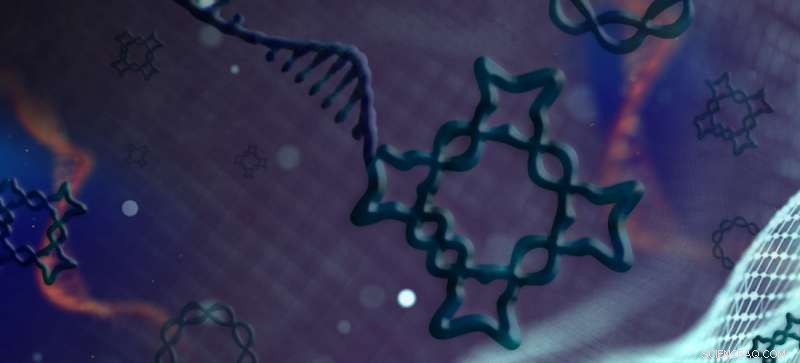
Hao Yan og hans kolleger designede og konstruerede en række knyttede former, (hver måler kun milliarder af en meter i længden), fra en enkelt streng af DNA. De selvsamlende nanostrukturer følger en præcis, forprogrammeret foldeordre. Kredit:Shireen Dooling
Knuder er uundværlige værktøjer til menneskelige aktiviteter som sejlads, fiskeri og klatring, (for ikke at nævne, binde sko). Men ved at knytte en knude i en lacelike DNA -streng, måler kun milliarddeler af en meter i længden, kræver tålmodighed og højt specialiseret ekspertise.
Hao Yan, en forsker på ASU, er en øvet hånd i dette sarte og eksotiske felt, opererer ved krydset mellem nanoteknologi og kunst.
I ny forskning, der vises i tidsskriftet Naturkommunikation , Yan og hans kolleger Fei Zhang, Xiaodong Qi og andre beskriver en metode til at lokke segmenter af enkeltstrenget DNA til komplekse 2- og 3-D-knyttede strukturer.
Resultaterne repræsenterer et vigtigt fremskridt inden for det hurtige område inden for DNA-nanoteknologi, hvor livsmolekylet bruges som et strukturelt byggemateriale til en lang række små konfigurationer. Blandt disse er minimale robotudstyr, fotoniske applikationer, lægemiddelleveringssystemer, logiske porte, såvel som diagnostiske og terapeutiske anvendelser.
"De knyttede DNA -strukturer demonstreret i dette arbejde udviser topologisk kompleksitet uden fortilfælde, langt ud over hvad der er opnået før man brugte enkeltstrenget folde, "Siger Yan." Faktisk, det er ikke kun fantastisk, men også overraskende, at det enkeltstrengede DNA og RNA kan trænge igennem sine egne kæder og finde en måde at danne så stærkt knyttede strukturer på, i betragtning af, at den enkelte streng skal væve igennem så mange floker. "
Yan leder Biodesign Center for Molecular Design and Biomimetics og er Martin D. Glick Distinguished Professor på School of Molecular Sciences ved ASU.
At bringe DNA ind i folden
Den nye undersøgelse involverer innovationer inden for DNA -origami, hvilken, som navnet antyder, bruger nukleinsyrer som DNA og RNA til at folde og samle sig selv i komplekse former. Dette sker, når komplementære nukleotidbaser i DNA's 4-bogstavs alfabet kommer i kontakt og binder, ifølge et strengt regime:C -baser parres altid med G og A -baser parres altid med T.
I naturen, strenge af nukleinsyrer giver den kode, der er nødvendig for at lave komplekse proteiner. Denne grundlæggende biologi danner grundlaget for alt jordisk liv. Udnytter de simple baseparringsegenskaber ved DNA, det er muligt at designe strukturer, der vil samle sig selv i laboratoriet. Metoden er blevet anvendt på både enkeltstrengede og dobbeltstrengede DNA-former, resulterer i nanostrukturer med stigende kompleksitet og raffinement.
Mens DNA origami har gjort opsigtsvækkende fremskridt siden starten, en teknisk innovation har været irriterende svær at opnå. Indtil nu, at skabe komplekse knyttede strukturer i DNA på en forudsigelig og programmerbar måde har undgået forskere.
Det nye arbejde overvinder denne forhindring, etablering af præcise designregler, der tillader enkeltstrengede segmenter af DNA (eller RNA), der spænder fra 1800-7500 nukleotider til dannelse af knudelignende nanostrukturer med krydsende tal (hvor DNA-strengen væver ind og ud af sin egen længde), der spænder fra 9 til 57 .
Gruppen demonstrerede endvidere, at disse nukleinsyrenanostrukturer kan replikeres og amplificeres, både under laboratorieforhold og inden for levende systemer.

Hao Yan leder Biodesign Center for Molecular Design and Biomimetics og er Martin D. Glick Distinguished Professor på School of Molecular Sciences ved ASU. Kredit:Biodesign Institute
Naturens knuder
Knyttede strukturer, ligesom dem Yan har fremstillet, (men meget enklere end de syntetiske), har korrelater i den naturlige verden. De er blevet observeret i DNA og proteiner og dannes generelt under replikation og transkription, (når en DNA -sekvens kopieres til messenger -RNA). De kan også forekomme i fagenes genomer - vira, der inficerer bakterieceller.
Alligevel, konstruktion af molekylære knuder i nanometerskalaen, at vise veldefinerede og konsistente geometrier kræver enorm kontrol og præcision. Som det sker, nukleinsyrer som DNA er ideelle til design og syntese af sådanne molekylære knuder.
Tidligere har længder af dobbeltstrenget DNA er blevet brugt til nanoskala konstruktioner, med tilføjelse af korte stykker eller "hæfteklammer" for at fastgøre resulterende strukturer sammen. Den nye undersøgelse bruger i stedet en enkelt DNA -længde designet til at vikle sig rundt i en præcis, forprogrammeret sekvens af trin.
Når de knyttede DNA -nanostrukturer med succes samler sig selv, de er afbildet ved hjælp af atomkraftmikroskopi. Omhyggelig beregning giver forskerne mulighed for at optimere foldebanerne for at producere det højeste udbytte for hver syntetisk struktur. Anvendelsen af enkelt- snarere end dobbeltstrenget DNA gør det muligt at producere strukturerne i overflod til meget lavere omkostninger.
En enkeltstrenget tilgang åbner døren for design af nanoarchitectures med specifikke, veldefinerede funktioner, som kan produceres gennem på hinanden følgende runder med in vitro -evolution, hvor de ønskede attributter vælges til i en gentagen proces med forfining. Yderligere, den fremgangsmåde, der er skitseret i den nye undersøgelse, giver en generel platform for design af molekylære strukturer af øget størrelse og hidtil uset kompleksitet, baner vejen for fremskridt inden for nanofotonik, levering af medicin, cryo-EM analyse og DNA-baseret hukommelseslagring.
Designer DNA (og RNA)
For et af de første knudesigner var den strategi, Yan og hans kolleger udviklede, involveret at tråde en enkelt streng af DNA eller RNA gennem sig selv 9 gange i henhold til en forprogrammeret sekvens, demonstrerer, at den nye metode er i stand til at producere indviklede geometriske former, der er programmerbare, replikerbar og skalerbar.
Designstrategien blev efterfølgende udvidet til at omfatte enkeltstrengede RNA-strukturer og 3D-knuder, hvis former blev rekonstrueret ved hjælp af en teknik kendt som kryogen transmissionselektronmikroskopi, bekræfter deres korrekte foldning i de ønskede former.
"En af udfordringerne i dette arbejde er, hvordan man kan øge samleudbyttet af stærkt knyttede strukturer." Sagde Fei. I modsætning til klassiske DNA -nanostrukturer, de enkeltstrengede knuder er mindre tilgivende med hensyn til præcis folderækkefølge på grund af den topologiske kompleksitet. Hvis en enkelt krydsning foldes forkert under processen, fejlen vil næppe blive selvkorrigeret, og de fleste fejlfoldninger vil forblive i den færdige struktur. "Vi udviklede en hierarkisk foldestrategi til at styre den korrekte knobdannelse. Vi sammenlignede en knuds foldningseffektivitet med 23 krydsninger ved hjælp af forskellige foldningsveje. AFM-billederne viste en dramatisk stigning i foldudbyttet af velformede strukturer fra 0,9 % til 57,9% ved at anvende optimeret hierarkisk foldevej. " Fei tilføjet.
De designregler, der bruges til at optimere foldebanerne, er baseret på antallet af krydsningspunkter, længden af DNA og antallet af basepar i den designede struktur. Tre primære regler blev fastlagt. Først, lineære foldestier viste sig at være at foretrække frem for forgrenede stier. Sekund, den udfoldede sektion af en DNA -streng bør ikke trænge igennem sig selv i de tidlige stadier, når strengen stadig er lang. Endelig, kanter af den ønskede form, der har tre krydsninger, skal foldes før dem med to krydsninger.
Efter designstrategien, holdet var i stand til at skabe mere komplekse DNA -knuder med stigende krydsningstal.
Længere kæder af enkeltstrenget DNA udgør unikke udfordringer for at designe programmerede nanostrukturer på grund af den øgede sandsynlighed for utilsigtet selvkomplementering af de baser, der udgør kæden. En DNA -knudestruktur med 57 krydsede noder, der er samlet vellykket, dog med lavere udbytte og mindre præcision. Da krydsetallet blev øget til 67, udbyttet faldt betydeligt og de resulterende strukturer, billedet af AFM, viste flere monteringsfejl.
Undersøgelsen rapporterer de største DNA -knuder, der endnu er samlet, dannet fra op til 7,5k baser, med de mest komplicerede topologier, med op til 57 krydsende regioner. De enkeltstrengede DNA-sekvenser kan masseproduceres i levende celler for større effektivitet til lavere omkostninger. Ultimativt, DNA -nanostrukturer med forskellig funktion kan dannes i celler, innovationer, der skal forfølges i det fremtidige arbejde.
 Varme artikler
Varme artikler
-
 Grafen brugt som frekvensmixer i ny forskningTop:Tre vibrationstilstande spændende i cirkulære tromler. I grafen, disse tilstande er koblet til hinanden og kan udveksle energi. Nederst:En kunstners skildring af en grafentromme. Grafenen vibrerer
Grafen brugt som frekvensmixer i ny forskningTop:Tre vibrationstilstande spændende i cirkulære tromler. I grafen, disse tilstande er koblet til hinanden og kan udveksle energi. Nederst:En kunstners skildring af en grafentromme. Grafenen vibrerer -
 Forskere udvikler hårdere keramik til panservinduerSpinelvinduer kan bruges som elektro-optiske/infrarøde dækshusvinduer i den nye klasse af US Navy destroyere, ligesom USS Elmo Zumwalt på billedet ovenfor, der har en lav radarsignatur sammenlignet me
Forskere udvikler hårdere keramik til panservinduerSpinelvinduer kan bruges som elektro-optiske/infrarøde dækshusvinduer i den nye klasse af US Navy destroyere, ligesom USS Elmo Zumwalt på billedet ovenfor, der har en lav radarsignatur sammenlignet me -
 Enkeltmolekyle elektronik og kemisk lodning(PhysOrg.com) -- Enkeltmolekyleelektronik er en division af nanoteknologi, der bruger enkelte molekyler som elektroniske komponenter, og dens undersøgelse har det ultimative mål at reducere størrelsen
Enkeltmolekyle elektronik og kemisk lodning(PhysOrg.com) -- Enkeltmolekyleelektronik er en division af nanoteknologi, der bruger enkelte molekyler som elektroniske komponenter, og dens undersøgelse har det ultimative mål at reducere størrelsen -
 Finjustering af twist mellem 2-D materialer i van der Waals heterostrukturer for at hjælpe med at a…Dette billede viser tegnefilm og mikrografik, der fremhæver den nye teknik med in situ twistronics. Kredit:Artem Mishchenko/The University of Manchester En gruppe internationale forskere ved Unive
Finjustering af twist mellem 2-D materialer i van der Waals heterostrukturer for at hjælpe med at a…Dette billede viser tegnefilm og mikrografik, der fremhæver den nye teknik med in situ twistronics. Kredit:Artem Mishchenko/The University of Manchester En gruppe internationale forskere ved Unive
- Vidnesbyrd om mennesker på Azorernes øhav 700 år tidligere end antaget
- En asteroide summede lige forbi Jorden, og vi bemærkede knap i tide
- Forsinket internetprojekt i Kentucky står over for et nyt egern -tilbageslag
- Forskere finder vridende 3-D raceway for elektroner i nanoskala krystalskiver
- CT-scanningsundersøgelse gør det muligt at 3D-udskrive kraniet af dinosaurarten massospondylus
- Genekspression i Prokaryotes


