Forskere, der først simulerede en storstilet virus, M13
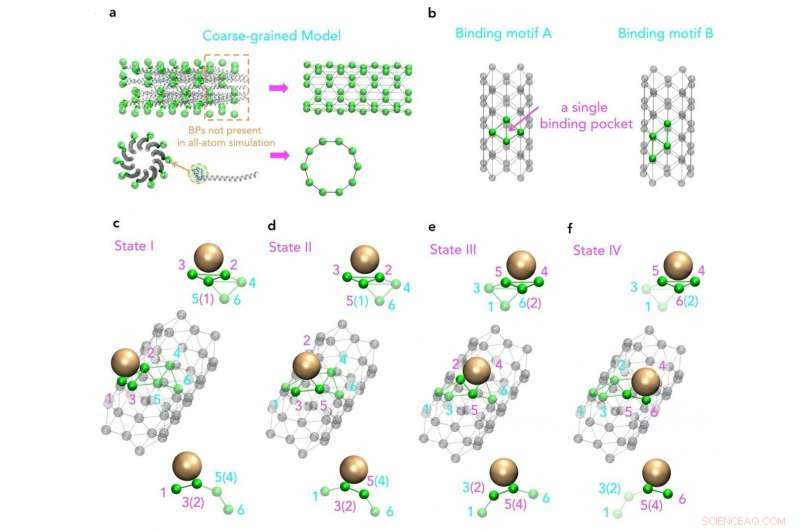
Molekylær visning af en grovkornet model baseret på den oprindelige struktur af M13 hovedpelsproteiner Kredit:SUTD
Atomistiske simuleringer er et kraftfuldt værktøj til at studere bevægelser og interaktioner mellem atomer og molekyler. I mange biologiske processer, storstilede effekter, for eksempel, samling af store vira til nanopartikler er vigtig. Samlingsprocessen for disse store vira er af fundamental betydning for designet af mange enheder og virale protein-målrettede terapeutika. Imidlertid, tids- og længdeskalaen for disse samlingsprocesser er normalt for store til simuleringer ved molekylær opløsning.
I øvrigt, selvom en stigning i computerkraft giver mulighed for mere komplekse og længere simuleringer, virus strukturer, såsom M13, stadig er uden for rækkevidde. Derfor har en forskergruppe fra Singapore University of Technology and Design (SUTD) og Massachusetts Institute of Technology (MIT) udviklet en procedure, der forbinder storskala montageprocesser med molekylære simuleringer. Assistent Prof Desmond Loke fra SUTD's Science, Matematik og teknologiklynge sagde, "Til simulering af M13, vi startede med forskellige sæt kraftfelter. Egnede kraftfelter blev valgt, og de blev brugt som input til en molekyle dynamik simuleringer med grovkornet model designet til at fange nøglemønsteret i samlingsprocessen."
"Selvom vi ved, at M13-baseret fremstilling grundlæggende kan være drevet af nanopartikel-peptid-interaktioner, hvilket også kan være et nøgleprincip bag M13-type bioteknik, vi har ringe viden om, hvordan gentagne mønstre af short-end-peptider på en M13-overflade faktisk er involveret i disse interaktioner. For at studere dette, Vi skal ideelt set inkludere en fuld struktur af det virale kappeprotein, hvilket er en vanskelig opgave for nuværende state-of-the-art simuleringer af molekylær dynamik, " tilføjer Dr. Lunna Li, første forfatter til artiklen.
Proceduren giver brugerne mulighed for at tilføje forskellige typer nanopartikler til en opløsning, på et realistisk niveau. Inspireret af denne procedure, Adjunkt Loke og hans kolleger var i stand til at simulere en storstilet virus med nanopartikler og inde i en opløsning i halvtreds nanosekunder.
Dr. Li sagde, "Virusstrukturen og -løsningen indeholder omkring 700, 000 atomer samlet." I betragtning af formen og størrelsen af funktionerne, kompleksiteten af denne simulering kan være større end nogen tidligere udført simulering.
"En simulering udført i mikrosekunder ville have været mulig, hvis en mindre M13-model blev brugt, men det kan være umagen værd at reducere tiden til rent faktisk at observere, hvordan den fulde struktur kan påvirke samlingen mellem M13 og nanopartikler, " forklarede assistent prof Loke.
 Varme artikler
Varme artikler
-
 Ballistisk transport i grafen antyder en ny type elektronisk enhedDette er en konceptuel tegning af et elektronisk kredsløb bestående af indbyrdes forbundne grafen nanobånd (sorte atomer), der er epitaksialt dyrket på trin ætset i siliciumcarbid (gule atomer). Elekt
Ballistisk transport i grafen antyder en ny type elektronisk enhedDette er en konceptuel tegning af et elektronisk kredsløb bestående af indbyrdes forbundne grafen nanobånd (sorte atomer), der er epitaksialt dyrket på trin ætset i siliciumcarbid (gule atomer). Elekt -
 Halvleder qubits skalerer i to dimensionerSkematisk af fire-qubit kvanteprocessoren lavet ved hjælp af halvlederfremstillingsteknologi. Kredit:Nico Hendrickx (QuTech) CPUer er bygget ved hjælp af halvlederteknologi, som er i stand til at
Halvleder qubits skalerer i to dimensionerSkematisk af fire-qubit kvanteprocessoren lavet ved hjælp af halvlederfremstillingsteknologi. Kredit:Nico Hendrickx (QuTech) CPUer er bygget ved hjælp af halvlederteknologi, som er i stand til at -
 Grafen deltager i kapløbet om at omdefinere amperenKredit:University of Manchester En ny fælles innovation fra National Physical Laboratory (NPL) og University of Cambridge kan bane vejen for at omdefinere ampere i form af fundamentale fysikkonsta
Grafen deltager i kapløbet om at omdefinere amperenKredit:University of Manchester En ny fælles innovation fra National Physical Laboratory (NPL) og University of Cambridge kan bane vejen for at omdefinere ampere i form af fundamentale fysikkonsta -
 Plankton inspirerer til skabelse af stealth-panser til mikroskopiske lægemiddelkøretøjer med lang…Fire forskellige rustningstyper tilføjet af forskerne fra University of Warwick (PhysOrg.com) -- Nogle former for plankton og bakteriers evne til at bygge et ekstra naturligt lag af nanopartikelli
Plankton inspirerer til skabelse af stealth-panser til mikroskopiske lægemiddelkøretøjer med lang…Fire forskellige rustningstyper tilføjet af forskerne fra University of Warwick (PhysOrg.com) -- Nogle former for plankton og bakteriers evne til at bygge et ekstra naturligt lag af nanopartikelli
- Solcreme og kosmetikforbindelser kan skade koraller ved at ændre fedtsyrer
- Tilbage til rumvuggen:ESA-astronauter igangværende eksperimenter i ISS
- At få folk til at lytte til hinanden reducerer ulighed og forbedrer gruppens præstationer
- Ny metode til at levere terapeutiske lægemidler ved hjælp af aflange nanokrystaller
- Forskning understøtter ny tilgang til minegenvinding
- Detektering af et hidtil uset udvalg af potentielt skadelige luftbårne forbindelser


