Løftet om nye antibiotika ligger i at binde små giftige tøjler til bakterier
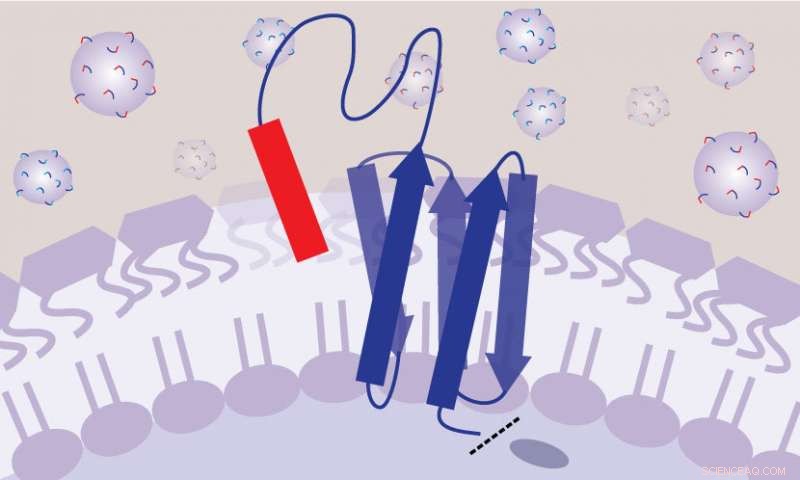
I SLAY -metoden, hver bakterie er gensplejset til at producere et molekyle på dens celleoverflade, som dels er peptid og dels tøjring - som en legepladstetherball. Dette arrangement gør det muligt for peptiderne at efterligne fritflydende lægemidler i den menneskelige krop. Kredit:Ashley Tucker
Biologer ved University of Texas i Austin har udviklet en metode til hurtigt at screene hundredtusindvis af potentielle lægemidler til bekæmpelse af infektioner, en innovation, der lover at bekæmpe den voksende plage af antibiotika-resistente bakterier. Metoden involverer manipulering af bakterier til at producere og teste molekyler, der er potentielt giftige for dem selv.
En beskrivelse af metoden findes i den trykte udgave af tidsskriftet den 25. januar Celle .
Ingen ny klasse af antibiotika er blevet opdaget i 40 år - mange af de mest tilgængelige i naturen er allerede fundet, og processen med at oprette og teste nye fra bunden er langsom og besværlig - men moderne medicin har stort brug for dem. Ifølge Verdenssundhedsorganisationen antibiotika har tilføjet omkring 20 år til den gennemsnitlige menneskelige levetid. Men deres beskyttende fordele glider væk, efterhånden som bakterier udvikler antibiotikaresistens.
I deres proof of concept, UT Austin-holdet, ledet af Bryan Davies, vist omkring 800, 000 molekyler kaldet peptider for at se, om de havde antimikrobielle virkninger, hvilket betyder, at de dræbte skadelige bakterier. Af disse, flere tusinde dræbte E. coli bakterier, gør dem til potentielle fører til antibiotika. Nogle antibiotika, der i øjeblikket er i brug, er peptider. Opfølgende forskning vil være nødvendig for at afgøre, hvilken hvis nogen, af de tusindvis af nye kundeemner er virkelig effektive og sikre hos mennesker, men forskerne viste, at mindst et sådant molekyle, døbt P7, dræber også andre former for patogene bakterier og er sikkert hos mus.
Med denne metode, kaldet SLAY (Surface Localized Antimicrobial Display), én person kan screene hundredtusindvis af lignende peptider hurtigere og mere omkostningseffektivt, end eksisterende metoder kan. Davies ser gerne, at metoden bliver et standardværktøj i den globale jagt på nye antibiotika.
"Så hvad nu hvis vi har tusind grupper, der alle bruger dette system til at følge deres egne interesser og deres egne peptider?" sagde Davies, adjunkt i molekylær biovidenskab. "Når du aktiverer et fællesskab af den størrelse, så tror jeg, at du har større chancer for rent faktisk at finde et nyt antibiotikum, der virker."
Et vigtigt fremskridt i dette arbejde var at finde ud af, hvordan man får bakterier til at producere molekyler, der kan være giftige for dem selv, og at kontrollere, hvordan disse molekyler interagerer med deres værtsbakterier.
"Vi troede, ville det ikke være fantastisk, hvis en bakterie kunne syntetisere forbindelsen for os, fordi bakterier er billige og nemme at dyrke, og test derefter stoffet på sig selv og meld tilbage og fortæl os, var det et antimikrobielt middel eller ej?" sagde Davies.
Deres løsning var at genetisk manipulere bakterierne til at producere et molekyle på celleoverfladen, der er en del af et peptid og en del tether - som en legepladsbinding og dens tether - med den ene ende fastgjort til cellemembranen og den anden ende fri til at flyde rundt. Dette gør det muligt for peptidet at bevæge sig rundt og komme i kontakt med den bakterielle celleoverflade, som om det var frit svævende som et stof i din blodbane, men uden at interagere med andre nærliggende bakterier.
Ved at sikre, at hver version af bindebolden kun interagerer med de bakterier, der producerede den, forskerne kunne derefter gøre et stort spring i effektivitet. De kunne skabe hundredtusindvis af stammer af bakterier – hver gensplejset til at producere en lidt anderledes version af tøjlen – og sætte alle disse stammer i det samme reagensglas for at vokse. Ved at køre hundredtusindvis af eksperimenter samtidigt, deres metode sparer enormt meget plads, tid og omkostninger.
En del af denne proces er baseret på en teknik udviklet af UT Austins George Georgiou i 1990'erne, der får bakterier til at producere proteiner eller peptider på deres overflader.
For at finde ud af, hvilke bindebolde (peptider) der slår deres værter ud, forskerne bruger gensekventering til at identificere, hvilke versioner der produceres af bakterier i starten, og hvilke der produceres i slutningen.
Efter opdagelsen af, at P7 dræber patogener, holdet planlægger nu at skabe tusindvis af subtile variationer af dette molekyle, kaldet derivater, og kør dem gennem den samme screeningsproces for at søge efter en endnu mere effektiv version.
Postdoc-stipendiat Ashley Tucker ledede det eksperimentelle arbejde for at demonstrere brugen af platformen.
Davies, Tucker og UT Austin har indgivet patentansøgninger for SLAY-metoden og for de specifikke genetiske sekvenser for de tusindvis af antimikrobielle peptider, de har opdaget indtil videre.
 Varme artikler
Varme artikler
-
 At undersøge kartoflers fortid kunne forbedre fremtidens spudsUndersøgelse af det modernes forfædre, Nordamerikanske dyrkede kartofler har afsløret et sæt almindelige gener og vigtige genetiske veje, der har hjulpet spuds med at tilpasse sig gennem tusinder af å
At undersøge kartoflers fortid kunne forbedre fremtidens spudsUndersøgelse af det modernes forfædre, Nordamerikanske dyrkede kartofler har afsløret et sæt almindelige gener og vigtige genetiske veje, der har hjulpet spuds med at tilpasse sig gennem tusinder af å -
 Kan sove med en hjernerystelse dræbe dig?En hjernerystelse er en alvorlig hjerneskade, men søvn ser ikke ud til at udgøre en ekstra risiko. Hero Images/Getty Images Bethany spiller første base på virksomhedens softballhold og har ry for at
Kan sove med en hjernerystelse dræbe dig?En hjernerystelse er en alvorlig hjerneskade, men søvn ser ikke ud til at udgøre en ekstra risiko. Hero Images/Getty Images Bethany spiller første base på virksomhedens softballhold og har ry for at -
 Den ulige empati for stedfortræder forlegenhedKan du genkende nogle af disse øjeblikke? Der er Ted Cruz og hans datter; Obama og hans døtre (da han benådede en Thanksgiving -kalkun i 2014); Jennifer Lawrence udslettede ved Oscars 2013; og, selvfø
Den ulige empati for stedfortræder forlegenhedKan du genkende nogle af disse øjeblikke? Der er Ted Cruz og hans datter; Obama og hans døtre (da han benådede en Thanksgiving -kalkun i 2014); Jennifer Lawrence udslettede ved Oscars 2013; og, selvfø -
 Rede af truet kæmpe softshell skildpadde fundet i CambodjaPå dette udaterede billede udgivet af Wildlife Conservation Society (WCS), en truet asiatisk kæmpe softshell skildpadde ses nær en rede af æg på en sandbanke i Mekong-floden mellem Kratie og Stung Tre
Rede af truet kæmpe softshell skildpadde fundet i CambodjaPå dette udaterede billede udgivet af Wildlife Conservation Society (WCS), en truet asiatisk kæmpe softshell skildpadde ses nær en rede af æg på en sandbanke i Mekong-floden mellem Kratie og Stung Tre
- Selvkørende biler og mennesker står over for uundgåelige kollisioner
- Top 10 hoteller, der skræmmer dagslysene ud af dig
- Udholdenhed første store succeser på Mars - en opdatering fra missionsforskere
- Biobaseret harpiks:Et gennembrud inden for hurtig prototyping
- Ny forskning i nordlys vil forbedre satellitnavigationsnøjagtigheden
- En række proaktive politimetoder har succes med at reducere kriminalitet


