Knotty problemer i DNA
Når E. coli-bakterier deler sig, de skal adskille to forbundne cirkler af DNA i to separate cirkler. UC Davis-matematiker Mariel Vazquez og kolleger bruger matematikken i former (topologi) og computermodellering til at forstå denne proces. Kredit:Mariel Vazquez/UC Davis
Hvis du nogensinde har prøvet at løse et par øretelefoner, du vil forstå, hvordan løkker og snore kan blive snoet op. DNA kan blive viklet sammen på samme måde, og i nogle tilfælde, skal skæres og tilsluttes igen for at løse knuderne. Nu er et team af matematikere, biologer og dataloger har afsløret, hvordan E. coli-bakterier kan afkoble sammenfiltret DNA ved en lokal genforbindelsesproces. Matematikken bag forskningen, for nylig udgivet i Videnskabelige rapporter , kan have konsekvenser langt ud over biologi.
E. coli bakterier kan forårsage tarmsygdom, men de er også laboratoriearbejdsheste. E. coli's genom er en enkelt cirkel af dobbeltstrenget DNA. Før en E. coli-celle deler sig, den cirkel er kopieret. Åbning af den dobbelte helix for at kopiere den kaster vridningsspændinger andre steder ned i molekylet - ligesom udrulning af en ledning ét sted vil få den til at spole sig et andet sted. Processen resulterer i to snoede løkker af DNA, der passerer gennem hinanden som et "magiske ringe"-trick.
For at adskille ringene, E. coli bruger et enzym kaldet topoisomerase IV, som præcist skærer et DNA-segment, tillader løkkerne at passere gennem bruddet og forsegler derefter bruddet igen. Fordi topoisomerase IV er så vigtig for bakterier, det er et fristende mål for antibiotika såsom ciprofloxacin. Men når topoisomerase IV er fraværende, et andet enzymkompleks kan træde til for at udføre denne afkobling, selvom det er mindre effektivt. Dette kompleks introducerer to brud og afkoblinger ved at forbinde de fire løse ender igen.
"Der er andre måder at fjerne ringene på, men hvordan gør de det?" sagde Mariel Vazquez, professor i matematik og mikrobiologi og molekylær genetik ved University of California, Davis.
Én vej, Vazquez sagde, er, at genkoblingsenzymerne fjerner et led ad gangen, indtil de når nul. Den løsning var foretrukket af biologerne.
Men matematikere ser lidt anderledes på problemet. De forstår DNA'et som en fleksibel kurve i tredimensionelt rum. Visse punkter på kurven kan brydes og forbindes igen. Til en matematiker, der er mange potentielle ruter for genforbindelsesprocesser til at fungere - inklusive nogle, hvor antallet af links faktisk går op, før det går ned igen.
"Dette er det samme for en matematiker, men ikke til en biolog, " sagde Vazquez. For at bestemme den mest sandsynlige rute og løse problemet, de vendte sig til beregningsmodellering.
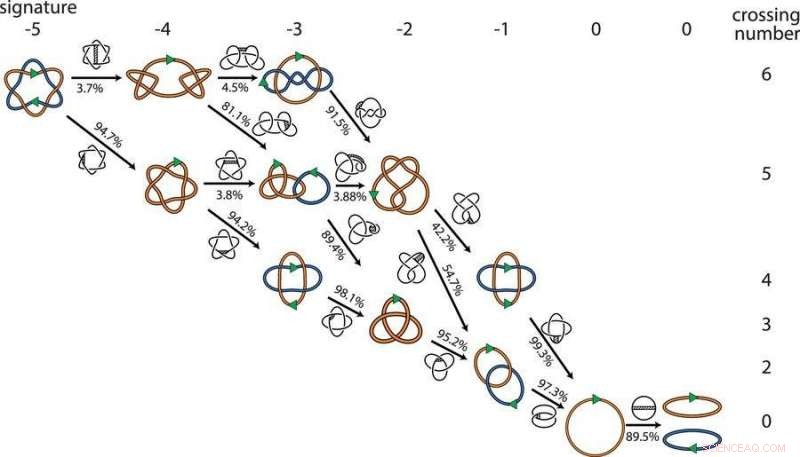
Vazquez og kolleger udviklede computersoftware med DNA repræsenteret som fleksible kæder til at modellere de mulige steder, hvor genforbindelsesenzymer kunne skære og genforbinde kæderne. Samlet set, de modellerede millioner af konfigurationer, der repræsenterede 881 forskellige topologier, eller matematiske former, og identificerede hundredvis af minimale veje for at få to DNA-cirkler forbundet på op til ni steder ned til to separate cirkler.
Computermodellen bekræftede biologernes anelse:At fortryde ét led ad gangen er den foretrukne vej til at adskille DNA-cirklerne.
Resultaterne kan have implikationer langt ud over DNA-biologi, sagde Vazquez. Der er andre eksempler i naturen på objekter, der kolliderer, bryde og genoprette forbindelsen – ligesom dynamikken i forbundne væskehvirvler, eller mønstrene dannet af røgringe, for eksempel. Når soludbrud skydes ud fra solen, kraftfulde magnetfeltlinjer krydser og forbinder igen.
"Matematikken er ikke DNA-specifik, og beregningen kan tilpasses, " sagde Vazquez.
 Varme artikler
Varme artikler
-
 RNA-opdagelse kan hjælpe med at booste plantevarme, tørketoleranceEn side-by-side sammenligning viser bedre varme- og tørketolerance i thale karseplanter, hvis niveau af et specifikt RNA blev øget. Kredit:Texas A&M AgriLife Research Texas A&M AgriLife Research-f
RNA-opdagelse kan hjælpe med at booste plantevarme, tørketoleranceEn side-by-side sammenligning viser bedre varme- og tørketolerance i thale karseplanter, hvis niveau af et specifikt RNA blev øget. Kredit:Texas A&M AgriLife Research Texas A&M AgriLife Research-f -
 Geovidenskabsmænd sammenligner mikroorganismer i polarområderneTübingen geoforsker Julia Kleinteich tager ferskvandsprøver nær forskningsstationen Rothera i Antarktis. Kredit:Daniel Farinotti Selvom de arktiske og antarktiske områder er i hver sin ende af jor
Geovidenskabsmænd sammenligner mikroorganismer i polarområderneTübingen geoforsker Julia Kleinteich tager ferskvandsprøver nær forskningsstationen Rothera i Antarktis. Kredit:Daniel Farinotti Selvom de arktiske og antarktiske områder er i hver sin ende af jor -
 Forskere foreslår universelle love om størrelsen og biologien af plantefrøForskere ved Universitetet i Granada (UGR) har opdaget, at forskellige typer frø tillader planter at tilpasse sig miljøets cyklusser, efterhånden som de udvikler sig. Kredit:University of Granada
Forskere foreslår universelle love om størrelsen og biologien af plantefrøForskere ved Universitetet i Granada (UGR) har opdaget, at forskellige typer frø tillader planter at tilpasse sig miljøets cyklusser, efterhånden som de udvikler sig. Kredit:University of Granada -
 Ny analyse placerer Hobbit på uventet lem på det menneskelige stamtræEn buste i David H. Koch Hall of Human Origins på Smithsonian Natural History Museum genskaber, hvordan en levende Homo floresiensis kan have set ud. Ryan Somma/Flickr Der var engang, der var ikke ba
Ny analyse placerer Hobbit på uventet lem på det menneskelige stamtræEn buste i David H. Koch Hall of Human Origins på Smithsonian Natural History Museum genskaber, hvordan en levende Homo floresiensis kan have set ud. Ryan Somma/Flickr Der var engang, der var ikke ba
- Super blodmåne:Dine spørgsmål besvaret
- Ind i submillimeteren - de tidlige universernes dannelse
- Forskere laver levende kort over ideer til vandfinansiering, der er klar
- Folketællingen bliver digital:3 ting at vide
- Videregående uddannelse var allerede moden til at blive forstyrret - dengang, COVID-19 skete
- Til e-, eller ej til e-, spørgsmålet til den eksotiske Si-III fase af silicium


