Ny teknologi belyser mikrober, der ikke kan dyrkes i et laboratorium

Forskere brugte dette integrerede mikrofluidiske kredsløb til at udføre mini-metagenomisk mikrobiel celleopdeling og genomisk DNA-amplifikation. Kredit:Brian Yu
På Stanford University, forskere har brugt et nyt mikrofluidisk analysesystem til at udtrække 29 nye mikrobielle genomer (det komplette sæt af genetisk materiale) fra prøver fra to varme kilder i Yellowstone National Park. De udtog genomerne, mens de stadig bevarede enkeltcelleopløsning, hvilket betyder, at de vidste, hvilke celler det genetiske materiale kom fra. Dette arbejde blev muliggjort af en ny teknologi, der deler prøven for at muliggøre nøjagtig analyse af en mikrobes genetiske materiale. Specifikt, det giver detaljer om genomets funktion og overflod. Arbejdet blev muliggjort af Emerging Technology Opportunity Program, en del af U.S. Department of Energy Joint Genome Institute (DOE JGI), en DOE Office of Science brugerfacilitet.
Denne nye teknologi belyser mikrobielt "mørkt stof, " genetisk information fra størstedelen af planetens mikrobielle mangfoldighed, som ikke er blevet dyrket i et laboratorium. Disse mikrober lever på steder så forskellige som varme kilder og ørkener, under antarktisk is og i sur minedræning fra Superfund-steder. Værktøjer, der kan bestemme mikrobers genetik og stofskifte, vil have anvendelser inden for områder lige fra bioenergi til bioteknologi til miljøforskning.
Der er mere end 50, 000 mikrobielle genomsekvenser i DOE JGI's Integrated Microbial Genomes offentligt tilgængelige database, og mange af dem er blevet afsløret ved hjælp af metagenomisk sekventering og enkeltcellet genomik. På trods af deres anvendelighed, disse sekventerings- og genomiske teknikker har begrænsninger:enkeltcellede genom-amplifikationer er tidskrævende, ofte ufuldstændige, og metagenomisk sekventering fungerer generelt bedst, hvis miljøprøven ikke er for kompleks. I eLife, et team af forskere fra Stanford University rapporterer udviklingen af en mikrofluidik-baseret, mini-metagenomics tilgang til at afbøde disse udfordringer. Teknikken starter med at reducere miljøprøvens kompleksitet ved at adskille den, ved hjælp af mikrofluidik, i 96 delprøver hver med 5 til 10 celler. Derefter, genomerne i cellerne i hver delprøve amplificeres, og der skabes biblioteker til sekventering af disse mini-metagenomer. De mindre delprøver kan holdes til enkeltcelleopløsning til statistiske analyser. Co-forekomst mønstre fra mange delprøver kan også bruges til at udføre sekvens-uafhængig genom binning. Teknologien blev udviklet gennem ressourcer leveret af DOE JGI's Emerging Technologies Opportunity Program, som blev lanceret i 2013.
Målet med JGI's Emerging Technologies Opportunity Program er at bruge disse nye teknologier til at tackle energi- og miljøapplikationer, tilføjer værdi til high-throughput sekventering og analyse, der udføres for DOE JGI-brugere. Holdet validerede teknikken ved hjælp af et syntetisk mikrobielt fællesskab, og derefter anvendt det på prøver fra Bijah og Mound varme kilder i Yellowstone National Park. Blandt deres resultater var, at mikroberne ved Mound Spring havde større potentiale til at producere metan end mikroberne fra Bijah Spring. De identificerede også et mikrobielt genom fra Bijah Spring, der kunne reducere nitrit til nitrogen. Anvendelse af denne nye teknologi på yderligere prøvesteder vil føje til rækken af hidtil ukarakteriserede mikrobielle egenskaber med potentiel DOE-missionsanvendelse.
 Varme artikler
Varme artikler
-
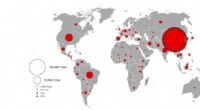 Ny undersøgelse fremhæver virkningerne af en global strategi til at reducere antibiotikaforbruget …Antimikrobielt forbrug til animalsk fødevareproduktion efter land i 2013 (lyserød) og fremskrevet for 2030 (mørkerød). Kredit:Center for Disease Dynamics, Økonomi og politik En ny undersøgelse i
Ny undersøgelse fremhæver virkningerne af en global strategi til at reducere antibiotikaforbruget …Antimikrobielt forbrug til animalsk fødevareproduktion efter land i 2013 (lyserød) og fremskrevet for 2030 (mørkerød). Kredit:Center for Disease Dynamics, Økonomi og politik En ny undersøgelse i -
 To nye krebsdyrarter opdaget på galicisk havbundPhotis guerrai . Kredit:José Antonio Peñas (SINC) Havbunden på kontinentalsoklen og skråningen er hjemsted for rige dyrelivssamfund. Men faunaen på dybe havbund har en tendens til at være relativ
To nye krebsdyrarter opdaget på galicisk havbundPhotis guerrai . Kredit:José Antonio Peñas (SINC) Havbunden på kontinentalsoklen og skråningen er hjemsted for rige dyrelivssamfund. Men faunaen på dybe havbund har en tendens til at være relativ -
 Hvornår er latter et medicinsk symptom?Det kan se ud som sjovt, men ukontrollabel latter kan være et symptom på noget langt mere alvorligt. Tom LeGoff/Digital Vision/Getty Images Normalt, vi griner, fordi noget er sjovt, men nogle gange k
Hvornår er latter et medicinsk symptom?Det kan se ud som sjovt, men ukontrollabel latter kan være et symptom på noget langt mere alvorligt. Tom LeGoff/Digital Vision/Getty Images Normalt, vi griner, fordi noget er sjovt, men nogle gange k -
 Sådan behandles Poison SumacGift sumac ( Toxicodendron vernix ) har typisk mellem syv og 13 blade, alle arrangeret i par, og sport små cremefarvede eller gule bær. Joshua Mayer/Flicker (CC By-SA 2.0) Poison ivy ser ud til at
Sådan behandles Poison SumacGift sumac ( Toxicodendron vernix ) har typisk mellem syv og 13 blade, alle arrangeret i par, og sport små cremefarvede eller gule bær. Joshua Mayer/Flicker (CC By-SA 2.0) Poison ivy ser ud til at
- Uber søger U-turn på regerende britiske chauffører er ansatte
- NASA sporer det østlige Atlanterhav i sensæsonen Tropical Storm Pablo
- PlayStation VR roundup:Stardust Odyssey, Audica fremhæver ferietilbud
- Hvad sker der med en dyrecelle i en hypotonisk løsning?
- Siliciumbaserede nanopartikler kunne gøre LED'er billigere, grønnere at producere
- En ny virtuel tilgang til videnskab i rummet


