Udvikling af en meget nøjagtig beregningsmodel for menneskelig metabolisme
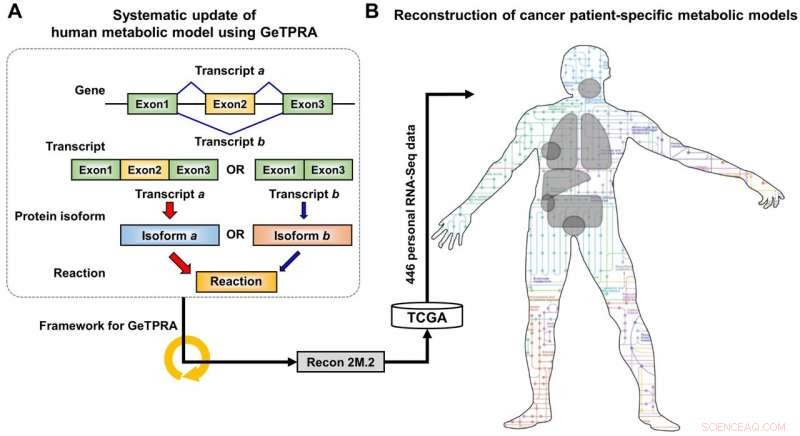
Skema for Recon 2M.2-udvikling og dets anvendelse til rekonstruktion af personlige genom-skala metaboliske modeller (GEM'er). (A) Ved udviklingen af Recon 2M.2, et koncept med alternativ splejsning af menneskelige gener blev overvejet, og inkorporeret i modellering gennem beregningsrammerne for Gene-Transcript-Protein-Reaction Associations (GeTPRA). (B) Ved udvikling af Recon 2M.2, cancerpatientspecifikke GEM'er kunne rekonstrueres ved hjælp af personlige biologiske data (RNA-Seq-data). I dette studie, patientspecifikke RNA-Seq-data blev opnået fra The Cancer Genome Atlas (TCGA; https://cancergenome.nih.gov/). Kredit:KAIST
Et koreansk forskerhold fra KAIST udviklede en beregningsramme, der muliggør rekonstruktionen af en omfattende beregningsmodel for menneskelig metabolisme, som giver mulighed for en nøjagtig forudsigelse af personlige metaboliske træk (eller fænotyper).
Forståelse af personlige metaboliske fænotyper giver os mulighed for at designe effektive terapeutiske strategier for forskellige kroniske og infektionssygdomme. En menneskelig beregningsmodel kaldet den metaboliske model i genomskala (GEM) indeholder information om tusindvis af metaboliske gener og deres tilsvarende reaktioner og metabolitter, og har spillet en vigtig rolle i at forudsige metaboliske fænotyper. Selvom flere versioner af menneskelige GEM'er er blevet frigivet, de havde plads til yderligere udvikling, især med hensyn til at inkorporere biologisk information, der kommer fra en menneskelig genetikmekanisme kaldet "alternativ splejsning". Alternativ splejsning er en genetisk mekanisme, der tillader et gen at give anledning til flere reaktioner, og er stærkt forbundet med patologi.
For at løse dette problem, Jae Yong Ryu (ph.d.-studerende), Dr. Hyun Uk Kim (forskerstipendiat), og den fremtrædende professor Sang Yup Lee, alle fra Institut for Kemi og Biomolekylær Teknik ved KAIST, udviklet en beregningsramme, der systematisk genererer metaboliske reaktioner, og føjer dem til den menneskelige GEM. Den resulterende humane GEM blev påvist at præcist forudsige metaboliske fænotyper under forskellige miljøforhold. Forskningsresultaterne blev offentliggjort online i Proceedings of the National Academy of Sciences ( PNAS ) den 24. oktober 2017, under titlen "Ramme og ressource for mere end 11, 000 gen-transkript-protein-reaktion associationer i menneskelig metabolisme."
Forskerholdet opdaterede først det biologiske indhold af en tidligere version af den menneskelige GEM. Det opdaterede biologiske indhold omfatter metaboliske gener og deres tilsvarende metabolitter og reaktioner. I særdeleshed, metaboliske reaktioner katalyseret af allerede kendte proteinisoformer blev yderligere inkorporeret i den humane GEM; proteinisoformer er flere varianter af proteiner genereret fra individuelle gener gennem den alternative splejsningsproces. Hver proteinisoform er ofte ansvarlig for driften af en metabolisk reaktion. Selvom flere proteinisoformer genereret fra ét gen kan spille forskellige funktioner ved at have forskellige sæt af proteindomæner og/eller subcellulære lokaliseringer, sådanne oplysninger blev ikke taget ordentligt i betragtning i tidligere versioner af menneskelige GEM'er.
Efter den første opdatering af den menneskelige GEM, navngivet Recon 2M.1, forskerholdet implementerede efterfølgende en beregningsramme, der systematisk genererer information om Gene-Transcript-Protein-Reaction Associations (GeTPRA) for at identificere proteinisoformer, der ikke tidligere var identificeret. Denne ramme blev udviklet i denne undersøgelse. Som et resultat af implementeringen af rammen for GeTPRA, mere end 11, 000 GeTPRA blev automatisk forudsagt, og grundigt valideret. Yderligere metaboliske reaktioner blev derefter tilføjet til Recon 2M.1 baseret på den forudsagte GeTPRA for de tidligere ukarakteriserede proteinisoformer; Recon 2M.1 blev omdøbt til Recon 2M.2 fra denne opgradering. Endelig, Recon 2M.2 blev integreret med 446 sæt personlige biologiske data (RNA-Seq-data) for at bygge patientspecifikke cancermodeller. Disse patientspecifikke cancermodeller blev brugt til at forudsige cancermetabolismeaktiviteter og anticancermål.
Udviklingen af en ny version af menneskelige GEM'er sammen med beregningsrammen for GeTPRA forventes at sætte skub i studier i fundamental human genetik og medicin. Modelfiler af de menneskelige GEM'er Recon 2M.1 og 2M.2, en komplet liste over GeTPRA'en og kildekoden til beregningsrammerne til at forudsige GeTPRA'en er alle tilgængelige som en del af udgivelsen af denne undersøgelse.
Den ærede professor Lee sagde, "Den forudsagte GeTPRA fra beregningsrammen forventes at tjene som en rettesnor for fremtidige eksperimenter med human genetik og biokemi, hvorimod den resulterende Recon 2M.2 kan bruges til at forudsige lægemiddelmål for forskellige menneskelige sygdomme."
Sidste artikelKendskab til larvefisk bare en dråbe i havet
Næste artikelHvor meget vejer livet?
 Varme artikler
Varme artikler
-
 Kroppssystemer og deres funktionerMenneskekroppen består af organiserede systemer, som virker ved at opretholde livet. Hvert system er en indeholdt del af kroppen, men disse systemer er afhængige af hinanden for at sikre, at kroppen e
Kroppssystemer og deres funktionerMenneskekroppen består af organiserede systemer, som virker ved at opretholde livet. Hvert system er en indeholdt del af kroppen, men disse systemer er afhængige af hinanden for at sikre, at kroppen e -
 Forskere opdager en evolutionær springbræt til roe-røde roerSkiver af forskellige farvede roer viser tilstedeværelsen af betalainer, en klasse af gule og røde pigmenter, der er unikke for den bredere roefamilie, Caryophyllales. Kredit:Sarah Friedrich Den
Forskere opdager en evolutionær springbræt til roe-røde roerSkiver af forskellige farvede roer viser tilstedeværelsen af betalainer, en klasse af gule og røde pigmenter, der er unikke for den bredere roefamilie, Caryophyllales. Kredit:Sarah Friedrich Den -
 Hvor præcist er vores mentale billede af os selv?Det, vi ser i spejlet, kan være anderledes end det, andre opfatter, når de ser på os. Se flere billeder af følelser. West Rock/Getty Images Når du ser dig selv i et spejl, hvad du ser, afhænger af kv
Hvor præcist er vores mentale billede af os selv?Det, vi ser i spejlet, kan være anderledes end det, andre opfatter, når de ser på os. Se flere billeder af følelser. West Rock/Getty Images Når du ser dig selv i et spejl, hvad du ser, afhænger af kv -
 Egenskaber ved en bakteriecelleCeller er de grundlæggende enheder i livet, og som sådan er de mindste distinkte elementer i levende ting, der bevarer alle de nøgleegenskaber, der er forbundet med levende ting, herunder metabolis
Egenskaber ved en bakteriecelleCeller er de grundlæggende enheder i livet, og som sådan er de mindste distinkte elementer i levende ting, der bevarer alle de nøgleegenskaber, der er forbundet med levende ting, herunder metabolis
- Kakerlakegårde gør madaffald til skat
- Sydafrika har stadig lang vej at gå med hensyn til retten til mad
- Store molekyler fra kostfibre kan ændre tarmmiljøet gennem fysiske kræfter
- G20 accepterer en aftale om havplastikforurening (opdatering)
- Undersøg sonder, hvordan nanopartikler binder sig til blodproteiner ved grænseflader
- Tekstilfiberindlejret multiluminescerende enhed til fremtidige bærbare enheder


