RNA-redigeringsværktøj en hurtig, følsom test for COVID-19
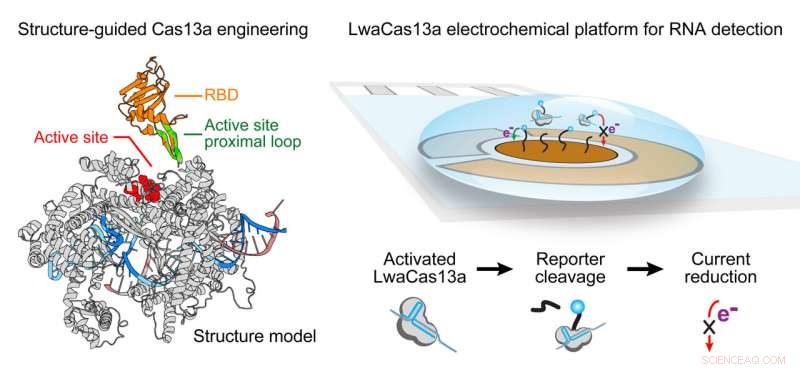
Ved hjælp af strukturstyret Cas13 modificerede forskere ved Rice University og University of Connecticut et genredigeringsværktøj til at fungere som en meget følsom diagnostisk test for tilstedeværelsen af SARS-CoV-2-virus. De brugte en elektrokemisk sensor til at levere resultater. Kredit:Jie Yang/Rice University
En konstrueret CRISPR-baseret metode, der finder RNA fra SARS-CoV-2, den virus, der forårsager COVID-19, lover at gøre testning for den og andre sygdomme hurtig og nem.
Samarbejdspartnere ved Rice University og University of Connecticut udviklede yderligere det RNA-redigerende CRISPR-Cas13-system for at øge deres kraft til at detektere små mængder af SARS-CoV-2-virus i biologiske prøver uden det tidskrævende RNA-ekstraktions- og amplifikationstrin, der er nødvendigt i guld-standard PCR-test.
Den nye platform var meget vellykket sammenlignet med PCR, idet den fandt 10 ud af 11 positive og ingen falsk positive for virussen i test på kliniske prøver direkte fra næsepodninger. Forskerne viste, at deres teknik finder tegn på SARS-CoV-2 i attomolar (10 -18 ) koncentrationer.
Undersøgelsen ledet af den kemiske og biomolekylære ingeniør Xue Sherry Gao ved Rices George R. Brown School of Engineering og postdoktorale forskere Jie Yang fra Rice og Yang Song fra Connecticut vises i Nature Chemical Biology .
Cas13 er ligesom sin bedre kendte fætter Cas9 en del af det system, hvorved bakterier naturligt forsvarer sig mod invaderende fager. Siden sin opdagelse er CRISPR-Cas9 blevet tilpasset af forskere til at redigere levende DNA-genomer og viser et stort løfte om at behandle og endda helbrede sygdomme.

Rice University postdoc-forsker Jie Yang førte et forsøg på at tilpasse Cas13-genomredigeringsværktøjer til at fungere som en meget følsom detektor for tilstedeværelsen af SARS-CoV-2-virussen, som forårsager COVID-19. Kredit:Jeff Fitlow/Rice University
Og det kan bruges på andre måder. Cas13 i sig selv kan forbedres med guide-RNA til at finde og klippe mål-RNA-sekvenser, men også for at finde "collateral", i dette tilfælde tilstedeværelsen af vira som SARS-CoV-2.
"Det konstruerede Cas13-protein i dette arbejde kan let tilpasses til andre tidligere etablerede platforme," sagde Gao. "Stabiliteten og robustheden af konstruerede Cas13-varianter gør dem mere velegnede til point-of-care diagnostik i områder med lav ressource, når dyre PCR-maskiner ikke er tilgængelige."
Yang sagde, at vildtype-Cas13, hentet fra en bakterie, Leptotrichia wadei, ikke kan detektere attomolært niveau af viralt RNA inden for en tidsramme på 30 til 60 minutter, men den forbedrede version, der blev oprettet hos Rice, klarer jobbet på omkring en halv time og detekterer SARS -CoV-2 i meget lavere koncentrationer end de tidligere tests.
Hun sagde, at nøglen er en godt skjult, fleksibel hårnålesløjfe nær Cas13s aktive side. "Det er i midten af proteinet nær det katalytiske sted, der bestemmer Cas13s aktivitet," sagde Yang. "Da Cas13 er stor og dynamisk, var det udfordrende at finde et websted til at indsætte et andet funktionelt domæne."
Forskerne fusionerede syv forskellige RNA-bindingsdomæner til løkken, og to af komplekserne var klart overlegne. Når de fandt deres mål, ville proteinerne fluorescere og afsløre tilstedeværelsen af virussen.
"Vi kunne se, at den øgede aktivitet var fem eller seks gange i forhold til vildtype Cas13," sagde Yang. "Dette tal virker lille, men det er ret forbløffende med et enkelt trin i proteinudvikling.
"But that was still not enough for detection, so we moved the whole assay from a fluorescence plate reader, which is quite large and not available in low-resource settings, to an electrochemical sensor, which has higher sensitivity and can be used for point-of-care diagnostics," she said.
With the off-the-shelf sensor, Yang said the engineered protein was five orders of magnitude more sensitive in detecting the virus compared to the wild-type protein.
The lab wants to adapt its technology to paper strips like those in home COVID-19 antibody tests, but with much higher sensitivity and accuracy. "We hope that will make testing more convenient and with lower cost for many targets," Gao said.
The researchers are also investigating improved detection of the Zika, dengue and Ebola viruses and predictive biomarkers for cardiovascular disease. Their work could lead to rapid diagnosis of the severity of COVID-19.
"Different viruses have different sequences," Yang said. "We can design guide RNA to target a specific sequence that we can then detect, which is the power of the CRISPR-Cas13 system."
But because the project began just as the pandemic took hold, SARS-CoV-2 was a natural focus. "The technology is quite amenable to all the targets," she said. "This makes it a very good option to detect all kinds of mutations or different coronaviruses."
"We are very excited about this work as a combinational effort of structure biology, protein engineering and biomedical device development," Gao added. "I greatly appreciate all the efforts from my lab members and collaborators."
Co-authors of the paper are Rice postdoctoral researcher Xiangyu Deng, undergraduate Jeffrey Vanegas and graduate student Zheng You; graduate students Yuxuan Zhang and Zhengyan Weng of the University of Connecticut; microbiology supervisor Lori Avery and Kevin Dieckhaus, a professor of medicine, of UConn Health; Yi Zhang, an assistant professor of biomedical engineering at the University of Connecticut; and Yang Gao, an assistant professor of biosciences at Rice. + Udforsk yderligere
Compact CRISPR system enables portable COVID-19 testing
 Varme artikler
Varme artikler
-
 Forskerteam introducerer fremskridt inden for automatisk skovkortlægningsteknologiDette billede viser input- og outputdataene for træsegmenteringsalgoritmen. Indtastningsdataene (til venstre) er farvet af højde. Resultaterne af algoritmen (højre) bruger farve til at segmentere hver
Forskerteam introducerer fremskridt inden for automatisk skovkortlægningsteknologiDette billede viser input- og outputdataene for træsegmenteringsalgoritmen. Indtastningsdataene (til venstre) er farvet af højde. Resultaterne af algoritmen (højre) bruger farve til at segmentere hver -
 Forsendelsesgigant ændrer kurs for at redde Sri Lanka-hvalerKampagnefolk mener, at mere end et dusin blåhvaler er blevet dræbt i kollisioner med kommercielle skibe i det sidste årti. Dyrerettighedsaktivister jublede fredag et skridt fra en skibsgigant om
Forsendelsesgigant ændrer kurs for at redde Sri Lanka-hvalerKampagnefolk mener, at mere end et dusin blåhvaler er blevet dræbt i kollisioner med kommercielle skibe i det sidste årti. Dyrerettighedsaktivister jublede fredag et skridt fra en skibsgigant om -
 Hvad er funktionerne ved mikrofilamenter og mikrotubuli?Mikrofilamenter og mikrotubuli er de dele af enhver organisms celler, der giver styrke og strukturel støtte. De er de vigtigste komponenter i cytoskelettet, en ramme af proteiner, der giver cellen sin
Hvad er funktionerne ved mikrofilamenter og mikrotubuli?Mikrofilamenter og mikrotubuli er de dele af enhver organisms celler, der giver styrke og strukturel støtte. De er de vigtigste komponenter i cytoskelettet, en ramme af proteiner, der giver cellen sin -
 Sammenligning af mængden af energi, som to arter af Galapagos-skildpadder skal bruge for at vende…Kredit:CC0 Public Domain (Phys.org) – Et team af forskere med medlemmer fra USA, Portugal og Frankrig har gennemført en undersøgelse af mængden af energi, der kræves af de to hovedtyper af skild
Sammenligning af mængden af energi, som to arter af Galapagos-skildpadder skal bruge for at vende…Kredit:CC0 Public Domain (Phys.org) – Et team af forskere med medlemmer fra USA, Portugal og Frankrig har gennemført en undersøgelse af mængden af energi, der kræves af de to hovedtyper af skild
- Kinas bildilemma:Beijing vil have elektrisk, købere vil have SUV'er
- Syntese af en nær-infrarød lysabsorberende makrocyklisk aromatisk forbindelse
- Forskere vurderer de accelererede ændringer af gletsjere i Yulong Snow Mountain
- Forskere designer nyt responsivt porøst materiale inspireret af proteiner
- Forskerteam omvendt måde kaliumkanaler fungerer fra bakterier til mennesker
- Sådan beregner du graden og vinklerne til pyramidehustakbyggeri


