Forskere konstruerer det mest komplekse, komplette syntetiske mikrobiom
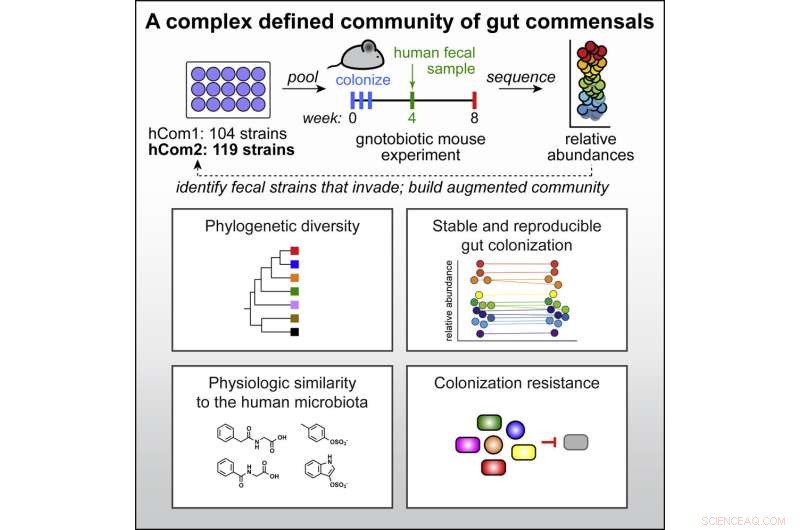
Grafisk abstrakt. Kredit:Celle (2022). DOI:10.1016/j.cell.2022.08.003
Nøgleundersøgelser i det sidste årti har vist, at tarmmikrobiomet, samlingen af hundredvis af bakteriearter, der lever i det menneskelige fordøjelsessystem, påvirker neural udvikling, respons på cancerimmunterapier og andre aspekter af sundhed. Men disse samfund er komplekse og uden systematiske måder at studere bestanddelene på, forbliver de nøjagtige celler og molekyler forbundet med visse sygdomme et mysterium.
Forskere fra Stanford University har bygget det mest komplekse og veldefinerede syntetiske mikrobiom og skabt et samfund af over 100 bakteriearter, der med succes blev transplanteret til mus. Muligheden for at tilføje, fjerne og redigere individuelle arter vil give videnskabsfolk mulighed for bedre at forstå sammenhængen mellem mikrobiomet og sundhed og til sidst udvikle førsteklasses mikrobiomterapier.
Mange vigtige mikrobiomundersøgelser er blevet udført ved hjælp af fækale transplantationer, som introducerer hele det naturlige mikrobiom fra en organisme til en anden. Mens videnskabsmænd rutinemæssigt slår et gen til tavshed eller fjerner et protein fra en specifik celle eller endda en hel mus, findes der ikke et sådant sæt værktøjer til at fjerne eller modificere én art blandt de hundredvis i en given fæcesprøve.
"Så meget af det, vi ved om biologi, ville vi ikke vide, hvis det ikke var for evnen til at manipulere komplekse biologiske systemer stykkevis," sagde Michael Fischbach, institutforsker ved Sarafan ChEM-H og tilsvarende forfatter til undersøgelsen, offentliggjort. i Celle den 6. september.
Fischbach, der er lektor i bioteknik og i mikrobiologi og immunologi, og andre så én løsning:Byg et mikrobiom fra bunden ved at vokse individuelt og derefter blande dets bestanddele.
Bygning af arken
Hver celle i mikrobiomet indtager en specifik funktionel niche, der udfører reaktioner, der nedbryder og opbygger molekyler. For at bygge et mikrobiom skulle holdet sikre, at den endelige blanding ikke kun var stabil, opretholde en balance, uden at nogen enkelt art overmandede resten, men også funktionel, og udføre alle handlingerne af et komplet, naturligt mikrobiom. At udvælge arter til at inkludere i deres syntetiske samfund var også vanskeligt i betragtning af den naturlige variation på tværs af individer; to personer, der er udvalgt tilfældigt, deler mindre end halvdelen af deres mikrobielle gener.
Forskerne besluttede at bygge deres koloni fra de mest udbredte bakterier og henvendte sig til Human Microbiome Project (HMP), et National Institutes of Health-initiativ for at sekventere de fulde mikrobielle genomer fra over 300 voksne.
"Vi ledte efter Noahs Ark af bakteriearter i menneskets tarm, og forsøgte at finde dem, der næsten altid var der hos ethvert individ," sagde Fischbach.
De udvalgte over 100 bakteriestammer, der var til stede i mindst 20 % af HMP-individerne. Tilføjelse af et par arter, der var nødvendige for nogle efterfølgende undersøgelser, bragte dem til 104 arter, som de dyrkede i individuelle bestande og derefter blandet i én kombineret kultur for at gøre det, de kalder menneskeligt samfund til ét, eller hCom1.
Selvom de var tilfredse med, at stammerne kunne eksistere sammen i laboratoriet, var den sande test, om deres nye koloni ville slå rod i tarmen. De introducerede hCom1 til mus, der er omhyggeligt designet til ikke at have nogen bakterier til stede. hCom1 var bemærkelsesværdigt stabilt, hvor 98 % af arterne koloniserede tarmen hos disse bakteriefrie mus, og de relative overflodsniveauer for hver art forblev konstante over to måneder.
Udenlandsk invasion
For at gøre deres koloni mere komplet, ønskede forskerne at sikre, at alle vitale mikrobiomfunktioner ville blive udført af en eller flere arter. They relied on a theory called colonization resistance, which explains that any bacterium, when introduced into an existing colony, will only survive if it can fill a niche not already occupied.
By introducing a complete microbiome, in the form of a human fecal sample, to their colony and tracking any new species that took up residence, they could build a more complete community.
Some were skeptical that this would work. "The bacterial species in hCom1 had lived together for only a few weeks," said Fischbach. "Here we were, introducing a community that had coexisted for a decade. Some people thought they would decimate our colony."
Remarkably, hCom1 held its own, and only about 10% of cells in the final community came from the fecal transplant.
They found over 20 new bacterial species that inserted themselves in at least two of their three fecal transplant studies. Adding those to their initial community and removing those that failed to take root in mouse guts gave them a new community of 119 strains, dubbed hCom2. This second iteration, still made from individually growing and then mixing the constituents, made mice even more resistant to fecal challenges than the first.
Final challenge
To demonstrate the utility of their synthetic microbiome, the team took hCom2-colonized mice and challenged them with a sample of E. coli. These mice, like those that were colonized with a natural microbiome, resisted infection.
Prior studies have shown that a healthy natural microbiome leads to protection, but Fischbach and colleagues could take this a step further by iteratively eliminating or modifying certain strains to determine which ones specifically conferred protection. They found several key bacteria and plan to conduct further studies to narrow down to the most critical species.
Fischbach believes that hCom2, or future versions of it, will enable similar reductionist studies that reveal the bacterial agents involved in other areas, like immunotherapy responses.
"We built this consortium for the broader research community. We want to get this into as many hands as possible to have an impact on the field," said Fischbach.
He also envisions that this method of building a microbiome from the ground up will make engineered microbiome-based therapies possible in the future. As the director of the Stanford Microbiome Therapies Initiative (MITI), an initiative launched in 2019 by Sarafan ChEM-H and the Department of Bioengineering, he aims to construct engineered communities that could one day be transplanted into people to treat or prevent a variety of diseases. + Udforsk yderligere
Ecological principles at play in gut microbiome
Sidste artikelForskere fanger live-optagelser af virusinficerende celle
Næste artikelRedet Irrawaddy delfinkalv dør trods ugers pleje
 Varme artikler
Varme artikler
-
 Team afslører regler for fremstilling af ribbenMuse ribben bur farvet for at vise brusk (blå) og knogle (rød) Kredit:Francesca Mariani Forskere fra USC Stem Cell lab of Francesca Mariani delte for nylig en opskrift på ribben, og det kræver ikk
Team afslører regler for fremstilling af ribbenMuse ribben bur farvet for at vise brusk (blå) og knogle (rød) Kredit:Francesca Mariani Forskere fra USC Stem Cell lab of Francesca Mariani delte for nylig en opskrift på ribben, og det kræver ikk -
 Hvad er de fire nitrogenholdige baser af DNA?Deoxyribonukleinsyre eller DNA er muligvis det mest berømte enkeltmolekyle i hele biologien. Opdagelsen af dens dobbelt-helix-struktur i 1953 katapulterede James Watson og Francis Crick en Nobelpris
Hvad er de fire nitrogenholdige baser af DNA?Deoxyribonukleinsyre eller DNA er muligvis det mest berømte enkeltmolekyle i hele biologien. Opdagelsen af dens dobbelt-helix-struktur i 1953 katapulterede James Watson og Francis Crick en Nobelpris -
 En sangfugl prangende gul hals? Det er der gener forForskning fra University of British Columbia (UBC) kunne have udpeget nogle af de genetiske maskiner, der er ansvarlige for fjerdragtfarven hos Audubons og myrtesanger, beslægtede, men tydeligt fjerbe
En sangfugl prangende gul hals? Det er der gener forForskning fra University of British Columbia (UBC) kunne have udpeget nogle af de genetiske maskiner, der er ansvarlige for fjerdragtfarven hos Audubons og myrtesanger, beslægtede, men tydeligt fjerbe -
 Anti-Aging-behandlinger - Videnskaben om at leve længereManeten Turritopsis dohrnii lever praktisk talt for evigt ved at omprogrammere sine gamle celler for at gøre dem unge igen. Forskere og forskere studerer ikke kun vandmændene, men de studerer en række
Anti-Aging-behandlinger - Videnskaben om at leve længereManeten Turritopsis dohrnii lever praktisk talt for evigt ved at omprogrammere sine gamle celler for at gøre dem unge igen. Forskere og forskere studerer ikke kun vandmændene, men de studerer en række
- Hvordan virker skeletsystemet med åndedrætssystemet?
- Da Kina hurtigt vedtager ren energi, brug af traditionelle ovne vedvarer
- Når de er blevet forringet, Brasiliansk savanne regenereres ikke naturligt
- Sådan beregnes tætheden af en Polymer Blend
- Flere republikanere følger COVID-retningslinjerne, når de får at vide, at det vil beskytte dem se…
- Forskere fremstiller alsidig katalysator til polyestersyntese og nedbrydning


