Læser RNA-modifikationer mere præcist i en enhed i lommestørrelse
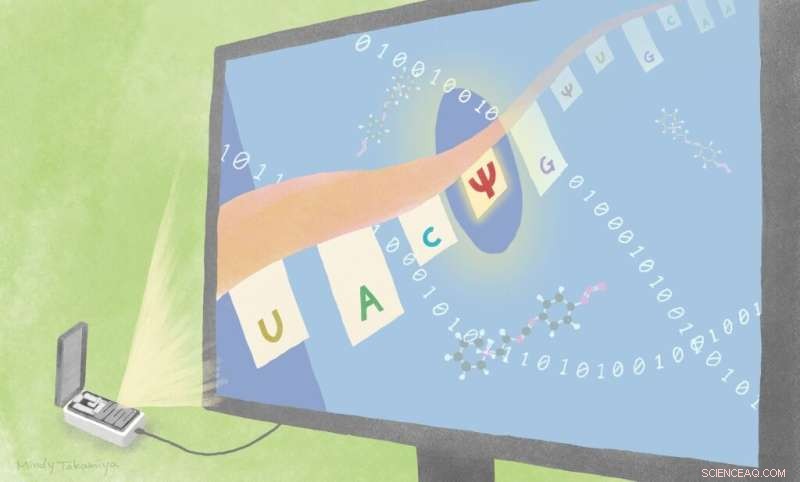
Forskere fandt to nye måder at identificere substitutioner fra uracil til pseudouridin ved at kombinere eksisterende sekventeringsteknologi med forskellige tilgange. En er med algoritme-baseret beregning, og en er med en kemisk sonde CMC. Kredit:Mindy Takamiya/Kyoto University iCeMS
To nye tilgange kunne hjælpe forskere med at bruge eksisterende sekventeringsteknologi til bedre at skelne RNA-ændringer, der påvirker, hvordan genetisk kode læses.
Forskere fra Kyoto University kommer tættere på at finde måder at identificere ændringer i RNA-sekvenser, der påvirker proteindannelsen og kan forårsage sygdomme. Deres tilgang, offentliggjort i tidsskriftet Genomics , bruger sandsynlighedsalgoritmer sammen med en allerede tilgængelig sekventeringsenhed i lommestørrelse.
"Modifikationer, der findes i alle typer af biologisk RNA, påvirker genreguleringen, som i sidste ende bestemmer, hvordan forskellige celler fungerer i vores krop," forklarer Ganesh Pandian Namasivayam fra Kyoto University's Institute for Integrated Cell-Material Science (WPI-iCeMS). "Anormaliteter i disse modifikationer kan føre til alvorlige sygdomme, såsom diabetes, neuroudviklingsforstyrrelser og kræft. At vide, hvordan og hvor disse RNA-modifikationer er, er af største betydning fra et klinisk synspunkt," tilføjer Soundhar Ramaswamy, undersøgelsens første forfatter.
Der er allerede måder at identificere RNA-modifikationer på, men de er utilstrækkelige. Biofysiske tilgange såsom kromatografi og massespektrometri kan kun behandle små mængder RNA ad gangen. High-throughput sekventeringsmetoder, som kan behandle store mængder RNA, involverer besværlig prøveforberedelse, kan ikke samtidig kortlægge flere modifikationer og er fejltilbøjelige.
Namasivayam, Hiroshi Sugiyama og kolleger ved Kyoto University testede og fandt to tilgange, der relativt succesfuldt kan skelne mellem en velkendt og rigelig RNA-modifikation, der involverer udskiftning af nukleotidbasen uracil med en anden kaldet pseudouridin.
I lighed med DNA er RNA dannet af en streng af forskellige kombinationer af fire forskellige nukleotidbaser:uracil, cytosin, adenin og guanin. Hvordan disse baser er arrangeret bestemmer koden, der signalerer, hvilket protein der er beregnet til at blive lavet. Når pseudouridin erstatter uracil i RNA-rygraden, kan det føre til øget proteinproduktion eller til at ændre koden fra en kode, der signalerer afbrydelse af informationsoversættelse til en, der signalerer aminosyredannelse.
Holdets tilgang involverer at bruge en allerede tilgængelig direkte RNA-sekventeringsplatform udviklet af Oxford Nanopore Technologies. I denne platform passerer RNA-strenge gennem bittesmå porer i en membran. Forstyrrelser er forårsaget af strømmen, der bevæger sig gennem membranen afhængigt af rækkefølgen af de forskellige RNA-baser. Dette gør det muligt for videnskabsmænd at "læse" sekvensen. Men forskere, der bruger denne tilgang, har ofte svært ved at skelne forskellige typer modifikationer fra hinanden.
Shubham Mishra, en fælles førsteforfatter af denne undersøgelse, udviklede algoritmer til at identificere en høj sandsynlighed for eksistensen af en pseudouridinsubstitution sammenlignet med muligheden for, at det var en anden slags baseændring.
En af deres strategier sammenligner korte RNA-kørsler af fem nukleotidbaser, hvor uracil, pseudouridin eller cytosin er omgivet på hver side af de samme baser. Aflæsningerne går derefter gennem algoritmer, der beregner sandsynligheden for, at den midterste base er en af de tre. De brugte deres strategi, kaldet Indo-Compare (Indo-C), på konstruerede RNA-sekvenser og derefter på gær og humant RNA og fandt ud af, at den var god til at skelne pseudouridinsubstitutionerne fra de andre.
De var også i stand til at identificere pseudouridinsubstitutioner ved at blande en kemisk probe med RNA-prøver, som derefter selektivt binder til dem. Dette ændrede sekvensaflæsningerne på en måde, der identificerer ændringen.
"Vi tror på, at vores arbejde vil gøre nanopore-sekventeringsbaserede metoder mindre besværlige til at detektere RNA-modifikationer og mere i stand til at karakterisere virkningerne af disse modifikationer på udvikling og sygdom," siger Namasivayam.
Holdet sigter derefter på at optimere brugen af begge tilgange sammen for mere præcist at identificere RNA- og DNA-modifikationer. Dette vil involvere fremstilling af nye kemiske sonder, der svarer til specifikke ændringer. De planlægger også at videreudvikle avancerede maskinlæringsalgoritmer, der komplementerer kemiske probe-baserede direkte RNA-sekventeringstilgange. + Udforsk yderligere
Sekventering af flere RNA-basemodifikationer samtidigt:En ny æra af RNA-biologi
 Varme artikler
Varme artikler
-
 Havskildpadder dør efter at være blevet viklet ind i plastikaffaldLevende læderskildpadde viklet ind i fiskereb, hvilket øger modstanden, Grenada 2014. Kredit:Kate Charles, Ocean Spirits Hundredvis af havskildpadder dør hvert år efter at være blevet viklet ind i
Havskildpadder dør efter at være blevet viklet ind i plastikaffaldLevende læderskildpadde viklet ind i fiskereb, hvilket øger modstanden, Grenada 2014. Kredit:Kate Charles, Ocean Spirits Hundredvis af havskildpadder dør hvert år efter at være blevet viklet ind i -
 Metafase: Hvad sker der i denne fase af mitose og meiose?Metaphase er den tredje af de fem faser af biologisk celledeling, eller mere specifikt, opdelingen af hvad der er inde i den celle kerne. I de fleste tilfælde er denne opdeling mitose, som er det
Metafase: Hvad sker der i denne fase af mitose og meiose?Metaphase er den tredje af de fem faser af biologisk celledeling, eller mere specifikt, opdelingen af hvad der er inde i den celle kerne. I de fleste tilfælde er denne opdeling mitose, som er det -
 Forskere præsenterer ny matematisk model til undersøgelse af nyrecellerKredit:Katherine E Shipman et al., Function (2022). DOI:10.1093/function/zqac046 Forskning offentliggjort forud for tryk i tidsskriftet Function præsenterer en ny matematisk model, der bruger oposs
Forskere præsenterer ny matematisk model til undersøgelse af nyrecellerKredit:Katherine E Shipman et al., Function (2022). DOI:10.1093/function/zqac046 Forskning offentliggjort forud for tryk i tidsskriftet Function præsenterer en ny matematisk model, der bruger oposs -
 Hvornår er latter et medicinsk symptom?Det kan se ud som sjovt, men ukontrollabel latter kan være et symptom på noget langt mere alvorligt. Tom LeGoff/Digital Vision/Getty Images Normalt, vi griner, fordi noget er sjovt, men nogle gange k
Hvornår er latter et medicinsk symptom?Det kan se ud som sjovt, men ukontrollabel latter kan være et symptom på noget langt mere alvorligt. Tom LeGoff/Digital Vision/Getty Images Normalt, vi griner, fordi noget er sjovt, men nogle gange k
- Forberedt til Barry:New Orleans spænder for ekstrem storm
- Elcykler nynner snart langs nationalparkens stier
- New York -staten forbereder sig på at forbyde plastikposer
- Nintendo udgiver remasterede Mario-klassikere til Switch i 2020
- Våd vinter kan hjælpe Colorado-floden med at afhjælpe problemer, men det vil ikke afslutte tørke…
- Grøn nanosyn er nu en køreplan for udvikling


