AI-system kan forudsige strukturerne af livets molekyler med forbløffende nøjagtighed

AlphaFold 3, der blev afsløret for verden den 9. maj, er den seneste version af en algoritme designet til at forudsige strukturerne af proteiner – vitale molekyler, der bruges af alt liv – ud fra "instruktionskoden" i deres byggesten.
At forudsige proteinstrukturer og den måde, de interagerer med andre molekyler på, har været et af de største problemer i biologien. Alligevel er AI-udvikleren Google DeepMind gået et stykke vej for at løse det i de sidste par år. Denne nye version af AI-systemet har forbedret funktion og nøjagtighed i forhold til sine forgængere.
Ligesom den næste udgivelse i en videospilsfranchise, har strukturbiologer – og senest – kemikere ventet med utålmodighed på at se, hvad det kan gøre. DNA er almindeligt forstået som instruktionsbogen for en levende organisme, men inde i vores celler er proteiner de molekyler, der faktisk udfører det meste af arbejdet.
Det er proteiner, der gør vores celler i stand til at fornemme verden udenfor, til at integrere information fra forskellige signaler, til at lave nye molekyler i cellen, til at beslutte at vokse eller stoppe med at vokse.
Det er også proteiner, der sætter kroppen i stand til at skelne mellem fremmede angribere (bakterier, vira) og sig selv. Og det er proteiner, der er målene for de fleste lægemidler, som du eller jeg tager for at behandle sygdom.
Protein Lego
Hvorfor har proteinstrukturen betydning? Proteiner er store molekyler, der består af tusindvis af atomer i meget bestemte rækkefølger. Rækkefølgen af disse atomer, og den måde, de er arrangeret i 3D-rum, er afgørende for, at et protein kan udføre sin biologiske funktion.
Det samme 3D-arrangement bestemmer også den måde, hvorpå et lægemiddelmolekyle binder til sit proteinmål og behandler sygdom.
Forestil dig at have et Lego-sæt, hvor klodserne ikke er baseret på cuboider, men kan have enhver form. For at sætte to klodser sammen i dette sæt, skal hver klods passe tæt mod hinanden uden huller. Men det er ikke nok – de to klodser skal også have den rigtige kombination af bump og huller, for at klodserne kan blive på plads.
At designe et nyt lægemiddelmolekyle er lidt som at lege med dette nye Lego-sæt. Nogen har allerede bygget en enorm model (proteinmålet fundet i vores celler), og lægemiddelopdagelseskemikerens opgave er at bruge deres værktøjssæt til at sætte en håndfuld klodser sammen, der vil binde til en bestemt del af proteinet og – i biologiske termer – stoppe den med at udføre sin normale funktion.
Så hvad gør AlphaFold? Baseret på at vide præcis, hvilke atomer der er i ethvert protein, hvordan disse atomer har udviklet sig forskelligt i forskellige arter, og hvordan andre proteinstrukturer ser ud, er AlphaFold meget god til at forudsige 3D-strukturen af ethvert protein.
AlphaFold 3, den seneste iteration, har udvidet mulighederne for at modellere nukleinsyrer, for eksempel stykker af DNA. Det kan også forudsige formerne af proteiner, der er blevet modificeret med kemiske grupper, der kan tænde eller slukke for proteinet, eller med sukkermolekyler. Dette giver forskerne mere end blot et større, mere farverigt Lego-sæt at lege med. Det betyder, at de kan udvikle mere detaljerede modeller til at læse og korrigere den genetiske kode og af cellulære kontrolmekanismer.
Dette er vigtigt for at forstå sygdomsprocesser på et molekylært niveau og for at udvikle lægemidler, der er målrettet mod proteiner, hvis biologiske rolle er at regulere, hvilke gener der tændes eller slukkes. Den nye version af AlphaFold forudsiger også antistoffer med større nøjagtighed end tidligere versioner.
Antistoffer er vigtige proteiner i biologien i sig selv og udgør en vital del af immunsystemet. De bruges også som biologiske lægemidler såsom trastuzumab, mod brystkræft og infliximab, mod sygdomme som inflammatorisk tarmsygdom og leddegigt.
Den seneste version af AlphaFold kan forudsige strukturen af proteiner bundet til lægemiddellignende små molekyler. Lægemiddelopdagelseskemikere kan allerede forudsige den måde, hvorpå et potentielt lægemiddel binder til sit proteinmål, hvis målets 3D-struktur er blevet identificeret gennem eksperimenter. Ulempen er, at denne proces kan tage måneder eller endda år.
Forudsigelse af den måde, hvorpå potentielle lægemidler og proteinmål binder til hinanden, bruges til at hjælpe med at beslutte, hvilke potentielle lægemidler, der skal syntetiseres og teste i laboratoriet. AlphaFold 3 kan ikke kun forudsige lægemiddelbinding i fravær af en eksperimentelt identificeret proteinstruktur, men i test udkonkurrerede den eksisterende softwareforudsigelser, selvom målstrukturen og lægemiddelbindingsstedet var kendt.
Disse nye muligheder gør AlphaFold 3 til en spændende tilføjelse til repertoiret af værktøjer, der bruges til at opdage nye terapeutiske lægemidler. Mere præcise forudsigelser vil gøre det muligt at træffe bedre beslutninger om, hvilke potentielle lægemidler der skal testes i laboratoriet (og hvilke der sandsynligvis ikke er effektive).
Tid og penge
Dette sparer både tid og penge. AlphaFold 3 giver også mulighed for at komme med forudsigelser om lægemiddelbinding til modificerede former af proteinmålet, som er biologisk relevante, men i øjeblikket vanskelige - eller umulige - at udføre ved hjælp af eksisterende software. Eksempler på dette er proteiner modificeret af kemiske grupper såsom fosfater eller sukkerarter.
Naturligvis, som med ethvert nyt potentielt lægemiddel, er omfattende eksperimentel testning for sikkerhed og virkning – også hos frivillige – altid nødvendig før godkendelse som licenseret medicin.
AlphaFold 3 har nogle begrænsninger. Ligesom sine forgængere er den dårlig til at forudsige adfærden af proteinområder, der mangler en fast eller ordnet struktur. Den er dårlig til at forudsige flere konformationer af et protein (som kan ændre form på grund af lægemiddelbinding eller som en del af dets normale biologi) og kan ikke forudsige proteindynamik.
Det kan også begå nogle lidt pinlige kemiske fejl, såsom at lægge atomer oven på hinanden (fysisk umuligt), og ved at erstatte nogle detaljer i en struktur med dens spejlbilleder (biologisk eller kemisk umuligt).
En mere væsentlig begrænsning er, at koden - i det mindste nu - vil være utilgængelig, så den skal bruges på DeepMind-serveren på et rent ikke-kommercielt grundlag. Selvom mange akademiske brugere ikke vil blive afskrækket af dette, vil det begrænse entusiasmen hos ekspertmodelbyggere, bioteknologer og mange anvendelser inden for lægemiddelopdagelse.
På trods af dette ser udgivelsen af AlphaFold 3 ud til at stimulere en ny bølge af kreativitet inden for både lægemiddelopdagelse og strukturel biologi mere bredt – og vi ser allerede frem til AlphaFold 4.
Leveret af The Conversation
Denne artikel er genudgivet fra The Conversation under en Creative Commons-licens. Læs den originale artikel. 
 Varme artikler
Varme artikler
-
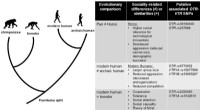 Hvad oxytocin kan fortælle os om udviklingen af menneskelig prosocialitetHypoteserede funktionelle forbindelser mellem identificerede SNPer på OTR-VTRerne og socialitetsrelaterede ændringer / ligheder på tværs af arter / afstamninger. Til venstre, et skematisk træ mellem s
Hvad oxytocin kan fortælle os om udviklingen af menneskelig prosocialitetHypoteserede funktionelle forbindelser mellem identificerede SNPer på OTR-VTRerne og socialitetsrelaterede ændringer / ligheder på tværs af arter / afstamninger. Til venstre, et skematisk træ mellem s -
 Termitter kan have en større rolle i fremtidige økosystemerTræfodrende termitter fra Far North Queensland, Australien. Kredit:Rebecca Clement. Kunstværk af Donna Davis De fleste mennesker tror, at termitter er en plage, der forbruger træ i boliger og vir
Termitter kan have en større rolle i fremtidige økosystemerTræfodrende termitter fra Far North Queensland, Australien. Kredit:Rebecca Clement. Kunstværk af Donna Davis De fleste mennesker tror, at termitter er en plage, der forbruger træ i boliger og vir -
 Tarmbakterie forårsager indirekte symptomer ved at ændre frugtflues mikrobiomEn frugtfluetarm. Kredit:Tiffani Jones CagA, et protein produceret af bakterien Helicobacter pylori , kan ændre populationen af mikrober, der lever i frugtfluens tarm, fører til sygdomssymptom
Tarmbakterie forårsager indirekte symptomer ved at ændre frugtflues mikrobiomEn frugtfluetarm. Kredit:Tiffani Jones CagA, et protein produceret af bakterien Helicobacter pylori , kan ændre populationen af mikrober, der lever i frugtfluens tarm, fører til sygdomssymptom -
 Fugle uden egen yngel hjælper andre fugle med forældre, men ikke uselviskKredit:Seychellen Rietzanger Fugle vil nogle gange passe på afkom fra andre fugle af deres egen art, hvis de forudser fremtidige fordele. At blive tolereret på en anden fugls område og chancen for
Fugle uden egen yngel hjælper andre fugle med forældre, men ikke uselviskKredit:Seychellen Rietzanger Fugle vil nogle gange passe på afkom fra andre fugle af deres egen art, hvis de forudser fremtidige fordele. At blive tolereret på en anden fugls område og chancen for


