Genomisk analyse af en dyreplanktonart sætter spørgsmålstegn ved antagelser om artsdannelse og genregulering
Når to dyr ser ens ud, spiser ens, opfører sig på samme måde og lever i lignende miljøer, kan man forvente, at de tilhører den samme art.
Imidlertid udfordrer et lille dyreplankton, der skimmer havets overflader af mikroskopiske fødevarepartikler, denne antagelse. Forskere fra Osaka University, University of Barcelona og Okinawa Institute of Science and Technology (OIST) har analyseret genomet af Oikopleura dioica fra Seto Indlandshav, Middelhavet og Stillehavet omkring Okinawa-øerne, og ved at gøre det har rejst adskillige spørgsmål om artsdannelse og genplaceringens rolle i genomet.
Deres resultater er blevet offentliggjort i Genome Research . "Oikopleura åbner nye veje til genomisk forskning," siger Dr. Charles Plessy fra Genomics and Regulatory Systems Unit på OIST og medførsteforfatter af papiret.
"Som et modeldyr giver det os mulighed for at studere mekanismerne for genomændringer i laboratoriet, når de sker i meget stor skala og hastighed, hvilket er en enorm mulighed."
Oikopleura dioica er et lille dyreplankton, der bebor havoverfladen verden over, og som bruges som modelorganisme i udviklingsbiologien.
Som en chordat deler organismen centrale genetiske og udviklingsmæssige træk med hvirveldyr, herunder tilstedeværelsen af en notochord, som er et akkord-lignende centralnervebundt som en rygsøjle, men uden knogler. Desuden letter dets kompakte genom, det mindste ikke-parasitære dyregenom, der er rapporteret til dato, storstilet genomisk analyse.
Babels genomiske tårn
Forskerne arbejdede på tre afstamninger af Oikopleura dioica udtaget fra tre have rundt om i verden, men selvom de morfologiske, adfærdsmæssige og økologiske karakteristika af afstamningerne er praktisk talt de samme, er genomerne meget forskellige.
Tænk på genomet som et fælles sprog mellem alle medlemmer af en enkelt art, lagret i kernen af hver celle og indeholder det komplette sæt af genetisk materiale til at lave den art. Ligesom hvordan grammatik bestemmer arrangementet af ord for at formidle specifikke betydninger, så er de grundlæggende informationsenheder i genomet - generne - også reguleret i forhold til hinanden, når de transskriberes og oversættes til livets grundlæggende byggesten, proteiner .
Genregulering involverer flere faktorer, der påvirker aktiveringen eller hastigheden af gentranskription, såsom andre gener, molekyler i cellen, hormoner og mange andre.
Det, der er forvirrende ved Oikopleura dioica-genomet, er, at sprogene i de tre slægter ikke ser ud til at matche, på trods af at de har næsten identiske fysiske egenskaber. Det vil sige, at 'meningen' produceret af deres gener er for det meste den samme, mens de genomiske sprog er meget forskellige mellem dem.
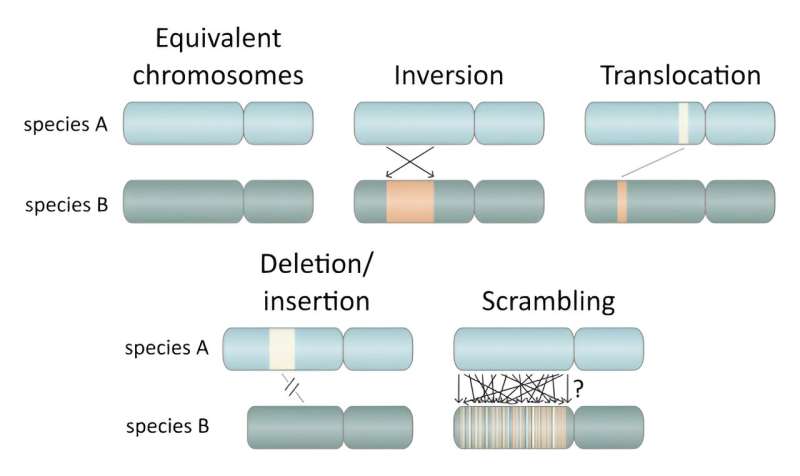
Forskerne bruger udtrykket "scrambling" til at beskrive det fænomen, der observeres i Oikopleura dioica, et udtryk, der stammer fra lingvistik for at betegne et fænomen, hvor sætninger formuleres ved hjælp af en række forskellige ordrækkefølger uden nogen ændring i betydning.
Selvom dette fænomen ikke forekommer på engelsk (men gør det på japansk og andre sprog), ville et engelsk eksempel være, hvis sætningen "genomet af Oikopleura dioica er stærkt forvrænget" kunne omarrangeres til "højt forvrænget Oikopleura dioica, genomet af er" uden betydningsændring. Mens genomiske omlejringer er fælles for alle arter, og genomforvrængning er blevet observeret i nogle få arter over meget lang tid, overgår Oikopleura dioica, hvad man tidligere troede var muligt.
Udvikling i rasende fart
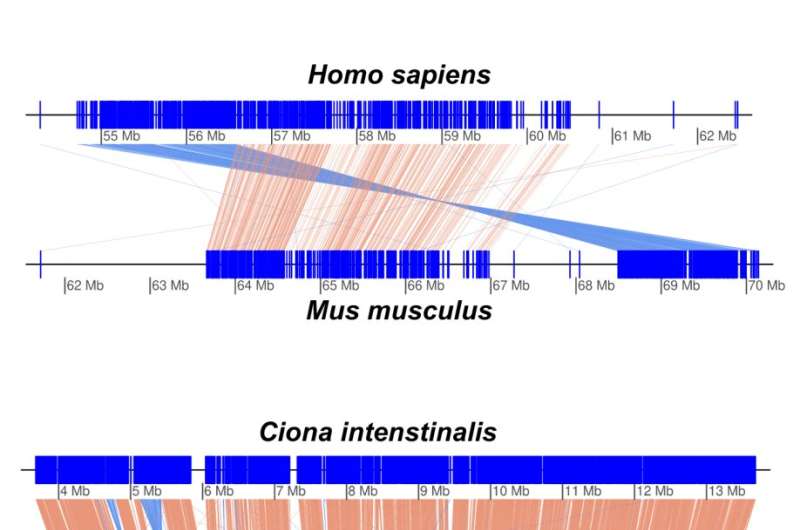
Forskerne sammenlignede de genetiske sekvenser af de tre slægter, hvilket fik dem til at anslå, at de delte en fælles forfader for omkring 25 millioner år siden, hvor slægterne fra Barcelona og Osaka var tættere beslægtede end Okinawa-slægten, idet de havde divergeret ~7 millioner år. siden. Til sammenligning afveg mennesker fra mus for 75-90 millioner år siden.
Ud fra deres fylogenetiske analyser estimerede forskerne hastigheden af genomiske omlejringer for forskellige arter som et kvantificerbart mål for, hvor hurtigt de udvikler sig. Ud fra dette fandt forskerne ud af, at frekvensen for Oikopleura dioica er mere end ti gange højere end sammenlignelige arter af Ciona havsprøjter.
Som Dr. Michael J. Mansfield fra enheden og medførsteforfatter på papiret udtrykker det, "Oikopleura er et af de hurtigst udviklende dyr i verden. Dyr, især chordater, omarrangerer normalt ikke deres genomer i denne grad. , ved denne hastighed."
Med al denne genomforvrængning, der finder sted mellem Oikopleura dioica-slægterne, er det fra et genomisk perspektiv mystificerende, at de kan bevare sådanne ens egenskaber.
"Vores resultater tyder på, at selvom genomisk organisering er vigtig, især for noget så komplekst som mennesker, bør vi ikke glemme de individuelle gener," foreslår Dr. Plessy. At studere gener og genomer kan tilbyde to forskellige perspektiver på det samme fænomen - som Dr. Mansfield forklarer det:"Der er forskere, der studerer anatomi og andre, der studerer individuelle neuroner - men begge besvarer spørgsmål om hjernen."
Hvem spørger?
Genomforvrængning stiller vigtige spørgsmål om evolution og klassificering af liv i arter. På den ene side viser forskerne, at selvom de tre slægter af Oikopleura dioica er praktisk talt identiske morfologisk og funktionelt, er deres genomer ekstremt forvrænget, hvilket kunne tyde på, at de tilhører forskellige arter, selvom forskerne understreger, at deres intention ikke er at klassificere dem her. På den anden side kunne den forvrængede, men analoge genekspression advare mod en overdreven afhængighed af genomik til klassificering af arter.
Men i sidste ende har "arter ikke brug for os. Hvis du fjerner mennesker, er dyrene de samme - det er lige meget, hvordan vi klassificerer dem," som Dr. Plessy formulerer det. I stedet er artsbegrebet flydende, alt efter om det er til bevaringsformål, til lovgivning, som mikrobiolog eller zoolog, eller hvad årsagen nu er. "Spørgsmålet 'hvad er en art?' kan besvares med et andet spørgsmål:hvorfor spørger du?"
For Dr. Plessy, Dr. Mansfield og deres samarbejdspartnere rundt om i verden er dette papir kulminationen på en lang proces med at dyrke forskellige afstamninger af Oikopleura dioica og udvikle bioinformatiske værktøjer, der er i stand til at analysere deres kaotiske genomer. Professor Nicholas Luscombe, leder af enheden ved OIST, er optimistisk med hensyn til forskningspotentialet i undersøgelsen og dyrene.
"Vi antog oprindeligt, at alle Oikopleura ville have lignende genomer, men vi var forbløffede over at se så store forskelle med så meget forvrængning mellem dem. Vi ønsker at bruge Oikopleura til at lære mere om naturen af genomiske omlejringer."
Dette er kun begyndelsen - forskerne er langt fra færdige med at studere det gådefulde zooplankton. "Vi har allerede lært så meget af Oikopleura, men vi mangler endnu at udforske det fulde omfang af arternes mangfoldighed på globalt plan," siger Dr. Plessy.
Dr. Mansfield citerer den store biolog Jacques Monod med "det, der er sandt for E. coli, er sandt for elefanten" - med de værktøjer, der er udviklet til denne undersøgelse, kan holdene nu vende deres opmærksomhed mod andre arter. "Vi gik ind i den tankegang, at alle Oikopleura dioica var ens, men vi har vist det modsatte. Hvor ofte er det sandt for andre arter, og hvor meget mere er der at vide om mekanismerne bag genomforvrængning?"
Flere oplysninger: Charles Plessy et al., Extreme genome scrambling in marine plankonicOikopleura dioicacryptic species, Genome Research (2024). DOI:10.1101/gr.278295.123
Journaloplysninger: Genomforskning
Leveret af Okinawa Institute of Science and Technology
 Varme artikler
Varme artikler
-
 Forskere efterlyser en global bakteriebankIkke alle bakterier er ødelæggende, og nogle forskere ser behovet for at bankere dem til mulig fremtidig brug. Kevin Frayer/Getty Images Folk hader bakterier. Se bare på håndrensningsindustrien, hvor
Forskere efterlyser en global bakteriebankIkke alle bakterier er ødelæggende, og nogle forskere ser behovet for at bankere dem til mulig fremtidig brug. Kevin Frayer/Getty Images Folk hader bakterier. Se bare på håndrensningsindustrien, hvor -
 Lizard release bringer sjældne arter tilbage til deres naturlige habitat i StorbritannienSouthampton PhD-forsker Rachel Gardner vil nøje overvåge en koloni af sjældne sandfirben. Kredit:University of Southampton En ph.d. forsker fra University of Southampton vil nøje overvåge en popul
Lizard release bringer sjældne arter tilbage til deres naturlige habitat i StorbritannienSouthampton PhD-forsker Rachel Gardner vil nøje overvåge en koloni af sjældne sandfirben. Kredit:University of Southampton En ph.d. forsker fra University of Southampton vil nøje overvåge en popul -
 Hvorfor er folks hjerner forskellige størrelser?Brain Image Gallery Er det store hoved mere end bare et enormt ego? Se flere hjernebilleder. Daniel Day/Stone+/Getty Images Der er mange taktikker, folk bruger til at afslutte argumenter og konfron
Hvorfor er folks hjerner forskellige størrelser?Brain Image Gallery Er det store hoved mere end bare et enormt ego? Se flere hjernebilleder. Daniel Day/Stone+/Getty Images Der er mange taktikker, folk bruger til at afslutte argumenter og konfron -
 Svampeudbrud truer trefarvet flagermus med udryddelseDette billede leveret af U.S. Fish and Wildlife Service i september 2022 viser trefarvede flagermus. Tirsdag den 13. september 2022 annoncerede føderale embedsmænd planer om at opføre dyret som truet
Svampeudbrud truer trefarvet flagermus med udryddelseDette billede leveret af U.S. Fish and Wildlife Service i september 2022 viser trefarvede flagermus. Tirsdag den 13. september 2022 annoncerede føderale embedsmænd planer om at opføre dyret som truet
- At omdanne træbark og kompost til flyvinger og plastikposer
- CO2 når sit højeste niveau i mere end 4 millioner år
- Hvordan fotosyntese fanger lys og styrker livet på jorden
- Leids økonofysiske model testet bedst af centralbanker
- De fleste syriske flygtninge længes efter at vende hjem, ikke immigrere
- Sammenligning af kontinentale hydrologiske modeller hjælper med at forbedre vandforvaltningen


