Forskere kortlægger proteinnetværkets dynamik under celledeling
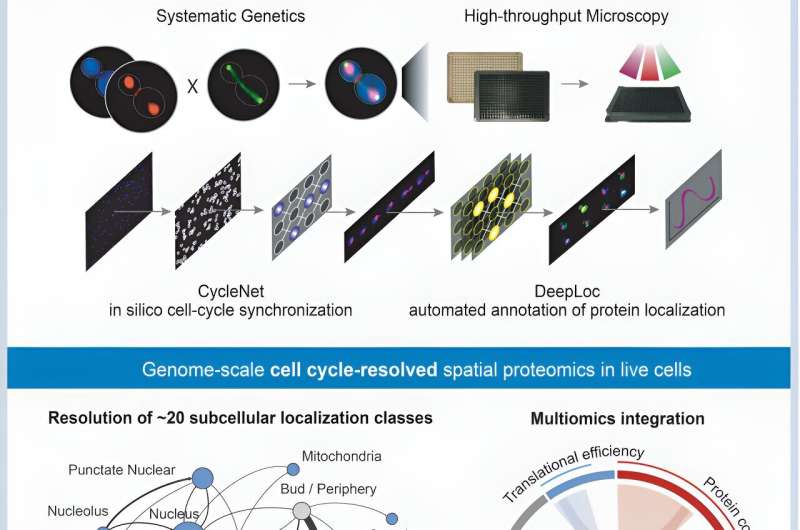
Et internationalt hold ledet af forskere ved University of Toronto har kortlagt bevægelsen af proteiner kodet af gærgenomet gennem dets cellecyklus. Dette er første gang, at alle proteiner i en organisme er blevet sporet på tværs af cellecyklussen, hvilket krævede en kombination af dyb læring og højkapacitetsmikroskopi.
Holdet anvendte to foldede neurale netværk, eller algoritmer, kaldet DeepLoc og CycleNet, til at analysere billeder af millioner af levende gærceller. Resultatet var et omfattende kort, der identificerer, hvor proteiner er placeret, og hvordan de bevæger sig og ændrer sig i overflod i cellen under hver fase af cellecyklussen.
"Vi fandt, at proteiner, der regelmæssigt øger og falder i koncentrationen i cellen, har en tendens til at være involveret i reguleringen af cellecyklussen, mens proteiner med forudsigelig bevægelse gennem cellen har tendens til at lette cyklussens biofysiske implementering," siger Athanasios Litsios, første forfatter på undersøgelsen. og postdoc ved U of T's Donnelly Center for Cellular and Biomolecular Research.
Undersøgelsen blev publiceret i tidsskriftet Cell .
Cellecyklussen forstås som de stadier, hvorigennem en celle skrider frem for i sidste ende at dele sig i separate celler. Det er denne proces, der ligger til grund for spredningen af liv – og er i gang i alt levende.
På molekylært niveau afhænger cellecyklussen af koordineringen af mange proteiner til at transportere cellen fra vækst og DNA-replikation hele vejen til celledeling. Dysregulering af proteiner kan kaste en skruenøgle ind i cellecyklussen, hvor dens forstyrrelse potentielt kan føre til sygdomme som kræft.
Forskerne observerede, at omkring en fjerdedel af kortlagte gærproteiner fulgte regelmæssige mønstre for fremkomst og forsvinden eller bevægelse til specifikke områder af cellen. De fleste proteiner fulgte disse mønstre for enten koncentration eller bevægelse, men ikke begge dele.
"Vi identificerede omkring 400 proteiner med kun periodisk lokalisering i løbet af cellecyklussen og omkring 800 med kun periodisk koncentration," sagde Litsios. "Dette betyder, at proteiner bliver reguleret på flere niveauer for at sikre, at cellecyklussen sker som programmeret."
Forskerholdet brugte fluorescensmikroskopi til at spore omkring 4.000 proteiner i billeder af gærceller for at klassificere cellecyklusfasen såvel som placeringen af proteiner inden for 22 kategoriserede områder af cellen, såsom kerne, cytoplasma og mitokondrier. Fase- og proteinlokationsidentifikation blev automatiseret ved brug af konvolutionelle neurale netværk med en cellecyklusfaseforudsigelsesnøjagtighed på mere end 93 %.
"Vi analyserede billeder af mere end 20 millioner levende gærceller, som vi tildelte forskellige cellecyklusstadier ved hjælp af maskinlæring," sagde Brenda Andrews, hovedforsker på studiet og universitetsprofessor i molekylær genetik ved Donnelly Center og Temerty Faculty of Medicin.
"Vi udviklede og anvendte derefter en anden beregningspipeline til at undersøge, hvordan proteiner ændrer sig i lokalisering og koncentration i løbet af cellecyklussen. Denne undersøgelse producerede et unikt datasæt, der tilbyder en genom-skala visning af molekylære ændringer, der opstår under celledeling."
"Gærcellen er en fantastisk model for eukaryotisk biologi," sagde Litsios. "Der er visse ting, vi kan gøre med gærceller, men ikke med andre organismer, som enten er mere enkle eller komplekse. Vi kan bruge gærceller til at observere processer i stor skala, hvilket gør den til den perfekte organisme til at studere cellecyklussen - i håbet om bedre at forstå den menneskelige cellecyklus."
Flere oplysninger: Athanasios Litsios et al., Proteom-skalabevægelser og kompartmentforbindelse under den eukaryote cellecyklus, Celle (2024). DOI:10.1016/j.cell.2024.02.014
Journaloplysninger: Celle
Leveret af University of Toronto
 Varme artikler
Varme artikler
-
 Der er en undersøgelse om det:Stenalderen havde lige så mange venstreorienteredeForskere undersøgte håndsilhuetmalerier for at finde spor til paleolitisk venstrehåndethed. Prisma/UIG/Getty Images Der var kun så mange ting, vores paleolitiske forfædre kunne gøre for at underholde
Der er en undersøgelse om det:Stenalderen havde lige så mange venstreorienteredeForskere undersøgte håndsilhuetmalerier for at finde spor til paleolitisk venstrehåndethed. Prisma/UIG/Getty Images Der var kun så mange ting, vores paleolitiske forfædre kunne gøre for at underholde -
 Blandede dækafgrøder fanger kulstof i jorden, kan hjælpe med at afbøde klimaændringerForskerne prøvede jord under dækafgrøder, der tilhører tre plantefunktionelle typer, på parceller, der er en del af et langsigtet dækafgrødeeksperiment ved Penn States Russell E. Larson Agricultural R
Blandede dækafgrøder fanger kulstof i jorden, kan hjælpe med at afbøde klimaændringerForskerne prøvede jord under dækafgrøder, der tilhører tre plantefunktionelle typer, på parceller, der er en del af et langsigtet dækafgrødeeksperiment ved Penn States Russell E. Larson Agricultural R -
 Hvad driver økosystemer til ustabilitet?MIT-forskere studerede økosystemer af op til 48 bakteriearter og opdagede, hvordan samfundene skifter fra stabile til ustabile tilstande. Kredit:William Lopes, Gore Lab At forsøge at tyde alle de f
Hvad driver økosystemer til ustabilitet?MIT-forskere studerede økosystemer af op til 48 bakteriearter og opdagede, hvordan samfundene skifter fra stabile til ustabile tilstande. Kredit:William Lopes, Gore Lab At forsøge at tyde alle de f -
 Bekæmpelse af klimaændringer med varmetolerante planterMarkrobotten DeBiFix undersøger hele hvedemarker for at vise, hvordan kernerne udvikler sig inde i ørerne. Dette kan bruges som grundlag for at afgøre, hvilke sorter der er særligt velegnede til foræd
Bekæmpelse af klimaændringer med varmetolerante planterMarkrobotten DeBiFix undersøger hele hvedemarker for at vise, hvordan kernerne udvikler sig inde i ørerne. Dette kan bruges som grundlag for at afgøre, hvilke sorter der er særligt velegnede til foræd
- En platform til at udtrække afgørende information fra satellitbilleder
- Undersøgelse viser, hvordan rester af avocadobeskæring kan bruges til at producere mere bæredygti…
- Miljøproblemer forårsaget af mineraler
- Hvorfor våbenkontrollove ikke vedtager Kongressen på trods af flertallets offentlig støtte og gen…
- Udvikling af smart isotopadskillelsessystem
- Hvordan påvirker produktionsvalg mikrofiberskuret?


