Genom-sekventering afslører, hvordan salmonella udskiller en niche inden for svinekødsproduktion
Abstrakt:
Salmonella enterica serovar Typhimurium (S. Typhimurium) er et vigtigt fødevarebåren patogen, der kan forårsage alvorlig gastroenteritis hos mennesker. Gris er et vigtigt naturligt reservoir og en væsentlig kilde til humane salmonelloseinfektioner verden over. At forstå det genetiske grundlag for S. Typhimurium-tilpasning og kolonisering i grisen er nøglen til at udvikle effektive interventioner til at kontrollere salmonella i svinekødsproduktionssystemer og reducere infektioner hos mennesker. I denne undersøgelse udførte vi helgenomsekventering af 200 S. Typhimurium-isolater opnået fra forskellige kilder langs svinekødsproduktionskæden inklusive svinefarme, slagterier og svinekødsprodukter. Komparativ genomisk analyse afslørede tilstedeværelsen af forskellige genomiske varianter forbundet med hvert trin i produktionskæden, hvilket fremhævede bakteriernes evne til at tilpasse sig de forskellige miljøforhold. Isolater, der huser specifikke virulensgener, såsom dem involveret i intestinal invasion og systemisk spredning, viste sig at være mere udbredt hos grise end i andre kilder, hvilket indikerer den selektive fordel ved disse gener for overlevelse og persistens i værten. Derudover identificerede vi genomiske øer unikke for S. Typhimurium-isolater fra grise, som kunne bidrage til deres tilpasning og kolonisering i denne værtsart. Disse resultater giver ny indsigt i de genetiske mekanismer for salmonellaværts tilpasning og persistens og identificerer potentielle mål for at kontrollere salmonella i svinekødsproduktionen og reducere risikoen for infektion hos mennesker.
 Varme artikler
Varme artikler
-
 Forskere opdager vejen til forbedret bygkvalitetKredit:University of Dundee Forskere fra International Barley Hub har opdaget en genetisk vej til forbedret bygkornstørrelse og ensartethed, et fund, som kan hjælpe forædlere med at udvikle fremti
Forskere opdager vejen til forbedret bygkvalitetKredit:University of Dundee Forskere fra International Barley Hub har opdaget en genetisk vej til forbedret bygkornstørrelse og ensartethed, et fund, som kan hjælpe forædlere med at udvikle fremti -
 Undersøgelse finder, at australske huler er op til 500.000 år ældre, end vi troede, og det kunne …Kredit:Steve Bourne, Forfatter leveret, Forfatter leveret South Australias Naracoorte-huler er et af verdens bedste fossile steder, med en rekord, der strækker sig over mere end en halv million år.
Undersøgelse finder, at australske huler er op til 500.000 år ældre, end vi troede, og det kunne …Kredit:Steve Bourne, Forfatter leveret, Forfatter leveret South Australias Naracoorte-huler er et af verdens bedste fossile steder, med en rekord, der strækker sig over mere end en halv million år. -
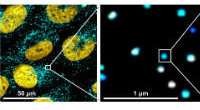 Ny zoomteknik afslører celleelektriske kredsløb for første gangKredit:University of Reading Cellebiologer har brugt en ny superopløsningsmikroskopiteknik til for første gang at kunne observere reaktioner på molekylært niveau. I et blad udgivet i Videnskaben
Ny zoomteknik afslører celleelektriske kredsløb for første gangKredit:University of Reading Cellebiologer har brugt en ny superopløsningsmikroskopiteknik til for første gang at kunne observere reaktioner på molekylært niveau. I et blad udgivet i Videnskaben -
 Undersøgelse viser, hvordan skildpadder har klaret sig et årti efter olieudslipJosh Otten, der i maj dimitterede fra UToledo med en ph.d. i biologi, holder en skildpadde, mens han downloader data til sin computer på Kalamazoo-floden. Otten er hovedforfatter til en ny undersøgels
Undersøgelse viser, hvordan skildpadder har klaret sig et årti efter olieudslipJosh Otten, der i maj dimitterede fra UToledo med en ph.d. i biologi, holder en skildpadde, mens han downloader data til sin computer på Kalamazoo-floden. Otten er hovedforfatter til en ny undersøgels
- Ny forskning afslører, hvordan man afslører løgnere ved distraktion
- EXPLAINER:Kunne Facebook sagsøge whistlebloweren Frances Haugen?
- Energifri superhurtig computing opfundet af forskere, der bruger lysimpulser
- Forskere finger skyldige decimering Frances østers
- Hvad har lasere og appelsiner til fælles? En mulig kur mod citrusgrønning
- Hvordan planter aktiverer deres immunsystem mod patogener i regn


