Deep learning-drevet DeepEC-ramme hjælper med nøjagtigt at forstå enzymfunktioner
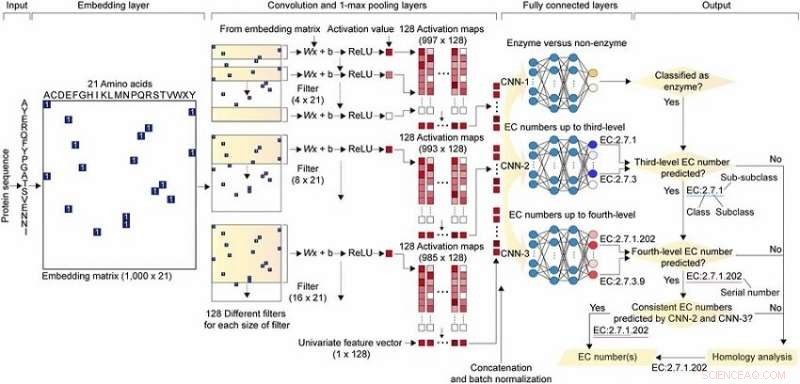
Overordnet skema for DeepEC. Kredit:KAIST
En dyb læringsdrevet beregningsramme, 'DeepEC, ' vil tillade forudsigelse af høj kvalitet og høj gennemstrømning af enzymkommissionstal, som er afgørende for den nøjagtige forståelse af enzymfunktioner.
Et team bestående af Dr. Jae Yong Ryu, Professor Hyun Uk Kim, og den fremtrædende professor Sang Yup Lee ved KAIST rapporterede om den beregningsmæssige ramme drevet af dyb læring, der forudsiger enzymkommission (EC) tal med høj præcision på en måde med høj gennemstrømning.
DeepEC tager en proteinsekvens som input og forudsiger nøjagtigt EC-tal som output. Enzymer er proteiner, der katalyserer biokemiske reaktioner og EC-numre, som består af fire niveautal (dvs. en, b, c, d) angive biokemiske reaktioner. Dermed, identifikation af EC-numre er afgørende for nøjagtigt at forstå enzymfunktioner og metabolisme.
EC-numre gives normalt til en proteinsekvens, der koder for et enzym, under en genomannoteringsprocedure. På grund af vigtigheden af EF-numre, der er udviklet adskillige værktøjer til forudsigelse af EC-nummer, men de har plads til yderligere forbedringer med hensyn til beregningstid, præcision, dækning, og den samlede størrelse af de nødvendige filer til forudsigelse af EC-nummer.
DeepEC bruger tre konvolutionelle neurale netværk (CNN'er) som en hovedmotor til forudsigelse af EC-tal, og implementerer også homologianalyse for EC-numre, hvis de tre CNN'er ikke producerer pålidelige EC-numre for en given proteinsekvens. DeepEC blev udviklet ved at bruge et guldstandarddatasæt, der dækker 1, 388, 606 proteinsekvenser og 4, 669 EF-numre.
I særdeleshed, benchmarkingundersøgelser af DeepEC og fem andre repræsentative værktøjer til forudsigelse af EF-tal viste, at DeepEC lavede de mest præcise og hurtigste forudsigelser for EF-tal. DeepEC krævede også den mindste diskplads til implementering, hvilket gør det til en ideel tredjeparts softwarekomponent.
Desuden, DeepEC var den mest følsomme til at detektere enzymatisk funktionstab som følge af mutationer i domæner/bindingsstedrest af proteinsekvenser; i denne sammenlignende analyse, alle domænerne eller bindingsstedsresterne blev substitueret med L-alaninrest for at fjerne proteinfunktionen, som er kendt som L-alanin-scanningsmetoden.
Denne undersøgelse blev offentliggjort online i Proceedings of the National Academy of Sciences ( PNAS ) den 20. juni, 2019, med titlen "Deep learning muliggør forudsigelse af høj kvalitet og high-throughput af enzymkommissionstal."
"DeepEC kan bruges som et uafhængigt værktøj og også som en tredjeparts softwarekomponent i kombination med andre beregningsplatforme, der undersøger metaboliske reaktioner. DeepEC er frit tilgængeligt online, sagde professor Kim.
Den ærede professor Lee sagde, "Med DeepEC, det er blevet muligt at behandle stadigt stigende mængder af proteinsekvensdata mere effektivt og mere præcist."
 Varme artikler
Varme artikler
-
 Siemens, Alstom rejser tvivl om megafusionAldrig vil de to mødes? Tilliden til, at den politisk følsomme fusion af deres jernbanevirksomheder ville blive vinket igennem af Europa-Kommissionen, ebbede af hos både Siemens og Alstom torsdag.
Siemens, Alstom rejser tvivl om megafusionAldrig vil de to mødes? Tilliden til, at den politisk følsomme fusion af deres jernbanevirksomheder ville blive vinket igennem af Europa-Kommissionen, ebbede af hos både Siemens og Alstom torsdag. -
 Smart handske til Industry 4.0:Forbinder den fysiske hånd til den virtuelle verdenDoktorand Sebastian Gratz-Kelly fra professor Stefan Seeleckes forskergruppe demonstrerer prototypehandsken, der vil blive fremvist på Hannover Messe. Kredit:Oliver Dietze Forskere ved Saarland Un
Smart handske til Industry 4.0:Forbinder den fysiske hånd til den virtuelle verdenDoktorand Sebastian Gratz-Kelly fra professor Stefan Seeleckes forskergruppe demonstrerer prototypehandsken, der vil blive fremvist på Hannover Messe. Kredit:Oliver Dietze Forskere ved Saarland Un -
 Hvorfor vi ikke bør forbyde små køretøjerDen eksploderende popularitet af e-scootere kan omforme mobiliteten i vores byer. Regulatorer skal tilpasse deres tilgange til at håndtere innovationen i stedet for at forbyde den helt. Kredit:Ivan Ma
Hvorfor vi ikke bør forbyde små køretøjerDen eksploderende popularitet af e-scootere kan omforme mobiliteten i vores byer. Regulatorer skal tilpasse deres tilgange til at håndtere innovationen i stedet for at forbyde den helt. Kredit:Ivan Ma -
 Forskere bruger kunstige neurale netværk til at forudsige nye stabile materialerSkematisk over et kunstigt neuralt netværk, der forudsiger en stabil prototype af granatkrystal. Kredit:Weike Ye Kunstige neurale netværk - algoritmer inspireret af forbindelser i hjernen - har læ
Forskere bruger kunstige neurale netværk til at forudsige nye stabile materialerSkematisk over et kunstigt neuralt netværk, der forudsiger en stabil prototype af granatkrystal. Kredit:Weike Ye Kunstige neurale netværk - algoritmer inspireret af forbindelser i hjernen - har læ
- Hvordan bakterier producerer manganoxid-nanopartikler
- Sidste års bedste:De bedste TechXplore-artikler i 2019
- Ny undersøgelse viser tilbagetrækning af det østlige Antarktis islag i tidligere varme perioder
- NASA finder et par stærke storme tilbage i resterne af Fernands over det nordøstlige Mexico
- Et miljøvenligt alternativ til giftige skibsbelægninger
- 10 hold går videre i international kuldioxidkonkurrence


