AlphaFold gør sit præg i at forudsige proteinstrukturer

Kredit:DeepMind
Spillerne klapper, sige ord som Whoo, bank plastikknive i bordet og nyd de bedste weekender med kunstig intelligens som hovedakt, takket være AI udløst i spil.
WIRED UK videnskabsredaktør, Matt Reynolds, så på DeepMinds indvirkning på AI-milepæle:"Det har udspillet Go-mestre, slog professionelle StarCraft-spillere og vendte sin opmærksomhed mod skak og shogi."
Lad legene fortsætte, men de seriøse ting skal for alvor skinne. Kort om, vi kan beundre, at frigørelse af AI med henblik på videnskabelig opdagelse er blevet særligt levende og har det godt takket være forskning hos DeepMind.
Teknikere kommenterede i denne uge forskningsartikler, der viser styrkerne ved AI. "Efterhånden som AI modnes som et felt (og løber tør for videospil at erobre), vil flere af dens resultater sandsynligvis se sådan ud:solide forbedringer inden for vigtige forskningsdomæner."
En forskningsartikel offentliggjort i Natur lavede øjeblikkelige nyheder som et papir om, hvordan AI kan bruges til at forudsige proteinfoldning. VentureBeat henvist til formidable udfordringer, der nærmer sig målene.
Artiklen er "Forbedret forudsigelse af proteinstruktur ved hjælp af potentialer fra dyb læring, "Artiklen blev offentliggjort online den 15. december i Natur .
DeepMind-systemet kaldes AlphaFold. Reynolds havde en interessant note om AlphaFold og datasættet:"Proteinfoldningsfeltet er også godt sat op til træning af kunstigt intelligente midler. Det har et stort datasæt - Protein Data Bank, et lager af 3D-strukturen og den genetiske sammensætning af 150, 000 proteiner, som blev brugt til at træne DeepMinds proteinstruktur-forudsigende system, kaldet AlphaFold."
Papiret udforskede DeepMinds resultater indtil videre med at bruge AI til at forudsige proteinfoldning. Det store ved proteinfoldning er, at som Kelsey Piper i Vox bemærkede, det er et "afgørende spørgsmål for at udvikle nye lægemidler."
Tre fra DeepMind-teamet, Andrew Senior, John Jumper og Demis Hassabis, postede et blogindlæg den 15. januar, der forklarer proteinstrukturer, proteinfoldningsudfordringen og hvad de ønskede at opnå.
"Som demonstreret af Levinthals paradoks, det ville tage længere tid end det kendte univers' alder at tilfældigt opregne alle mulige konfigurationer af et typisk protein, før de når den sande 3-D-struktur - alligevel folder proteiner selv spontant, inden for millisekunder. At forudsige, hvordan disse kæder vil folde sig ind i et proteins indviklede 3D-struktur, er det, der er kendt som 'proteinfoldningsproblemet' – en udfordring, som videnskabsmænd har arbejdet på i årtier."
Holdet viste, at de kunne træne et neuralt netværk til at lave nøjagtige forudsigelser "af afstandene mellem par af rester, som formidler mere information om strukturen end kontaktforudsigelser."
De talte om optimering via "en simpel gradient-nedstigningsalgoritme til at generere strukturer uden komplekse prøveudtagningsprocedurer."
Det store ved proteinfedtning er, at sagde Piper, det er et "afgørende spørgsmål for at udvikle nye lægemidler." Hvordan det? Og, hvorfor er "former" så vigtige? "Den form, som proteiner tager, forudsiger, hvilke andre stoffer de vil interagere med, så forståelse af proteinfoldning er afgørende for lægemiddelopdagelse og kan endda bruges til at udvikle nye fremstillingsprocesser, " sagde Piper.
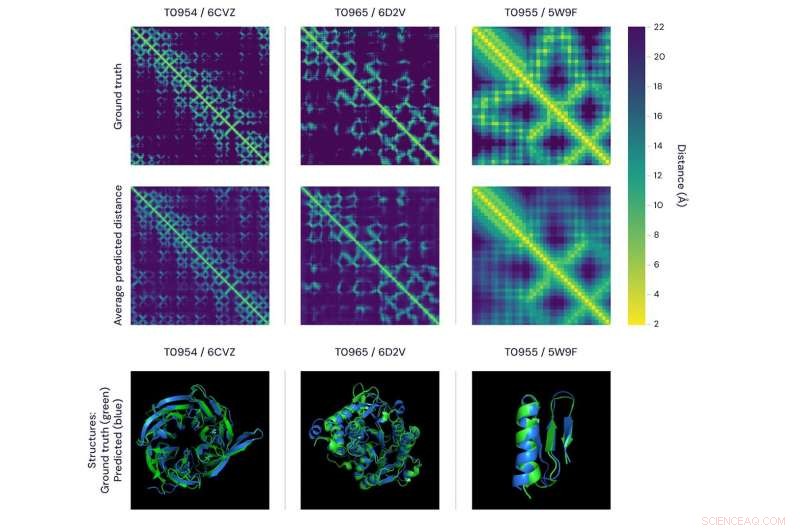
To måder at visualisere nøjagtigheden af alphafolds forudsigelser. den øverste figur viser afstandsmatricerne for tre proteiner. Lysstyrken af hver pixel repræsenterer afstanden mellem aminosyrerne i sekvensen, der består af proteinet – jo lysere pixel, jo tættere parret. I øverste række er de rigtige, eksperimentelt bestemte afstande og, i nederste række, gennemsnittet af AlphaFolds forudsagte afstandsfordelinger. Vigtigt, disse matcher godt på både global og lokal skala. bundpanelerne repræsenterer den samme sammenligning ved brug af 3D-modeller, med AlphaFolds forudsigelser (blå) versus jordsandhedsdata (grøn) for de samme tre proteiner. Kredit:DeepMind
DeepMind-bloggen sagde, at "et proteins form menes at diktere dets funktion. Når først et proteins form er forstået, dens rolle i cellen kan gættes på, og forskere kan udvikle lægemidler, der arbejder med proteinets unikke form."
AlphaFold kunne hjælpe forskere med at få et bedre kendskab til proteinernes funktion - og fejlfunktion.
Et bemærkelsesværdigt citat i VentureBeat fra lederen af UCL-bioinformatikgruppen David Jones, der rådgav DeepMind-teamet om dele af projektet:
"Eksperimentelle teknikker til at bestemme proteinstrukturer er tidskrævende og dyre, så der er en enorm efterspørgsel efter bedre computeralgoritmer til at beregne strukturerne af proteiner direkte fra de gensekvenser, der koder for dem, og DeepMinds arbejde med at anvende AI på dette langvarige problem i molekylærbiologi er et klart fremskridt."
Hvilken slags eksperimentelle teknikker har været tidskrævende og dyre? Bloggen nævnte kryo-elektronmikroskopi, kernemagnetisk resonans og røntgenkrystallografi, hver afhængig af forsøg og fejl, "hvilket kan tage mange års arbejde, og koster titusinder eller hundredtusindvis af dollars pr. proteinstruktur."
Kyle Wiggers ind VentureBeat :"Med AlphaFold, DeepMinds forskerhold fokuserede på problemet med at modellere målformer fra bunden uden at trække på løste proteiner som skabeloner."
Hvordan naturen definerer proteinfoldning:"Proteinfoldning er den proces, hvorved proteiner opnår deres modne funktionelle (native) tertiære struktur, og begynder ofte co-translationelt. Proteinfoldning kræver chaperoner og involverer ofte trinvis etablering af regulære sekundære og supersekundære strukturer, nemlig α-helixer og β-sheets, der folder sig hurtigt, stabiliseret af hydrogenbinding og disulfidbroer, og derefter tertiær struktur."
Tilbage i juli, før udgivelsen af DeepMind-avisen, Natur havde allerede noteret sig, at "kapløbet om at knække en af biologiens største udfordringer - at forudsige 3D-strukturerne af proteiner ud fra deres aminosyresekvenser - intensiveres, takket være nye kunstig intelligens (AI) tilgange."
DeepMind blogindlægget sagde, at de gjorde kode tilgængelig (GitHub), og tilbød linket, for alle, der ønsker at lære mere eller kopiere deres resultater.
I deres blogindlæg sagde de, hvorfor deres system betyder noget:"Vores system, AlphaFold... bygger på årtiers tidligere forskning ved hjælp af store genomiske datasæt til at forudsige proteinstruktur. De 3-D-modeller af proteiner, som AlphaFold genererer, er langt mere nøjagtige end nogen, der er kommet før."
Bevæger sig fremad, bloggen sagde, at et værktøj som AlphaFold kan hjælpe forskere med sjældne sygdomme med at forudsige formen af et protein af interesse hurtigt og økonomisk og i sidste ende kan hjælpe dem med at bidrage til effektiv opdagelse af lægemidler, samtidig med at omkostningerne forbundet med eksperimenter reduceres.
Forskning i sjældne sygdomme er ikke det eneste område, der kan drage fordel af det, de har opnået. Bloggen talte om forurenende stoffer som plastik og olie; fremskridt inden for biologisk nedbrydelige enzymer, der er muliggjort af proteindesign, kan hjælpe med at nedbryde affald på måder, der er mere miljøvenlige.
"Faktisk, forskere er allerede begyndt at konstruere bakterier til at udskille proteiner, der vil gøre affald biologisk nedbrydeligt, og lettere at behandle, " ifølge bloggen.
© 2020 Science X Network
 Varme artikler
Varme artikler
-
 Sydkorea lancerer første nationale 5G -netværk - to dage for tidligtTre førende sydkoreanske udbydere sparkede deres 5G -netværk tidligt i gang for at få et spring på deres amerikanske kolleger Sydkorea lancerede verdens første landsdækkende 5G -mobilnet to dage f
Sydkorea lancerer første nationale 5G -netværk - to dage for tidligtTre førende sydkoreanske udbydere sparkede deres 5G -netværk tidligt i gang for at få et spring på deres amerikanske kolleger Sydkorea lancerede verdens første landsdækkende 5G -mobilnet to dage f -
 I 2020, Californiere vil have nye privatlivsrettigheder online. Men de skal muligvis vise IDKredit:CC0 Public Domain Internettet kommer til at se ud, og arbejde, lidt anderledes fra i dag. Det er fordi californiere har nye rettigheder over, hvordan deres personlige oplysninger indsamles,
I 2020, Californiere vil have nye privatlivsrettigheder online. Men de skal muligvis vise IDKredit:CC0 Public Domain Internettet kommer til at se ud, og arbejde, lidt anderledes fra i dag. Det er fordi californiere har nye rettigheder over, hvordan deres personlige oplysninger indsamles, -
 At bygge computere, som vores hjerner fungererKredit:CC0 Public Domain Vi nærmer os grænsen for, hvor meget flere mikroprocessorer der kan udvikles. Gunnar Tufte foreslår at bygge computere på en helt ny måde, inspireret af den menneskelige h
At bygge computere, som vores hjerner fungererKredit:CC0 Public Domain Vi nærmer os grænsen for, hvor meget flere mikroprocessorer der kan udvikles. Gunnar Tufte foreslår at bygge computere på en helt ny måde, inspireret af den menneskelige h -
 Personlig DJ-musikafspilningsliste-algoritme matcher sange til lyttere, der skifter stemningMaytal Saar-Tsechansky, professor i information, Risiko, og Operations Management på McCombs School of Business, sammen med et par datalogiske forskere på universitetet, skabt en personlig DJ. Deres n
Personlig DJ-musikafspilningsliste-algoritme matcher sange til lyttere, der skifter stemningMaytal Saar-Tsechansky, professor i information, Risiko, og Operations Management på McCombs School of Business, sammen med et par datalogiske forskere på universitetet, skabt en personlig DJ. Deres n
- Opfanget vand, kuldioxid fra bilers udstødning kan hjælpe med at dyrke mad
- Brug af maskinlæring til at rekonstruere forringede Van Gogh-tegninger
- Filippinernes oversvømmelser styrker 66, 000 fra hjem
- Elon Musks nye tunnel lidt ru rundt om kanterne (Opdatering)
- Hvordan man skriver en sats lov i Chemistry
- Sådan kalibrerer du Digiweigh Scale


