Superopløsningsmikroskopi udnytter digital skærmteknologi
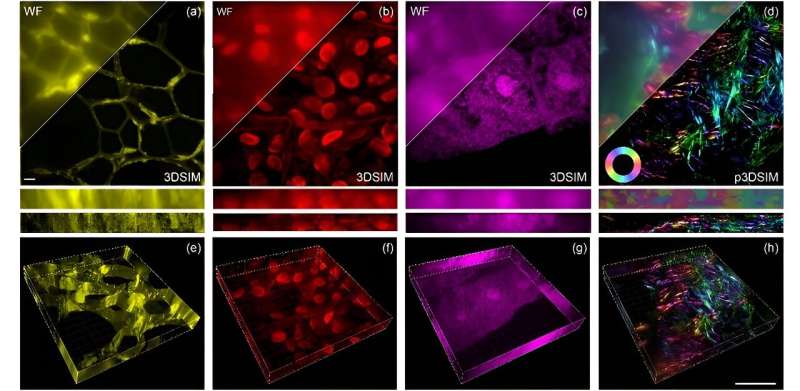
I det stadigt udviklende område af mikroskopi har de seneste år været vidne til bemærkelsesværdige fremskridt inden for både hardware og algoritmer, hvilket driver vores evne til at udforske livets uendelig lille vidundere. Rejsen mod tredimensionel struktureret belysningsmikroskopi (3DSIM) er imidlertid blevet hæmmet af udfordringer, der opstår som følge af hastigheden og indviklede polarisationsmodulering.
Gå ind i højhastighedsmodulations 3DSIM-systemet "DMD-3DSIM", der kombinerer digital skærm med superopløsningsbilleddannelse, så videnskabsmænd kan se cellulære strukturer i hidtil usete detaljer.
Som rapporteret i Advanced Photonics Nexus , udviklede professor Peng Xis team ved Peking University dette innovative setup omkring en digital mikrospejlenhed (DMD) og en elektro-optisk modulator (EOM). Den tackler opløsningsudfordringer ved væsentligt at forbedre både lateral (side-til-side) og aksial (top-til-bund) opløsning, for en 3D rumlig opløsning, der angiveligt er dobbelt så høj som opnået med traditionelle bredfeltsbilledteknikker.
Rent praktisk betyder dette, at DMD-3DSIM kan fange indviklede detaljer af subcellulære strukturer, såsom det nukleare porekompleks, mikrotubuli, actinfilamenter og mitokondrier i dyreceller. Systemets anvendelse blev udvidet til at studere meget spredte plantecelle-ultrastrukturer, såsom cellevægge i oleanderblade og hule strukturer i sorte algeblade. Selv i en musenyreskive afslørede systemet en udtalt polarisationseffekt i actinfilamenter.
En åben gateway til opdagelse
Det, der gør DMD-3DSIM endnu mere spændende, er en forpligtelse til åben videnskab. Xi's team har gjort alle hardwarekomponenter og kontrolmekanismer åbent tilgængelige på GitHub, hvilket fremmer samarbejde og opmuntrer det videnskabelige samfund til at bygge videre på denne teknologi.
DMD-3DSIM-teknikken letter ikke kun betydelige biologiske opdagelser, men lægger også grunden til den næste generation af 3DSIM. I applikationer, der involverer live cell imaging, lover fremskridt inden for lysere og mere fotostabile farvestoffer, denoising-algoritmer og deep learning-modeller baseret på neurale netværk at forbedre billedbehandlingsvarighed, informationshentning og realtidsgendannelse af 3DSIM-billeder fra støjende data. Ved at kombinere hardware- og softwareåbenhed håber forskerne at bane vejen for fremtiden for multidimensionel billeddannelse.
Flere oplysninger: Yaning Li et al, højhastigheds autopolariseringssynkroniseringsmodulation tredimensionel struktureret belysningsmikroskopi, Advanced Photonics Nexus (2023). DOI:10.1117/1.APN.3.1.016001
Leveret af SPIE
 Varme artikler
Varme artikler
-
 Ny holografisk teknik åbner vejen for kvanteberegningKredit:CC0 Public Domain Fotografering måler, hvor meget lys af forskellig farve, der rammer den fotografiske film. Imidlertid, lys er også en bølge, og er derfor præget af fasen. Fase angiver pla
Ny holografisk teknik åbner vejen for kvanteberegningKredit:CC0 Public Domain Fotografering måler, hvor meget lys af forskellig farve, der rammer den fotografiske film. Imidlertid, lys er også en bølge, og er derfor præget af fasen. Fase angiver pla -
 5 Forvirrende subatomære partiklerGaderen, Jokeren, Catwoman and the Penguin ham det op i filmen Batman fra 1966. Se flere Batman -billeder. Silver Screen Collection/Contributor/Getty Images Batman og partikelfysikere har meget tilfæ
5 Forvirrende subatomære partiklerGaderen, Jokeren, Catwoman and the Penguin ham det op i filmen Batman fra 1966. Se flere Batman -billeder. Silver Screen Collection/Contributor/Getty Images Batman og partikelfysikere har meget tilfæ -
 Hvordan kvantemekanik arbejder for at beskytte sikkerhed onlineHovedforfatter, Phil Sibson i laboratoriet. Kredit:University of Bristol Krypterede og uoverskuelige beskeder er rygraden i internettet, som vi kender det. Kompleks kryptografi beskytter vores ba
Hvordan kvantemekanik arbejder for at beskytte sikkerhed onlineHovedforfatter, Phil Sibson i laboratoriet. Kredit:University of Bristol Krypterede og uoverskuelige beskeder er rygraden i internettet, som vi kender det. Kompleks kryptografi beskytter vores ba -
 Forskere øger følsomheden af terahertz -gasanalyser væsentligtForskere udviklede et nyt optisk hulrum, der forbedrer følsomheden af terahertz -spektroskopi. Det tillader molekyler under analyse at interagere med terahertz -frekvenserne over en effektiv afstand
Forskere øger følsomheden af terahertz -gasanalyser væsentligtForskere udviklede et nyt optisk hulrum, der forbedrer følsomheden af terahertz -spektroskopi. Det tillader molekyler under analyse at interagere med terahertz -frekvenserne over en effektiv afstand
- Hvilke elementer reagerer med saltsyre?
- Sådan finder du længden på siden af en trekant, hvis du kender de andre to sider
- Kvælstoffikserende træer hjælper tropiske skove med at vokse hurtigere og lagre mere kulstof
- Proteiner fanget i aktion i intakte celler ved hjælp af ny elektronmikroskopiteknik
- CycleMatch:en ny tilgang til at matche billeder og tekst
- Interaktivt kort over masseopstande rundt om i verden viser ikke-voldsværker


