Benzen-baserede prober fremhæver to skjulte bindingssteder på et anticancer-lægemiddelmål
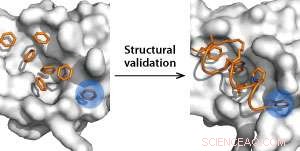
Bindingslommedetektion ved hjælp af benzenmolekyler som prober (venstre) og strukturel validering ved brug af hæftede peptider (højre). Kredit:Y. S. Tan et al.
I jagten på nye kræftbehandlinger, A*STAR-forskere har udtænkt en beregningsstrategi, der afdækker eventuelle hidtil ukendte bindingssteder eller 'lommer' på lægemiddelmål.
Mere effektive kræftbehandlinger vil sandsynligvis dukke op fra lægemiddeludviklingspipelinen. Opdagelse af kræftlægemidler afhænger af at identificere og karakterisere bindingslommer i målproteiner. Typisk, denne evaluering bruger beregningsteknikker, der er afhængige af statiske proteinstrukturer. Imidlertid, proteiner har en iboende fleksibilitet, der forårsager en tendens til at ændre form ved kontakt med stofferne. Visse bindingslommer forbliver uopdagelige, medmindre de interagerer med et passende stof og, derfor, er savnet af konventionelle simuleringer. Disse skjulte lommer, imidlertid, er normalt vandafvisende eller hydrofobe steder, der kun åbner, når der er stoffer med lav polaritet.
For at tackle dette, Yaw Sing Tan og Chandra Verma fra Bioinformatics Institute har udviklet en probe-baseret metode kaldet ligand-mapping molecular dynamics (LMMD). De brugte denne teknik til at søge skjulte bindingslommer i anticancer-målproteinet MDM2. De resulterende forudsigelser blev eksperimentelt valideret af mangeårige samarbejdspartnere fra A*STAR's p53 Laboratory og Institute of Chemical and Engineering Sciences samt strukturelle biologer fra Newcastle University, Storbritannien.
Tan forklarer, at han oprindeligt havde designet denne probe-baserede metode til et andet målprotein og med succes brugte den til at finde en skjult bindingslomme, der forblev lukket i konventionelle simuleringer. "Vi besluttede derefter at anvende denne tilgang til MDM2 for at se, om vi kunne opdage nogen hidtil ukendte bindingssteder, der kunne øge styrken af eksisterende MDM2-hæmmere, " tilføjer han.
Brug af benzenmolekyler som hydrofobe lommedetektionsonder, forskerne identificerede beregningsmæssigt to nye bindingssteder på MDM2. "Vi var spændte på at se, at disse steder ligger meget tæt på bindingslommen af tumorundertrykkerproteinet p53, " siger Tan.
Desuden, forskerne forventer, at de nyfundne steder vil føre til mere potente hæftede peptider – disse er aminosyrespiraler, der er kemisk stabiliseret af en kulbrintekæde, der for nylig er dukket op som kraftige p53-aktivatorer. Følgelig, de skabte hæftede peptider fra analoger kendt for at binde MDM2 tæt og reaktivere p53, og bestemte affiniteten af disse peptider til MDM2. Deres simuleringer viste, at peptiderne bandt MDM2 stærkere end p53 i lommerne og matchede biofysiske og røntgenkrystallografiske eksperimenter.
"Denne metode kunne bruges til at udspørge andre anticancerproteinmål for at afdække nye bindingssteder, der kunne målrettes for inhibering, " siger Tan. Holdet arbejder nu på at udvide rækkevidden af LMMD-prober til andre ligandtyper.
 Varme artikler
Varme artikler
-
 Hvorfor brænder Wood Pop & Crackle?De poppende og knitrende lyde fra en træbrand er så betryggende og hyggelige, at de er blevet markedsført i DVD-form til glæde for lejlighedsboere og andre, der ikke er i stand til at have en rigtig i
Hvorfor brænder Wood Pop & Crackle?De poppende og knitrende lyde fra en træbrand er så betryggende og hyggelige, at de er blevet markedsført i DVD-form til glæde for lejlighedsboere og andre, der ikke er i stand til at have en rigtig i -
 Vågn op og duft til kaffen:Forskning viser, at frysebønner kan bevare aromaenAndrew Cotter demonstrerer den projektive kortlægningsøvelse, der blev brugt i hans forskning. De mørktonede vinglas indeholder de forskellige malede kaffer, der blev brugt i undersøgelsen. Kredit:Pen
Vågn op og duft til kaffen:Forskning viser, at frysebønner kan bevare aromaenAndrew Cotter demonstrerer den projektive kortlægningsøvelse, der blev brugt i hans forskning. De mørktonede vinglas indeholder de forskellige malede kaffer, der blev brugt i undersøgelsen. Kredit:Pen -
 Team opdager, at polymorfe selektion under krystalvækst kan være termodynamisk drevetSnapshots fra MD-simuleringer af selvmontering med interaktionsstyrker EAA/EAB =EBB/EAB =0,15, temperatur kBT/EAB =0,1, og tæthed rs2 =0,1. Eksempler på kvadrat-til-hexagonale transformationer, der fo
Team opdager, at polymorfe selektion under krystalvækst kan være termodynamisk drevetSnapshots fra MD-simuleringer af selvmontering med interaktionsstyrker EAA/EAB =EBB/EAB =0,15, temperatur kBT/EAB =0,1, og tæthed rs2 =0,1. Eksempler på kvadrat-til-hexagonale transformationer, der fo -
 Den højeste varmebestandige plast nogensinde er udviklet af biomasseFigur 1. Udviklingsstrategi for cellulose-afledt PBI og PBI/PA film med ultrahøj termomodstand og rammehæmning. Kredit:Japan Advanced Institute of Science and Technology Brugen af biomasseafledt
Den højeste varmebestandige plast nogensinde er udviklet af biomasseFigur 1. Udviklingsstrategi for cellulose-afledt PBI og PBI/PA film med ultrahøj termomodstand og rammehæmning. Kredit:Japan Advanced Institute of Science and Technology Brugen af biomasseafledt
- Hvordan man fortæller forskellen mellem mandlige og kvindelige kalkuner
- Studiet hjælper med at forstå usynlige, men mægtige partikler
- Forskere identificerer et metal, der kan modstå ultrahøje temperaturer og tryk
- Syntetisk tosidet gekkoerfod kunne muliggøre undervandsrobotik
- Maksimering af MXenes potentiale
- Nanobelter understøtter manipulation af lys


