Computerprogram kan oversætte en 2-D-tegning i fri form til en DNA-struktur

Forskere fra MIT og Arizona State University har skabt et computerprogram, der kan oversætte tegninger af vilkårlige former til todimensionelle strukturer lavet af DNA. Kredit:Hyungmin Jun
Forskere ved MIT og Arizona State University har designet et computerprogram, der giver brugerne mulighed for at oversætte enhver tegning i fri form til en todimensionel, nanoskala struktur lavet af DNA.
Indtil nu, at designe sådanne strukturer har krævet teknisk ekspertise, der sætter processen uden for rækkevidde for de fleste mennesker. Ved at bruge det nye program, alle kan skabe en DNA-nanostruktur af enhver form, til anvendelser inden for cellebiologi, fotonik, og kvanteregistrering og databehandling, blandt mange andre.
"Det, dette arbejde gør, er at give enhver mulighed for at tegne bogstaveligt talt enhver 2D-form og automatisk konvertere den til DNA-origami, " siger Mark Bathe, en lektor i biologisk ingeniørvidenskab ved MIT og seniorforfatter af undersøgelsen.
Forskerne offentliggjorde deres resultater i 4. januar-udgaven af Videnskabens fremskridt , og programmet, kaldet PERDIX, er tilgængelig online. Hovedforfatterne af papiret er Hyungmin Jun, en MIT postdoc, og Fei Zhang, en assisterende forskningsprofessor ved Arizona State University. Andre forfattere er MIT-forskningsassocieret Tyson Shepherd, nyligt MIT Ph.D. modtager Sakul Ratanalert, ASU assisterende forsker Xiaodong Qi, og ASU-professor Hao Yan.
Automatiseret design
DNA origami, videnskaben om at folde DNA til små strukturer, opstod i begyndelsen af 1980'erne, da Ned Seeman fra New York University foreslog at udnytte DNA's baseparringsevner til at skabe vilkårlige molekylære arrangementer. I 2006 Paul Rothemund fra Caltech skabte det første stillads, todimensionelle DNA-strukturer, ved at væve en lang enkelt DNA-streng (stilladset) gennem formen, således at DNA-strenge kendt som "hæfteklammer" ville hybridisere til det for at hjælpe den overordnede struktur med at bevare sin form.
Andre brugte senere en lignende tilgang til at skabe komplekse tredimensionelle DNA-strukturer. Imidlertid, alle disse anstrengelser krævede kompliceret manuel design for at føre stilladset gennem hele strukturen og generere sekvenserne af hæftestrengene. I 2016 Bathe og hans kolleger udviklede en måde at automatisere processen med at generere en 3-D polyhedral DNA-struktur, og i denne nye undersøgelse, de satte sig for at automatisere designet af vilkårlige 2-D DNA-strukturer.
For at opnå det, de udviklede en ny matematisk tilgang til processen med at føre det enkeltstrengede stillads gennem hele strukturen for at danne den korrekte form. Det resulterende computerprogram kan tage en hvilken som helst fri-form tegning og oversætte den til DNA-sekvensen for at skabe den form og til sekvenserne for hæftestrengene.
Formen kan skitseres i et hvilket som helst computertegneprogram og derefter konverteres til en computer-aided design (CAD) fil, som føres ind i DNA-designprogrammet. "Når du har den fil, alt er automatisk, meget ligesom udskrivning, men her er blækket DNA, " siger Bade.
Efter sekvenserne er genereret, brugeren kan bestille dem til nemt at fremstille den specificerede form. I denne avis, forskerne skabte former, hvor alle kanterne består af to duplekser af DNA, men de har også et arbejdsprogram, der kan bruge seks duplekser pr. kant, som er mere stive. Det tilsvarende softwareværktøj til 3-D polyedre, kaldet TALOS, er tilgængelig online og vil snart blive offentliggjort i tidsskriftet ACS Nano. Formerne, som varierer fra 10 til 100 nanometer i størrelse, kan forblive stabil i uger eller måneder, suspenderet i en bufferopløsning.
"Det faktum, at vi kan designe og fremstille disse på en meget enkel måde, er med til at løse en stor flaskehals på vores felt, " siger Bathe. "Nu kan feltet gå over i retning af meget bredere grupper af mennesker i industrien og den akademiske verden, der er i stand til at funktionalisere DNA-strukturer og implementere dem til forskellige anvendelser."
Nanoskala mønstre
Fordi forskerne har så præcis kontrol over strukturen af de syntetiske DNA-partikler, de kan vedhæfte en række andre molekyler på bestemte steder. Dette kunne være nyttigt til skabeloner af antigener i nanoskalamønstre for at kaste lys over, hvordan immunceller genkender og aktiveres af specifikke arrangementer af antigener fundet på vira og bakterier.
"Hvordan nanoskalamønstre af antigener genkendes af immunceller er et meget dårligt forstået område af immunologi, "Siger Bathe. "At vedhæfte antigener til strukturerede DNA-overflader for at vise dem i organiserede mønstre er en effektiv måde at undersøge den biologi på."
En anden nøgleapplikation er at designe lysindsamlingskredsløb, der efterligner de fotosyntetiske komplekser, der findes i planter. For at opnå det, forskerne vedhæfter lysfølsomme farvestoffer kendt som kromoforer til DNA-stilladser. Ud over at høste lys, sådanne kredsløb kunne også bruges til at udføre kvanteregistrering og rudimentære beregninger. Hvis det lykkes, disse ville være de første kvanteberegningskredsløb, der kan fungere ved stuetemperatur, siger Bade.
 Varme artikler
Varme artikler
-
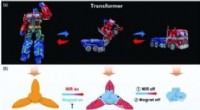 Magnetisk styret, hydrogelbaserede smarte transformerea) Billeder, der viser en transformers formtransformation. b) Formtransformationsprocessen for en blød hydrogeltransformator under koblingen af magnetfelt og NIR. c) SEM -billederne af HG -Fe3O4 -hy
Magnetisk styret, hydrogelbaserede smarte transformerea) Billeder, der viser en transformers formtransformation. b) Formtransformationsprocessen for en blød hydrogeltransformator under koblingen af magnetfelt og NIR. c) SEM -billederne af HG -Fe3O4 -hy -
 Denne bærbare enhed camouflerer sin bærer uanset vejretEnheden, som er på proof-of-concept-stadiet, har en overflade, der hurtigt afkøles eller opvarmes, så den passer til omgivelsestemperaturerne, camouflerer bærerens kropsvarme. Overfladen kan gå fra 10
Denne bærbare enhed camouflerer sin bærer uanset vejretEnheden, som er på proof-of-concept-stadiet, har en overflade, der hurtigt afkøles eller opvarmes, så den passer til omgivelsestemperaturerne, camouflerer bærerens kropsvarme. Overfladen kan gå fra 10 -
 En måde at bruge vand til at omdanne metan til methanolKugle og pind model af metan. Kredit:Ben Mills/Public Domain (Phys.org) – Et team af forskere fra Paul Scherrer Institut og ETH Zürich, både i Schweiz, har udviklet en et-trins proces, der bruger
En måde at bruge vand til at omdanne metan til methanolKugle og pind model af metan. Kredit:Ben Mills/Public Domain (Phys.org) – Et team af forskere fra Paul Scherrer Institut og ETH Zürich, både i Schweiz, har udviklet en et-trins proces, der bruger -
 Ny katalysator kunne muliggøre bedre lithium-svovl-batterier, power næste generations elektronikKredit:CC0 Public Domain I hjertet af det meste af elektronik i dag er genopladelige lithium-ion-batterier (LIBer). Men deres energilagringskapacitet er ikke nok til storskala energilagringssystem
Ny katalysator kunne muliggøre bedre lithium-svovl-batterier, power næste generations elektronikKredit:CC0 Public Domain I hjertet af det meste af elektronik i dag er genopladelige lithium-ion-batterier (LIBer). Men deres energilagringskapacitet er ikke nok til storskala energilagringssystem
- Hvordan cryptocurrency-svindel fungerer
- Nyt lavprofileret ankeleksoskelet passer under tøj til potentiel bred anvendelse
- Typer af talemønstre i Math
- Forskere anvender twistronics til lysudbredelse og gør et gennembrud
- Nye nanoskalateknologier kan revolutionere mikroskoper, undersøgelse af sygdom
- Undersøgelse identificerer ny vej for Grønlands smeltevand til at nå havet


