Ny teknik til bestemmelse af afstand i celler
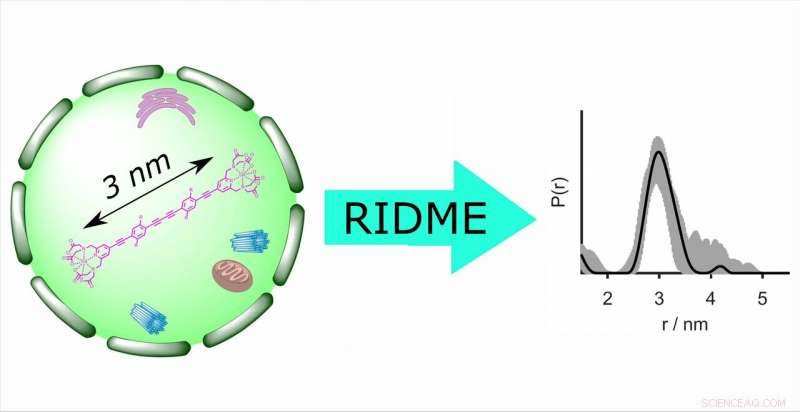
In-celleafstandsbestemmelse ved EPR afslører væsentlig strukturel information om biomakromolekyler under native betingelser. For første gang, den pulserede EPR -teknik RIDME (relaxation induced dipolar modulation enhancement) blev anvendt til afstandsmålinger inde i celler. Det giver en fem gange forbedret følsomhed i forhold til den tidligere anvendte dobbelte elektron-elektron-resonansmetode. Kredit:Forskningsgruppen professor Malte Drescher, Universitetet i Konstanz
I et fælles papir, forskere fra universitetet i Konstanz, Bielefeld University og ETH Zürich demonstrerer for første gang, at elektronparamagnetisk resonans (EPR) teknik RIDME (afslapningsinduceret dipolær moduleringsforbedring) kan anvendes til at bestemme afstande mellem gadolinium (III) -baserede spinetiketter i celler. Bestemmelse af afstand i celler ved elektronparamagnetisk resonans (EPR) afslører væsentlig strukturel information om biomakromolekyler, herunder deres konformation samt folde- og udfoldelsesprocesser.
Konventionelle metoder til bestemmelse af afstande i celler som dobbelt elektron-elektronresonans (DEER eller PELDOR) er hovedsageligt meget mindre følsomme end RIDME, give op til fem gange mindre modulationsdybder, har visse begrænsninger med hensyn til excitationsbåndbredde og er teknisk mere krævende. Som en enkeltfrekvent teknik, der gør brug af afslapningsinducerede spinflips til at bestemme afstanden mellem to spinetiketter, dvs. mellem to uparede elektroner, RIDME overvinder alle disse ulemper.
Denne teknik giver forskerne mulighed for at arbejde med molekyler under indfødte forhold, som professor Malte Drescher og hovedforfatter Dr. Mykhailo Azarkh, begge fra universitetet i Konstanz, understrege:"Vi startede med at analysere konformationen af et protein inde i cellen. Med mindre følsomme teknikker, vi er tvunget til at indsætte og mærke en masse protein for at kunne observere det, hvilket slet ikke er det, der sker i naturen. Ideelt set, vi ønsker at arbejde med koncentrationer, der er fysiologisk relevante. Da RIDME er meget mere følsom end Hjorte, det tillader os at gøre netop det. Vi er nu i stand til at løse spørgsmål, som vi ellers ikke ville kunne løse ”.
Ydeevnen for RIDME i celle blev vurderet ved Q-bånd ved hjælp af stive molekylære linealer mærket med Gd (III) -PyMTA og mikrosamlet i Xenopus laevis (afrikansk klofrø) oocytter. Med andre ord, forskerne brugte et modelsystem, hvor den præcise afstand mellem spin -etiketterne allerede var kendt, giver dem mulighed for at verificere RIDME -målingerne. Det resulterende papir med titlen "Gd (III) –Gd (III) Relaxation-induced Dipolar Modulation Enhancement for In-Cell Electron Paramagnetic Resonance Distance Determination" blev offentliggjort online i Journal of Physical Chemistry Letters den 13.03.2019.
RIDME afstandsbestemmelse i celle blev udviklet og testet som en del af det igangværende ERC-finansierede projekt "SPICE-Spektroskopi i celler", hvortil Malte Drescher, Heisenberg professor for spektroskopi af komplekse systemer ved universitetet i Konstanz, og hans forskerhold blev anerkendt med et ERC Consolidator Grant til en værdi af cirka to millioner euro i 2017. Deres mål er at udvikle nye metoder til spektroskopi, der giver dem mulighed for at udforske større og mere komplekse biologiske strukturer på cellens molekylære niveau.
Det næste trin i denne forskningslinje vil være at identificere andre egnede spin -mærker og at udvikle RIDME til anvendelse i molekyler, hvor afstanden mellem spin -etiketterne er ukendt. Et særligt fokus vil være på molekyler forbundet med neurodegenerative sygdomme, såsom Alzheimers og Parkinsons.
Sidste artikelForskere skaber brintbrændstof fra havvand
Næste artikelNy metode til test af kokain fundet
 Varme artikler
Varme artikler
-
 En forenklet metode til at kategorisere olivenolieLourdes Arce og Natividad Jurado, efterforskere fra Universidad de Cordoba. Kredit:University of Cordoba Klassificering af olivenolie er i øjeblikket meget dyr og langsom. For at kategorisere olie
En forenklet metode til at kategorisere olivenolieLourdes Arce og Natividad Jurado, efterforskere fra Universidad de Cordoba. Kredit:University of Cordoba Klassificering af olivenolie er i øjeblikket meget dyr og langsom. For at kategorisere olie -
 Naturlignende belægning gør batterier mere holdbare og effektiveCarbon nanorør belagt med en belægning ved anvendelse af kuldioxid i molekylær lagdeponering. Kredit:Aalto University Når batterier oplades og bruges, et komplekst SEI (fast elektrolytinterfase) l
Naturlignende belægning gør batterier mere holdbare og effektiveCarbon nanorør belagt med en belægning ved anvendelse af kuldioxid i molekylær lagdeponering. Kredit:Aalto University Når batterier oplades og bruges, et komplekst SEI (fast elektrolytinterfase) l -
 Kemikalierne i salt, peber og sukkerSukker, salt og peber er blandt de mest anvendte køkkeningredienser. Sukker og salt er kemiske forbindelser, og peber er et naturligt forekommende krydderi. Sort peber eller Piper nigrum er den mes
Kemikalierne i salt, peber og sukkerSukker, salt og peber er blandt de mest anvendte køkkeningredienser. Sukker og salt er kemiske forbindelser, og peber er et naturligt forekommende krydderi. Sort peber eller Piper nigrum er den mes -
 Hvad opløser olie?Petroleum indeholder forskellige former for olie, såsom fyringsolie og smøremidler, og mange andre olier kommer fra vegetabilske stoffer, såsom olivenolie, palmeolie og rapsolie. Ingen af disse o
Hvad opløser olie?Petroleum indeholder forskellige former for olie, såsom fyringsolie og smøremidler, og mange andre olier kommer fra vegetabilske stoffer, såsom olivenolie, palmeolie og rapsolie. Ingen af disse o
- Rester af opdagelsesrejsende, der først rundede Australien, fundet i Storbritannien
- Hot nanostrukturer køler hurtigere ned, når de er fysisk tæt på hinanden
- FN's biodiversitetsmøde afsluttes, indberetning på mandag
- Udforskning af nye spintronics-enhedsfunktioner i grafen-heterostrukturer
- Friske DNA-tests autentificerer knogler fra Ruslands zar, familie
- Radioteleskop optager en sjælden fejl i et pulsars regelmæssige pulserende beat


