Supercomputere simulerer nye veje til potentiel behandling af RNA-virus
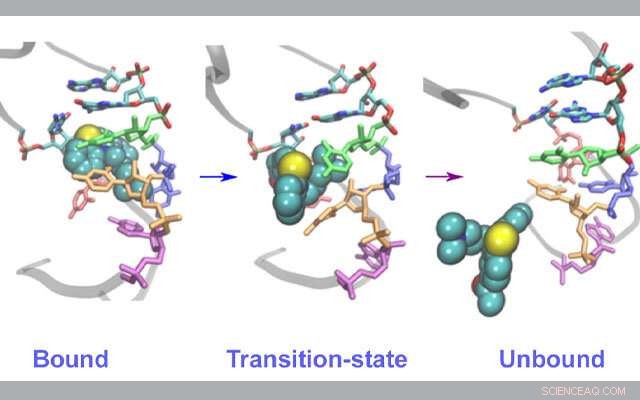
Strukturelle ændringer i RNA under lægemiddelbinding/afbinding. Kredit:Lev Levintov, University of New Hampshire
University of New Hampshire (UNH) forskere brugte for nylig Comet ved San Diego Supercomputer Center ved UC San Diego og Stampede2 ved Texas Advanced Computing Center til at identificere nye inhibitorbindings-/afbindingsveje i en RNA-baseret virus. Resultaterne kan være gavnlige til at forstå, hvordan disse inhibitorer reagerer og potentielt hjælpe med at udvikle en ny generation af lægemidler til at målrette virus med høje dødsrater, såsom HIV-1, Zika, Ebola, og SARS-CoV2, virus, der forårsager COVID-19.
"Da vi først startede denne forskning, vi havde aldrig forventet, at vi ville være midt i en pandemi forårsaget af en RNA-virus, " sagde Harish Vashisth, lektor i kemiteknik ved UNH. "Når disse typer vira dukker op, vores resultater vil forhåbentlig give en forbedret forståelse af, hvordan virale RNA'er interagerer med inhibitorer og bruges til at designe bedre behandlinger."
Svarende til hvordan mennesker koder deres genom ved hjælp af DNA, mange vira har en genetisk sammensætning af RNA-molekyler. Disse RNA-baserede genomer indeholder potentielle steder, hvor inhibitorer kan binde og deaktivere virussen. En del af udfordringen i lægemiddeludvikling er, at variationer eller mutationer i det virale genom, der kan forhindre inhibitorerne i at binde sig.
I deres undersøgelse, for nylig offentliggjort i Journal of Physical Chemistry Letters , Vashisth og hans team skabte simuleringer af molekylær dynamik ved hjælp af Comet- og Stampede2-supercomputere til at se specifikt på et RNA-fragment fra HIV-1-virussen og dets interaktion med acetylpromazin, et lille molekyle, der vides at forstyrre virusreplikationsprocessen.
Forskerne fokuserede på de strukturelle elementer fra HIV-1 RNA-genomet, fordi de betragtes som en god model til at studere de samme processer på tværs af en bred vifte af RNA-vira. Disse simuleringer gjorde dem i stand til at opdage veje for inhibitorens afbinding fra det virale RNA i flere sjældne hændelser - som ofte er svære at observere eksperimentelt - som uventet viste en koordineret bevægelse i mange dele af bindingslommen, som er byggestenene i RNA.
Takket være National Science Foundations (NSF) Extreme Science and Engineering Environment (XSEDE) tildelinger på Comet and Stampede2, forskerne var i stand til at køre hundredvis af simuleringer på samme tid for at observere, hvad der kaldes sjældne base-flipping-hændelser involveret i inhibitorens binding/afbindingsproces, der gav de nye detaljer om den underliggende mekanisme i denne proces.
"Vores håb er, at dette tilføjer nye muligheder til et felt, der traditionelt fokuserer på statiske biomolekylære strukturer og fører til ny medicin, " sagde Vashisth.
 Varme artikler
Varme artikler
-
 Holografisk stråleformning for at give et boost til metallisk 3-D-printAfdelingens set-up for additiv fremstilling (AM). Kredit:Peter Christopher Cambridge ingeniører har påbegyndt et treårigt forskningsprogram for at hjælpe med at fremskynde fremstillingen af meta
Holografisk stråleformning for at give et boost til metallisk 3-D-printAfdelingens set-up for additiv fremstilling (AM). Kredit:Peter Christopher Cambridge ingeniører har påbegyndt et treårigt forskningsprogram for at hjælpe med at fremskynde fremstillingen af meta -
 Fremtidige muligheder for lagring af kuldioxid:Syntese af uorganiske heteroalkenerStrukturer af gallaphosphen (1) og komplekset af CO 2 (2) og acetophenon (3). (c) UDE/S.Schulz Gruppe 13/15 heteroalkener RMER med M-E dobbeltbindinger (M =B-Tl; E =N-Bi) tilbyder lovende potent
Fremtidige muligheder for lagring af kuldioxid:Syntese af uorganiske heteroalkenerStrukturer af gallaphosphen (1) og komplekset af CO 2 (2) og acetophenon (3). (c) UDE/S.Schulz Gruppe 13/15 heteroalkener RMER med M-E dobbeltbindinger (M =B-Tl; E =N-Bi) tilbyder lovende potent -
 Forskere finder intermolekylære kræfter stabiliserer klynger, fremme aerosolproduktionKredit:CC0 Public Domain En gåde, der har forvirret videnskabsmænd, er, hvordan nye partikler dannes i atmosfæren. De ved, hvordan aerosoler kan vokse til størrelser, der er store nok til at udså
Forskere finder intermolekylære kræfter stabiliserer klynger, fremme aerosolproduktionKredit:CC0 Public Domain En gåde, der har forvirret videnskabsmænd, er, hvordan nye partikler dannes i atmosfæren. De ved, hvordan aerosoler kan vokse til størrelser, der er store nok til at udså -
 Opdagelse af, hvordan mennesker oplever lugten af død, kan redde livEdvard Munchs Lugten af død. Og himlen så på det fantastiske kadaver Blomstrer som en blomst. Så forfærdelig var stanken, at du troede Du ville besvime på græsset. Slagfluerne summede rundt
Opdagelse af, hvordan mennesker oplever lugten af død, kan redde livEdvard Munchs Lugten af død. Og himlen så på det fantastiske kadaver Blomstrer som en blomst. Så forfærdelig var stanken, at du troede Du ville besvime på græsset. Slagfluerne summede rundt
- Genmanipuleret gær opsuger tungmetalforurening
- Kan du gemme solenergi i rust og vand?
- Forhistorisk haj skjulte sine største tænder
- Ny forskning afslører hemmeligheden bag Jupiters nysgerrige nordlysaktivitet
- Forskere arbejder på vandskelmoment i vandrensning
- Klassificerede knob:Forskere skaber optiske indrammede knob for at kode information


