En ny bioimaging metode til at fremskynde og forenkle kemikalieidentifikation i væv
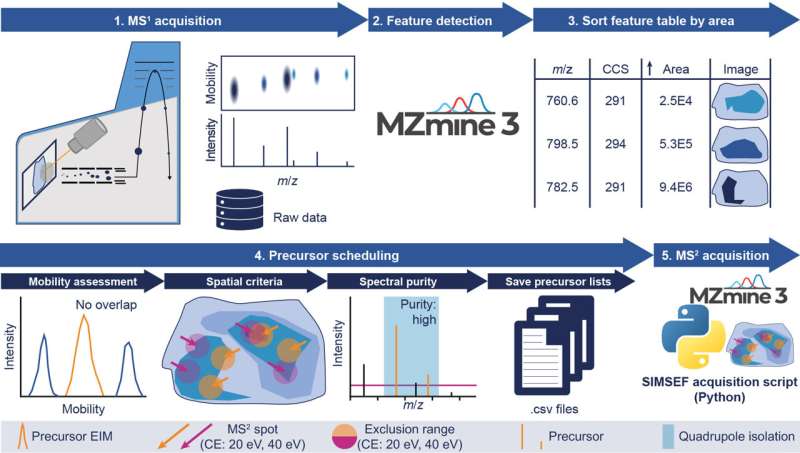
Forskere tilknyttet det internationale softwareprojekt MZmine, ledet af Dr. Robin Schmid og Dr. Tomáš Pluskal fra Institut for Organisk Kemi og Biokemi ved Det Tjekkiske Videnskabsakademi, er kommet med et nyt stykke software, der markant fremskynder og forenkler identifikation af kemikalier i væv.
Det gør det muligt for forskere at genkende kemikalier med sikkerhed og visualisere deres fordeling i organer. Sammenlignet med etablerede arbejdsgange kræver den nye pipeline mindre arbejde i laboratoriet for at få lignende indsigt i den kemi, der sker inden for for eksempel tumorer og inflammatoriske læsioner. Artiklen, der præsenterer denne software for det globale videnskabelige samfund, er netop blevet offentliggjort i tidsskriftet Nature Communications .
Hos IOCB Prag er Dr. Robin Schmid mest involveret i udviklingen af softwaren SIMSEF (spatial ion mobility-scheduled exhaustive fragmentation) sammen med sine kolleger fra gruppen af Prof. Dr. Karst ved universitetet i Münster, Tyskland, hvor han tidligere var baseret.
Arbejdet er udført i samarbejde med udviklerne af timsTOF fleX state-of-the-art massespektrometer fra Bruker Daltonics, som muliggør belysning på højt niveau af sammensætningen af molekyler ved at måle ioners mobilitet.
"Indtil nu kunne forskerne fortælle med et massespektrometer, at det var formlen for det molekyle, de så på, men da de så på databasen og forsøgte at identificere dette stof, var det meget svært. Årsagen er, at biologiske prøver kan indeholde adskillige forskellige lipider og kombinationer af molekyler, der ofte er biologisk meget forskellige."
"Nu er det, takket være den nye algoritme, muligt at kigge ind i molekylet, lære, hvad det består af og endda sammenligne billeder med hinanden," forklarer Robin Schmid. Dette kan give vigtig information, for eksempel at en specifik del af hjernen indeholder en anden type lipid end en anden del af dette essentielle organ, og at netop dette lipid er fraværende i andet væv.
SIMSEF-algoritmen er udviklet i samarbejde med medicinske eksperter fra tyske og schweiziske universiteter. Det er afgørende for læger, at påvisningen af kliniske biomarkører til diagnostiske formål også fremskyndes betydeligt. At lære meget hurtigt, for eksempel, om de har at gøre med et lipid eller en metabolit, der kun findes i kræftvæv, spiller en afgørende rolle i beslutningen om yderligere behandling. Det er overflødigt at sige, at dette har store konsekvenser for resultatet.
Den nye algoritme er en del af open source-softwarepakken MZmine, som har hjulpet eksperter verden over med at analysere massespektrometridata siden 2005. Steffen Heuckeroth fra MZmine-teamet og Institute of Inorganic and Analytical Chemistry ved University of Münster har været hovedudvikler af SIMSEF-algoritmen og er den første forfatter til avisen, der nu er udgivet i Nature Communications .
Han tilføjer:"Den pågældende artikel indeholder ikke nogen specifik opdagelse inden for biologi. Men takket være vores nye metode kan mange forskere forbedre og accelerere deres arbejde, hvilket åbner plads til nye opdagelser."
"Jeg synes altid, det er fantastisk, når vi gør det muligt for andre mennesker at forbedre arbejdsvilkårene for deres forskning og også at kommunikere det til andre forskere. Takket være feedbacken fra det videnskabelige samfund, der bruger MZmine, skubber programmet i sig selv direkte deres forskning fremad," tilføjer Robin Schmid fra IOCB Prag.
Direktøren for IOCB Prag prof. Jan Konvalinka påpeger:"Alle taler om maskinlæring og kunstig intelligens i disse dage. Vores kolleger fra Dr. Pluskals team bruger det til at løse meget specifikke problemer inden for forskning såvel som i klinisk praksis."
Den tredje generation af MZmine, rapporteret i foråret 2023 i tidsskriftet Nature Biotechnology , kan behandle tusindvis af prøver i timen. Algoritmerne, der er nyligt udviklet af dette internationale team, forbedrer således de eksisterende muligheder for at sammenkæde forskellige typer data, herunder kombination af data fra analytiske og billeddannelsesmetoder, hvilket indtil nu ikke har været muligt med nogen akademisk eller kommerciel software.
Flere oplysninger: Steffen Heuckeroth et al., On-tissue datasæt-afhængig MALDI-TIMS-MS2 bioimaging, Nature Communications (2023). DOI:10.1038/s41467-023-43298-9
Journaloplysninger: Nature Communications , Naturbioteknologi
Leveret af Institut for Organisk Kemi og Biokemi ved Det Tjekkiske Videnskabsakademi (IOCB Prag)
 Varme artikler
Varme artikler
-
 Teamet bruger varmt vand til at danne fotokatalysatorSyntese af n-alkyl-PBI i nærvær af TEOS (n =1, 5, 10 og 100 ækv.). R =n-propylamin (C3), n-pentylamin (C5), n-octylamin (C8), n-tetradecylamin (C14). Kredit:Hipassia M. Moura et al., Journal of Materi
Teamet bruger varmt vand til at danne fotokatalysatorSyntese af n-alkyl-PBI i nærvær af TEOS (n =1, 5, 10 og 100 ækv.). R =n-propylamin (C3), n-pentylamin (C5), n-octylamin (C8), n-tetradecylamin (C14). Kredit:Hipassia M. Moura et al., Journal of Materi -
 Bakterielt enzym kan blive et nyt mål for antibiotikaMIT-forskere har opdaget strukturen af et usædvanligt enzym, som nogle mikrober bruger til at nedbryde en komponent af kollagen i den menneskelige tarm. Kredit:Christine Daniloff, MIT MIT og Har
Bakterielt enzym kan blive et nyt mål for antibiotikaMIT-forskere har opdaget strukturen af et usædvanligt enzym, som nogle mikrober bruger til at nedbryde en komponent af kollagen i den menneskelige tarm. Kredit:Christine Daniloff, MIT MIT og Har -
 Plast, brændstoffer og kemiske råstoffer fra CO2? De arbejder på detForskere ved Stanford og SLAC arbejder på måder at omdanne kuldioxidaffald (CO2) til kemiske råvarer og brændstoffer, gør en potent drivhusgas til værdifulde produkter. Processen kaldes elektrokemisk
Plast, brændstoffer og kemiske råstoffer fra CO2? De arbejder på detForskere ved Stanford og SLAC arbejder på måder at omdanne kuldioxidaffald (CO2) til kemiske råvarer og brændstoffer, gør en potent drivhusgas til værdifulde produkter. Processen kaldes elektrokemisk -
 Video:Hvad er evige kemikalier, og holder de evigt?For altid er kemikalier kendt for at være vand-, varme- og oliebestandig, hvilket gør dem nyttige i alt fra regnjakker til brandslukningsskum. Men den kemi, der gør dem så nyttige, får dem også til at
Video:Hvad er evige kemikalier, og holder de evigt?For altid er kemikalier kendt for at være vand-, varme- og oliebestandig, hvilket gør dem nyttige i alt fra regnjakker til brandslukningsskum. Men den kemi, der gør dem så nyttige, får dem også til at
- Ny undersøgelse forklarer, hvad der driver Great Sitkin Volcanos langlivede udbrud
- Eksperiment sender konstruerede anlæg til ISS
- NASA satellitbillede Tropisk depression Hector forlænges, svækkelse
- Højnøjagtig batteriovervågning af elbiler med diamantkvantesensorer til forlængelse af køreafst…
- At bygge bro over kløften:Dataloger udvikler model til at forbedre vanddata fra satellitter
- Kemikere demonstrerer legeringer i nanoskala så lyse, at de kan have potentielle medicinske anvende…


