Storstilet undersøgelse viser ny, rigelige nitrogenfikserende mikrober i overfladehav
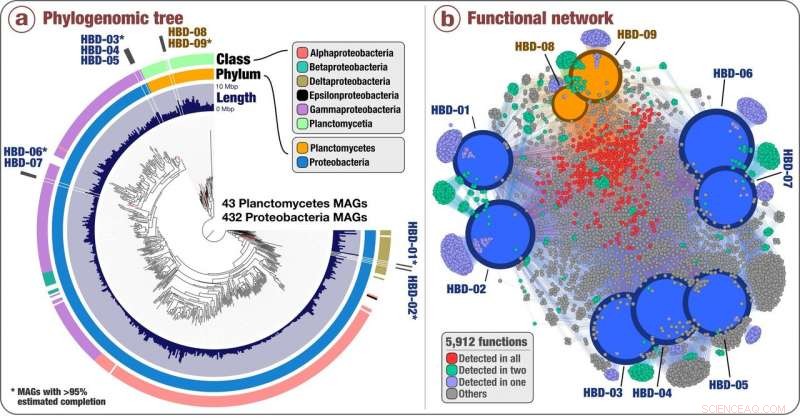
Sammenhæng mellem fylogeni og funktion af de nyopdagede nitrogenfiksere i overfladehaves metagenomer. (a) Fylogenomisk analyse af 432 Proteobacteria-genomer og 43 Planctomycetes-genomer, som Delmont og kolleger karakteriserede (herunder de ni ikke-fotosyntetiske N-fiksere) ved hjælp af en samling af 37 fylogenetiske markørgenfamilier. Lag, der omgiver det fylogenomiske træ, angiver genomstørrelse og taksonomi af hvert genom på phylum- og klasseniveau. (b) Funktionelt netværk af de ni ikke-fotosyntetiske N-fiksere baseret på i alt 5, 912 identificerede genfunktioner. Størrelse og farve af genomiske knuder repræsenterer antallet af detekterede funktioner og genomisk taksonomi, henholdsvis. Farven på funktionelle noder indikerer deres forekomst i de forskellige genomer. Kredit:Delmont et al (2018) Naturens mikrobiologi , doi:10.1038/s41564-018-0176-9
Flyt dig, cyanobakterier! En storstilet undersøgelse af jordens overfladehav viser, at de mikrober, der er ansvarlige for at fiksere nitrogen der - tidligere antaget at være næsten udelukkende fotosyntetiske cyanobakterier - omfatter en rigelig og vidt udbredt række af ikke-fotosyntetiske bakteriepopulationer.
Den internationale undersøgelse, udgivet i denne uge i Naturens mikrobiologi , blev ledet af A. Murat Eren (Meren) fra University of Chicago og Marine Biological Laboratory (MBL), Woods Hole, og Tom O. Delmont fra University of Chicago.
Nitrogenfiksering er en kritisk økologisk proces, hvor atmosfærisk nitrogen omdannes til ammoniak, at gøre nitrogen "biotilgængeligt" for levende organismer til brug som en grundlæggende byggesten i DNA, RNA og proteiner.
"Mikrober, der kan fikse nitrogen eller kulstof, er i centrum for mikrobielle samfunds økologi i mange miljøer, inklusive overfladehavet, " siger Delmont. "Før vores undersøgelse, man troede, at de marine mikrober, der var ansvarlige for kulstoffiksering, også i høj grad var ansvarlige for at fiksere nitrogen. Det viser sig ikke at være så enkelt."
"Mikrobers evne til at binde nitrogen er afgørende for alt liv, " siger David Mark Welch, MBL forskningsdirektør. "Denne undersøgelse udvider vores forståelse af den biologiske mangfoldighed af nitrogenfiksering ved at give det første genomiske bevis på, at ikke-fotosyntetiske bakterier på havoverfladen kan udføre disse reaktioner."
Ved hjælp af anvi'o, en state-of-the-art, open source bioinformatikplatform til at analysere metagenomer (puljen af DNA-sekvenser, der repræsenterer alle de mikrobielle organismer, der findes i et miljø), holdet afslørede indsigt i hidtil ukendte marine mikrober med nitrogenfikseringsevner tilknyttet Proteobacteria såvel som Planctomycetes, en udbredt bakteriel phylum, der aldrig før har været forbundet med nitrogenfiksering.
Disse nyligt beskrevne mikrobielle populationer forekommer bredt og er særligt rigelige i Stillehavet, hvor de i gennemsnit skønner 700, 000 celler per liter havvand og op til 3 millioner celler per liter - størrelsesordener mere end tidligere skøn for ikke-cyanobakterielle nitrogenfiksere i det åbne hav.
Ved at bruge data genereret fra Tara Oceans ekspeditionen fra 2009 til 2013, Delmont og kolleger rekonstruerede omkring 1, 000 mikrobielle genomer fra mere end 30 milliarder korte metagenomiske sekvenser. Af de 1, 000 genomer, ni indeholdt de seks gener, der er nødvendige for nitrogenfiksering, og alligevel manglede de gener, der var nødvendige for fotosyntese. Dette er den første genomiske database over ikke-fotosyntetiske mikroorganismer, der bor i det åbne hav og er i stand til at fiksere nitrogen.

A. Murat Eren (Meren) og Tom Delmont arbejder med Tara Oceans data på University of Chicago i 2017. Kredit:Fran Jackson
Fordi holdet rekonstruerede og brugte næsten komplette genomer til deres undersøgelse (i stedet for at bruge et enkelt markørgen til nitrogenfiksering), de kunne løse de taksonomiske tilknytninger af disse nitrogenfikserende populationer. De kunne også undersøge deres overflod og udbredelsesmønstre i oceanerne og havene, hvorfra prøverne kom (Atlanterhavet, Stillehavet, Indiske og sydlige oceaner og Middelhavet og Rødehavet).
"Vi kan nu bruge disse populationsgenomer til at guide laboratoriedyrkningen af nitrogenfikserende Planctomycetes og Proteobacteria fra det åbne hav, " siger Delmont. "Dette vil hjælpe os med at forstå de forhold, hvorunder de fikserer nitrogen, kompleksiteten af deres funktionelle livsstil, og andre aspekter af deres økologi, som vi ikke kan forstå ved blot at se på deres genomer, gener og udledte funktioner."
Meren og Delmont begyndte denne forskning på Marine Biological Laboratory i 2015 med støtte fra en University of Chicago Lillie Innovation Award. Meren og hans gruppe fortsætter med at udvikle anvi'o, open source-softwareplatformen, der bruges i denne og andre undersøgelser, der undersøger mikrobers økologi og udvikling gennem komplekse miljøsekvenseringsdata.
"Miljømetagenomer giver os uforfalsket adgang til kompleksiteten af naturligt forekommende mikrobielle populationer, " siger Meren. "Mens vores evne til at forstå dem er prisgivet vores molekylære teknologier og beregningsværktøjer, det er forfriskende at se begge udvikle sig hurtigt, og der er stadig så meget at opdage. Vi ser frem til at se fremskridt på bench-side, der underbygger denne indledende indsigt i miljøpopulationer, der sandsynligvis bidrager til en af de mest essentielle biokemiske processer, der får vores planet til at tikke."
"Denne undersøgelse er endnu et eksempel på, hvordan opløsning af genomer direkte fra DNA'et fra hele mikrobielle samfund transformerer vores forståelse af mikrobiel mangfoldighed, " siger medforfatter Christopher Quince fra University of Warwick, Det Forenede Kongerige.
 Varme artikler
Varme artikler
-
 En ny forståelse af tilslutning og bio-arvKredit:Paolo Palladino EU-finansierede forskere dykkede ned i, hvordan naturen af ting er formet af deres tilslutningsmuligheder, især med hensyn til biologiske materialer. Forskningen resultere
En ny forståelse af tilslutning og bio-arvKredit:Paolo Palladino EU-finansierede forskere dykkede ned i, hvordan naturen af ting er formet af deres tilslutningsmuligheder, især med hensyn til biologiske materialer. Forskningen resultere -
 Vi har mere end nok kalorier, men hvad med andre næringsstoffer?Kredit:CC0 Public Domain De Forenede Nationers andet mål for bæredygtig udvikling sigter mod afslutningen af underernæring i alle former inden 2030. Men ny forskning viser, at for at nå dette må
Vi har mere end nok kalorier, men hvad med andre næringsstoffer?Kredit:CC0 Public Domain De Forenede Nationers andet mål for bæredygtig udvikling sigter mod afslutningen af underernæring i alle former inden 2030. Men ny forskning viser, at for at nå dette må -
 Brandmænd kæmper for at beskytte kæmpe sequoias i Californiens brandeGiant sequoia træer er de største træer i verden, men er stadig sårbare over for de skrækkelige naturbrande, der raser gennem Californien. Hundredvis af brandmænd kæmpede for at beskytte flere lun
Brandmænd kæmper for at beskytte kæmpe sequoias i Californiens brandeGiant sequoia træer er de største træer i verden, men er stadig sårbare over for de skrækkelige naturbrande, der raser gennem Californien. Hundredvis af brandmænd kæmpede for at beskytte flere lun -
 Øger indsatsen på plastikpillerSpildte piller kan ende på strandene. Kredit:Alasdair Neilson Plast er et utrolig nyttigt materiale, som meget af vores daglige liv afhænger af. COVID-19-pandemien sætter vigtigheden af plastik
Øger indsatsen på plastikpillerSpildte piller kan ende på strandene. Kredit:Alasdair Neilson Plast er et utrolig nyttigt materiale, som meget af vores daglige liv afhænger af. COVID-19-pandemien sætter vigtigheden af plastik
- Billede:Nordøst Kenya
- Menneskelig hjerne supercomputer med 1 million processorer tændt for første gang
- Fænomen beskrevet af Alan Turing forklarer anti-reflekterende insektøjne
- Hvordan man fortæller, om en påfugl er mand eller kvinde
- EU's øverste regulator for Big Tech ser ud til at bruge stærkere handling
- Alt-afvisende belægning kunne børnesikre telefoner, hjem


