Selvsamlende nanocages er de største selvstændige 3-D DNA-strukturer endnu

De fem burformede DNA-polyedre her har stivere, der stabiliserer deres ben, og denne innovation gjorde det muligt for et Wyss Institute-team at bygge langt de største og mest robuste DNA-bure til dato. Den største, et sekskantet prisme (til højre), er en tiendedel af størrelsen af en gennemsnitlig bakterie. Kredit:Yonggang Ke/Harvard's Wyss Institute
Flyt dig, nanoteknologer, og give plads til de største af de små. Forskere ved Harvard's Wyss Institute har bygget et sæt selvsamlende DNA-bure, der er en tiendedel så brede som en bakterie. Strukturerne er nogle af de største og mest komplekse strukturer, der nogensinde er konstrueret udelukkende ud fra DNA, beretter de om dagens netudgave af Videnskab .
I øvrigt, forskerne visualiserede dem ved hjælp af en DNA-baseret superopløsningsmikroskopimetode - og opnåede de første skarpe 3D-optiske billeder af intakte syntetiske DNA-nanostrukturer i opløsning.
I fremtiden, Forskere kunne potentielt belægge DNA-burene for at omslutte deres indhold, emballering af lægemidler til levering til væv. Og, som et rummeligt skab, buret kunne modificeres med kemiske kroge, der kunne bruges til at hænge andre komponenter såsom proteiner eller guld nanopartikler. Dette kan hjælpe videnskabsmænd med at bygge en række forskellige teknologier, herunder små kraftværker, små fabrikker, der producerer specialkemikalier, eller højfølsomme fotoniske sensorer, der diagnosticerer sygdom ved at detektere molekyler produceret af unormalt væv.
"Jeg ser spændende muligheder for denne teknologi, " sagde Peng Yin, Ph.D., et kernefakultetsmedlem ved Wyss Institute og assisterende professor i systembiologi ved Harvard Medical School, og seniorforfatter af papiret.
Bygning med DNA
DNA er bedst kendt som en keeper af genetisk information. Men forskere inden for det nye område af DNA-nanoteknologi udforsker måder at bruge det til at bygge små strukturer til en række forskellige anvendelser. Disse strukturer er programmerbare, ved at videnskabsmænd kan specificere rækkefølgen af bogstaver, eller baser, i DNA'et, og disse sekvenser bestemmer så den struktur, den skaber.
Hidtil har de fleste forskere på området brugt en metode kaldet DNA origami, hvor korte DNA-strenge hæfter to eller tre separate segmenter af en meget længere streng sammen, hvilket får den tråd til at folde sig til en præcis form. DNA-origami blev delvist udviklet af Wyss Institute Core Faculty-medlem William Shih, Ph.D., der også er lektor i Institut for Biologisk Kemi og Molekylær Farmakologi ved Harvard Medical School og Institut for Kræftbiologi ved Dana-Farber Cancer Institute.
Yins team har bygget forskellige typer DNA-strukturer, inklusive et modulært sæt dele kaldet enkeltstrengede DNA-fliser eller DNA-klodser. Ligesom LEGO® klodser, disse dele kan tilføjes eller fjernes uafhængigt. I modsætning til LEGO® klodser, de samler sig spontant selv.
Men for nogle applikationer, videnskabsmænd skal muligvis bygge meget større DNA-strukturer, end nogen har bygget hidtil. Så, at tilføje til deres værktøjskasse, Yins team søgte meget større byggeklodser til at matche.
Tekniske udfordringer
Yin og hans kolleger brugte først DNA-origami til at skabe ekstra store byggeklodser i form af en fotografs stativ. Planen var at konstruere disse stativben til at fastgøre ende til ende for at danne polyedre - genstande med mange flade flader, der i sig selv er trekanter, rektangler, eller andre polygoner.
Men da Yin og avisens tre hovedforfattere, Ryosuke Iinuma, en tidligere Wyss Institute Visiting Fellow, Yonggang Ke, Ph.D., en tidligere Wyss Postdoc-stipendiat, som nu er assisterende professor i biomedicinsk teknik ved Georgia Institute of Technology og Emory University, og Ralf Jungman, Ph.D, en Wyss postdoc-stipendiat, byggede større stativer og prøvede at samle dem til polyedre, de store stativers ben slingrede og vaklede, hvilket forhindrede dem i overhovedet at lave polyeder.
Forskerne kom uden om det problem ved at indbygge en vandret stiver for at stabilisere hvert par ben, ligesom en møbelsnedker ville bruge et stykke træ til at bygge bro over benene på en vaklende stol.
For at lime stativets ben sammen ende-til-ende, de udnyttede det faktum, at matchende DNA-strenge parrer sig og klæber til hinanden. De efterlod et DNA-mærke hængende på et stativben, og et matchende mærke på benet på et andet stativ, som de ville have det til at parre med.
Holdet programmerede DNA til at folde til robuste stativer, der er 60 gange større end tidligere DNA-stativlignende byggesten og 400 gange større end DNA-klodser. Disse stativer blev derefter selvsamlet til en specifik type tredimensionel polyeder - alt sammen i et enkelt reagensglas.
Ved at justere længden af stiveren, de byggede stativer, der spændte fra opretstående til sprælske ben. Flere opretstående stativer dannede polyedre med færre ansigter og skarpere vinkler, såsom et tetraeder, som har fire trekantede flader. Flere splaybenede stativer dannede polyedre med flere ansigter, såsom et sekskantet prisme, som er formet som et ostehjul og har otte flader, inklusive dens top og bund.
I alt, de skabte fem polyedre:et tetraeder, et trekantet prisme, en terning, a pentagonal prism, and a hexagonal prism.
Ultrasharp snapshots
After building the cages, the scientists visualized them using a DNA-based microscopy method Jungmann had helped developed called DNA-PAINT. In DNA-PAINT, short strands of modified DNA cause points on a structure to blink, and data from the blinking images reveal structures too small to be seen with a conventional light microscope. DNA-PAINT produced ultrasharp snapshots of the researchers' DNA cages – the first 3D snapshots ever of single DNA structures in their native, watery environment.
"Bioengineers interested in advancing the field of nanotechnology need to devise manufacturing methods that build sturdy components in a highly robust manner, and develop self-assembly methods that enable formation of nanoscale devices with defined structures and functions, " sagde Wyss Institutes stiftende direktør Don Ingber, M.D., Ph.D. "Peng's DNA cages and his methods for visualizing the process in solution represent major advances along this path."
 Varme artikler
Varme artikler
-
 Robotter i cellestørrelse kan mærke deres omgivelserOptiske billeder viser kredsløb lavet af forskerholdet, før de bliver knyttet til partikler blot et par hundrede nanometer på tværs. Kredit:Massachusetts Institute of Technology Forskere ved MIT h
Robotter i cellestørrelse kan mærke deres omgivelserOptiske billeder viser kredsløb lavet af forskerholdet, før de bliver knyttet til partikler blot et par hundrede nanometer på tværs. Kredit:Massachusetts Institute of Technology Forskere ved MIT h -
 En foldekeramikLagdelt keramisk papir:Scanningelektronmikrofotografier viser de stablede lag af vanadiumpentoxid og vand i kompositten. Materialet er så elastisk og sejt, at det kan bøjes (højre billede). Kredit:Adv
En foldekeramikLagdelt keramisk papir:Scanningelektronmikrofotografier viser de stablede lag af vanadiumpentoxid og vand i kompositten. Materialet er så elastisk og sejt, at det kan bøjes (højre billede). Kredit:Adv -
 At spille pool med atomerLehighs kraftfulde højopløselige røntgenfotoelektronspektrometre (HR-XPS), den eneste af slagsen i USA, kan bestemme den kemiske natur af atomer i et materiales overfladeområde og er et perfekt supple
At spille pool med atomerLehighs kraftfulde højopløselige røntgenfotoelektronspektrometre (HR-XPS), den eneste af slagsen i USA, kan bestemme den kemiske natur af atomer i et materiales overfladeområde og er et perfekt supple -
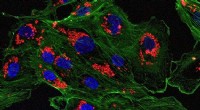 Nanopartikler narrer kroppen til at acceptere organtransplantationerEt mikroskopisk billede af endotelceller behandlet med lægemiddelfyldte nanopartikler. Kredit:Saltzman Lab Ved hjælp af nanopartikler, Yale-forskere har udviklet et system til levering af lægemidl
Nanopartikler narrer kroppen til at acceptere organtransplantationerEt mikroskopisk billede af endotelceller behandlet med lægemiddelfyldte nanopartikler. Kredit:Saltzman Lab Ved hjælp af nanopartikler, Yale-forskere har udviklet et system til levering af lægemidl
- Digital teknologi kan starte en ny videnskabelig revolution inden for samfundsforskning
- Indsigt i den enorme indflydelse, sæsoner har i landbrugsøkonomier
- En ny procedure for at opnå en billig, ultrahårdt materiale, der er modstandsdygtigt over for radi…
- NASA ser Debby overgå til en tropisk storm
- Forskere løser et 60-årigt puslespil om et superhårdt materiale
- Enkel detektion af magnetiske skyrmioner


