Ny billeddannelsesmetode afslører detaljer i nanoskala om DNA
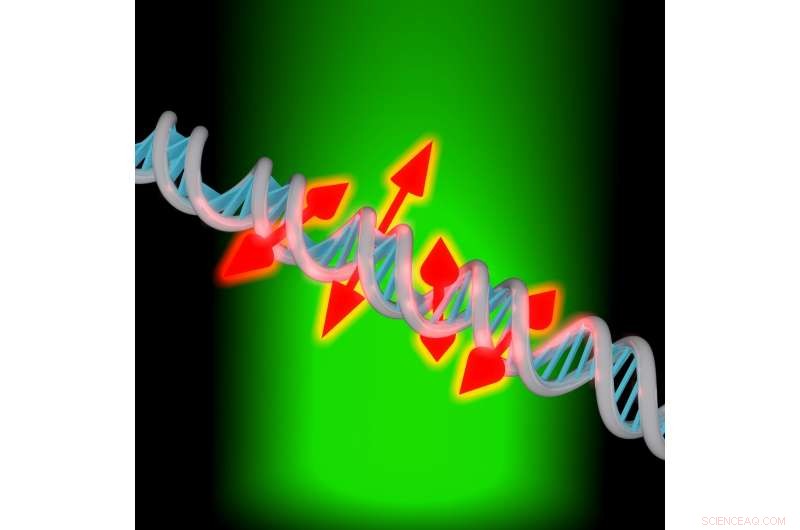
En ny billeddannelsesteknik giver forskere mulighed for at afbilde både positionen og orienteringen af enkelte fluorescerende molekyler knyttet til DNA. Kredit:Maurice Y. Lee, Stanford University
Forskere har udviklet en ny forbedret DNA-billeddannelsesteknik, der kan undersøge strukturen af individuelle DNA-strenge på nanoskala. Da DNA er roden til mange sygdomsprocesser, Teknikken kan hjælpe videnskabsmænd med at få vigtig indsigt i, hvad der går galt, når DNA bliver beskadiget, eller når andre cellulære processer påvirker genekspression.
Den nye billeddannelsesmetode bygger på en teknik kaldet enkeltmolekylemikroskopi ved at tilføje information om orienteringen og bevægelsen af fluorescerende farvestoffer knyttet til DNA-strengen.
W. E. Mørner, Stanford University, USA, er grundlæggeren af enkelt-molekyle spektroskopi, en banebrydende metode fra 1989, der gjorde det muligt for forskere at visualisere enkelte molekyler med optisk mikroskopi for første gang. Af 2014 Nobelpristagere for optisk mikroskopi ud over diffraktionsgrænsen (Moerner, Hell &Betzig), Moerner og Betzig brugte enkelte molekyler til at afbilde en tæt række af molekyler på forskellige tidspunkter.
I The Optical Society's journal for high impact research, Optica , forskerholdet ledet af Moerner beskriver deres nye teknik og demonstrerer den ved at opnå superopløsningsbilleder og orienteringsmålinger for tusindvis af enkelte fluorescerende farvestofmolekyler knyttet til DNA-strenge.
"Du kan tænke på disse nye målinger som at give små dobbelthovedede pile, der viser orienteringen af molekylerne fastgjort langs DNA-strengen, " sagde Moerner. "Denne orienteringsinformation rapporterer om den lokale struktur af DNA-baserne, fordi de begrænser molekylet. Hvis vi ikke havde denne orienteringsinformation, ville billedet bare være et sted."
Tilføjelse af mere nanoskala information
En DNA-streng er en meget lang, men smal streng, kun et par nanometer på tværs. Enkeltmolekyle mikroskopi, sammen med fluorescerende farvestoffer, der binder til DNA, kan bruges til bedre at visualisere denne lille streng. Indtil nu, det var svært at forstå, hvordan disse farvestoffer var orienteret og umuligt at vide, om det fluorescerende farvestof var knyttet til DNA'et på en stiv eller noget løs måde.
Adam S. Backer, avisens første forfatter, udviklet en ret simpel måde at opnå orientering og rotationsdynamik fra tusindvis af enkelte molekyler parallelt. "Vores nye billedbehandlingsteknik undersøger, hvordan hvert enkelt farvestofmolekyle, der mærker DNA'et, er justeret i forhold til den meget større struktur af DNA, " sagde Backer. "Vi måler også, hvor vaklende hvert af disse molekyler er, som kan fortælle os, om dette molekyle sidder fast i en bestemt justering, eller om det flopper rundt i løbet af vores målesekvens."
Den nye teknik tilbyder mere detaljeret information end nutidens såkaldte "ensemble"-metoder, som gennemsnit af orienteringerne for en gruppe molekyler, og det er meget hurtigere end konfokale mikroskopiteknikker, som analyserer et molekyle ad gangen. Den nye metode kan endda bruges til molekyler, der er relativt svage.
Fordi teknikken giver nanoskala information om selve DNA'et, det kan være nyttigt til at overvåge ændringer i DNA-konformation eller beskadigelse af en bestemt region af DNA'et, hvilket ville vise sig som ændringer i orienteringen af farvestofmolekyler. Det kan også bruges til at overvåge interaktioner mellem DNA og proteiner, som driver mange cellulære processer.
30, 000 enkelt-molekyle orienteringer
Forskerne testede den forbedrede DNA-billeddannelsesteknik ved at bruge den til at analysere et interkalerende farvestof; en type fluorescerende farvestof, der glider ind i områderne mellem DNA-baserne. I et typisk billedeksperiment, de får op til 300, 000 enkelt molekyle steder og 30, 000 enkelt-molekyle orienteringsmålinger på lidt over 13 minutter. Analysen viste, at de enkelte farvestofmolekyler var orienteret vinkelret på DNA-strengens akse, og at mens molekylerne havde en tendens til at orientere sig i denne vinkelrette retning, de bevægede sig også rundt inden for en begrænset kegle.
Efterforskerne udførte derefter en lignende analyse ved hjælp af en anden type fluorescerende farvestof, der består af to dele:en del, der fæstner til siden af DNA'et, og en fluorescerende del, der er forbundet via en floppy tether. Den forbedrede DNA-billeddannelsesteknik detekterede denne uklarhed, viser, at metoden kan være nyttig til at hjælpe videnskabsmænd med at forstå, på molekyle for molekyle basis, om forskellige mærker binder til DNA på en mobil eller fast måde.
I avisen, forskerne demonstrerede en rumlig opløsning på omkring 25 nanometer og målinger af enkeltmolekyleorientering med en nøjagtighed på omkring 5 grader. De målte også rotationsdynamikken, eller diskrethed, af enkeltmolekyler med en nøjagtighed på omkring 20 grader.
Hvordan det virker
For at opnå information om enkeltmolekyleorientering, forskerne brugte en velundersøgt teknik, der tilføjer et optisk element kaldet en elektro-optisk modulator til enkeltmolekylemikroskopet. For hver kameraramme, this device changed the polarization of the laser light used to illuminate all the fluorescent dyes.
Since fluorescent dye molecules with orientations most closely aligned with the laser light's polarization will appear brightest, measuring the brightness of each molecule in each camera frame allowed the researchers to quantify orientation and rotational dynamics on a molecule-by-molecule basis. Molecules that switched between bright and dark in sequential frames were rigidly constrained at a particular orientation while those that appeared bright for sequential frames were not rigidly holding their orientation.
"If someone has a single-molecule microscope, they can perform our technique pretty easily by adding the electro-optic modulator, " said Backer. "We've used fairly standard tools in a slightly different way and analyzed the data in a new way to gain additional biological and physical insight."
 Varme artikler
Varme artikler
-
 For første gang, forskere fanger elektronbevægelser inde i en solcelleSkemaet viser den tidsopløste fotoemissionselektronmikroskopiinstrumentering, der gjorde det muligt for Femtosecond Spectroscopy Unit at visualisere elektronbevægelser. 800nm pumpeimpulsen (rød) exc
For første gang, forskere fanger elektronbevægelser inde i en solcelleSkemaet viser den tidsopløste fotoemissionselektronmikroskopiinstrumentering, der gjorde det muligt for Femtosecond Spectroscopy Unit at visualisere elektronbevægelser. 800nm pumpeimpulsen (rød) exc -
 Forsamlingsfrihed:Forskere ser nanopartikler danne større strukturer i realtidGuld nanopartikler samles selv til lange kæder, når de bombarderes med elektroner. Kredit:Argonne National Laboratory (Phys.org) — I en ny undersøgelse udført ved Center for Nanoscale Materials ve
Forsamlingsfrihed:Forskere ser nanopartikler danne større strukturer i realtidGuld nanopartikler samles selv til lange kæder, når de bombarderes med elektroner. Kredit:Argonne National Laboratory (Phys.org) — I en ny undersøgelse udført ved Center for Nanoscale Materials ve -
 Nanobot -pumper ødelægger nervemidlerEnzym -nanobotter pumper væske og omdanner nervemidler til harmløse produkter. Kredit:Ayusman Sen En gang på science fiction -området, nanobotter er tættere på end nogensinde at blive en realitet,
Nanobot -pumper ødelægger nervemidlerEnzym -nanobotter pumper væske og omdanner nervemidler til harmløse produkter. Kredit:Ayusman Sen En gang på science fiction -området, nanobotter er tættere på end nogensinde at blive en realitet, -
 Nanospears leverer genetisk materiale til celler med stor nøjagtighedKredit:American Chemical Society UCLA-forskere har udviklet en ny metode, der anvender mikroskopiske splinterlignende strukturer kaldet nanospears til målrettet levering af biomolekyler såsom gene
Nanospears leverer genetisk materiale til celler med stor nøjagtighedKredit:American Chemical Society UCLA-forskere har udviklet en ny metode, der anvender mikroskopiske splinterlignende strukturer kaldet nanospears til målrettet levering af biomolekyler såsom gene
- Forurenet luft fra Delhi ligner dødsdom, siger læger
- Turbulens skader 30 på flyvningen fra Istanbul til New York
- På vej tilbage til kontoret? Ikke alle afbrydelser på arbejdspladsen er dårlige for erhvervslivet
- Forskere så på havniveau 125, 000 år i fortiden. Resultaterne er skræmmende
- Forskere skaber et bakteriebaseret lægemiddelleveringssystem, der udkonkurrerer konventionelle meto…
- Top 5 grønne gadgets


