Design af peptidhæmmere til mulige COVID-19-behandlinger
Kredit:American Chemical Society
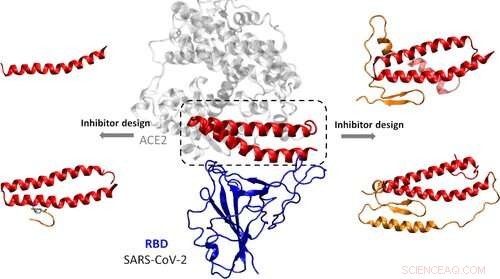
Forskere verden over skynder sig at finde hæmmere af SARS-CoV-2, den nye coronavirus bag COVID-19-pandemien. Nogle bruger computersimuleringer til at identificere lovende forbindelser, før de udfører egentlige eksperimenter i laboratoriet. Nu, forskere rapporterer i ACS Nano har brugt computermodellering til at vurdere fire peptider, der efterligner det virusbindende domæne for det humane protein, der tillader SARS-CoV-2 at komme ind i celler.
For at inficere celler, SARS-CoV-2 bruger sit spike-protein til at fæstne til ACE2-receptoren, et protein på overflader af visse menneskelige celler. Denne vedhæftning lader virussen smelte sammen med værtscellemembranen og få adgang. Mange forskere har forsøgt at finde forbindelser, der blokerer nøgleområder i piggproteinet, forhindrer virussen i at inficere celler. Yanxiao Han og Petr Král ønskede at bruge computermodellering til at designe forbindelser, der efterligner piggproteinets naturlige mål, ACE2.
For at gøre det, forskerne undersøgte den nyligt offentliggjorte røntgenkrystalstruktur af det receptorbindende domæne i SARS-CoV-2, når det er bundet til ACE2. De identificerede 15 aminosyrer fra ACE2, der interagerer direkte med det virale protein. Derefter, forskerne designede fire hæmmere, der indeholder de fleste eller alle disse aminosyrer, med yderligere sekvenser, som de troede ville stabilisere strukturerne. Gennem computersimuleringer, teamet undersøgte, hvordan inhibitorerne kan fæstne sig til piggproteinet i kroppen og de energier, der er nødvendige for binding. En af forbindelserne viste en særlig god pasform med det virale protein. Peptidet mangler stadig at blive testet i laboratoriet og hos patienter, men at kunne indsnævre lægemiddelkandidater på computeren kan hjælpe med at fremskynde denne proces, siger teamet.
Sidste artikelForskere opdager nye funktioner i molekylær elevator
Næste artikelSkubber grænserne for 2-D supramolekyler
 Varme artikler
Varme artikler
-
 Forskere opnår en universel vej til familien af penta-tvilling guld nanokrystallerKarakterisering af tre typiske penta-twinnede Au NCer. Kredit:Zhang Tao Et forskerhold ledet af prof. Li Yue fra Hefei Institutes of Physical Science (HFIPS) under det kinesiske videnskabsakademi
Forskere opnår en universel vej til familien af penta-tvilling guld nanokrystallerKarakterisering af tre typiske penta-twinnede Au NCer. Kredit:Zhang Tao Et forskerhold ledet af prof. Li Yue fra Hefei Institutes of Physical Science (HFIPS) under det kinesiske videnskabsakademi -
 Anden hud beskytter mod kemikalier, biologiske midlerDen smarte beskyttelsesmekanisme for responsive nanorørsmembraner mod miljøtrusler. Sammenbruddet af aktiverende polymerkæder på den forurenede membranoverflade forhindrer nervemidler som sarin i at t
Anden hud beskytter mod kemikalier, biologiske midlerDen smarte beskyttelsesmekanisme for responsive nanorørsmembraner mod miljøtrusler. Sammenbruddet af aktiverende polymerkæder på den forurenede membranoverflade forhindrer nervemidler som sarin i at t -
 Hjernetemperaturen kan nu måles ved hjælp af lysKredit:CC0 Public Domain Lys kunne erstatte invasive teknikker til måling af hjernetemperatur – hvilket eliminerer behovet for at placere et termometer i hjernen, når man behandler en række neurol
Hjernetemperaturen kan nu måles ved hjælp af lysKredit:CC0 Public Domain Lys kunne erstatte invasive teknikker til måling af hjernetemperatur – hvilket eliminerer behovet for at placere et termometer i hjernen, når man behandler en række neurol -
 Ny enhed i nanoskala til spinteknologiMagneto-optisk mikroskop brugt til billeddannelse af spin-bølger i en Fabry-Pérot-resonator. Kredit:Matt Allinson, Aalto Universitet Forskere ved Aalto Universitet har udviklet en ny enhed til spi
Ny enhed i nanoskala til spinteknologiMagneto-optisk mikroskop brugt til billeddannelse af spin-bølger i en Fabry-Pérot-resonator. Kredit:Matt Allinson, Aalto Universitet Forskere ved Aalto Universitet har udviklet en ny enhed til spi
- Hvordan landet genopretter sig efter naturbrande - mener en ekspert
- Trump ringede til Boeing CEO forud for MAX-lukningen:kilde
- Grafenforskere skaber overophedet vand, der kan korrodere diamanter
- Hvordan statistikker gælder for March Madness
- Blegning rammer verdens sydligste koralrev:videnskabsmænd
- Nye ledetråde til dybt jordskælvsmysterium


