Lagring af oplysninger i DNA:Forbedring af DNA-lagring med elektrodebrønde i nanoskala
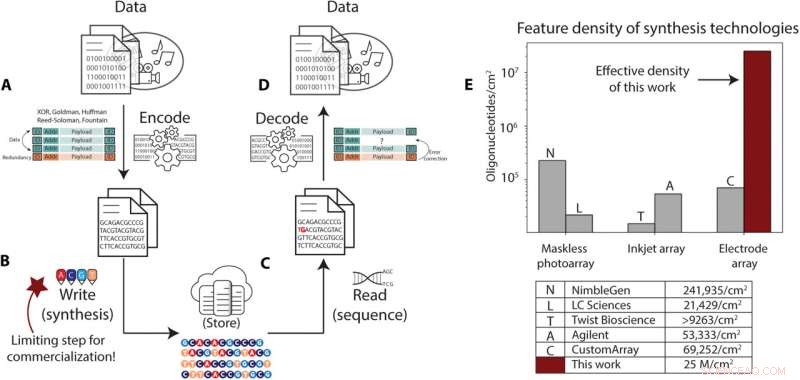
Lagring af DNA-data kræver højere syntesegennemstrømning, end det er muligt med nuværende teknikker. (A til D) Oversigt over pipeline til lagring af DNA-data. (A) Digitale data kodes fra deres binære repræsentation til sekvenser af DNA-baser, med en identifikator, der korrelerer dem med et dataobjekt, adresseringsinformation, der bruges til at omorganisere dataene ved læsning, og redundant information, der bruges til fejlkorrektion. (B) Disse sekvenser syntetiseres til DNA-oligonukleotider og opbevares. (C) Ved genfinding udvælges og kopieres DNA-molekylerne via PCR eller andre metoder og sekventeres tilbage til elektroniske repræsentationer af baserne i disse sekvenser. (D) Afkodningsprocessen tager dette støjende og nogle gange ufuldstændige sæt af sekventeringslæsninger, korrigerer for fejl og manglende sekvenser og afkoder informationen for at gendanne dataene. (E) Sammenfatning af de kommercielle synteseprocesser og tilsvarende estimerede oligonukleotiddensiteter, som rapporteret i litteraturen eller af virksomhederne selv. Vores elektrokemiske metodetæthed er fremhævet i mørkerødt. Kredit:Science Advances , 10.1126/sciadv.abi6714
Genetikere kan lagre data i syntetisk DNA som et medium til langtidslagring på grund af dens tæthed, nemme kopiering, levetid og bæredygtighed. Forskningen på området var for nylig kommet videre med nye kodningsalgoritmer, automatisering, bevaring og sekventering. Ikke desto mindre er den mest udfordrende forhindring i implementering af DNA-lagring fortsat skrivegennemstrømningen, som kan begrænse datalagringskapaciteten. I en ny rapport udviklede Bichlien H. Nguyen og et team af forskere inden for Microsoft Research og datalogi og teknik ved University of Washington, Seattle, USA, den første nanoskala-DNA-lagerskriver. Holdet havde til hensigt at skalere DNA-skrivetætheden til 25 x 10 6 sekvenser pr. kvadratcentimeter, en forbedret lagerkapacitet sammenlignet med eksisterende DNA-syntese-arrays. Forskerne skrev og afkodede med succes en besked i DNA for at etablere et praktisk DNA-datalagringssystem. Resultaterne er nu offentliggjort i Science Advances .
Langsigtede DNA-arkiver
Det nuværende tempo i datagenerering overstiger eksisterende lagerkapacitet, DNA er en lovende løsning på dette problem med en forventet praktisk tæthed på mere end 60 petabyte pr. kubikcentimeter. Materialet er holdbart under en række forhold, relevant og nemt at kopiere, med løfte om at være mere bæredygtigt eller grønnere end kommercielle medier. Under processen kan digitale data i form af sekvenser af bit kodes i sekvenser af de fire naturlige DNA-baser - guanin, adenin, thiamin og cytosin, selvom yderligere baser også er mulige. Holdet kan derefter skrive sekvenserne til molekylær form via de novo DNA-oligonukleotidsyntese for at skabe specifikke molekyler baseret på et sæt af gentagne kemiske trin. De resulterende oligonukleotider kan konserveres og opbevares efter syntese. For at få adgang til dataene kan DNA-lageret amplificeres ved hjælp af polymerasekædereaktioner og sekventeres for at returnere DNA-basesekvenserne til det digitale domæne, hvorefter DNA-basesekvenserne kan afkodes for at genvinde den oprindelige sekvens af bits.
Oversigt over 650-nm array med en pitch på 2 μm. (A) Finite element-analyse af anodsyredannelse og -diffusion ved en 650-nm-diameter elektrode med en 200-nm-brønd er afbildet med et tværsnit langs y =x-planet og (B) top-down-billede på z =0 plan. Farverne blå og gul repræsenterer områder med henholdsvis relativt lave og høje syrekoncentrationer. (C) En oversigt over DNA-syntesearrayet i nanoskala med scanningselektronmikroskopibilleder af 650-nm elektrodearrayet og forstørret billede af en elektrode. (D) Et fluorescerende billede, hvor brønden, der omgiver hver aktiveret anode, er mønstret med AAA-fluorescein. Tegneseriediagrammet viser, hvilke elektroder i layoutet der blev aktiveret. (E) Illustration af brøndene mønstret med AAA-fluorescein og AAA-AquaPhluor og (F) tilsvarende billedoverlejring af de to fluoroforer i enden af DNA syntetiseret på det samme 650-nm elektrodearray. Kredit:Science Advances , 10.1126/sciadv.abi6714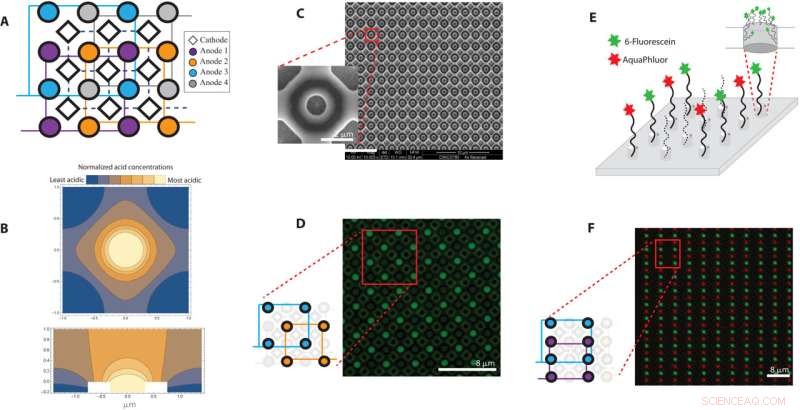
I denne undersøgelse har Nguyen et al. producerede et elektrodearray, som demonstrerede uafhængig elektrodespecifik kontrol af DNA-syntese med elektrodestørrelser og stigninger for at etablere syntesetæthed på 25 millioner oligonukleotider pr. cm 2 . Denne værdi estimeres som den elektrodetæthed, der kræves for at opnå minimumsmålet på kilobyte pr. sekund af datalagring i DNA. Holdet pressede på det nyeste inden for elektronisk-kemisk kontrol og leverede eksperimentelt bevis for den skrivebåndbredde, der er nødvendig for lagring af DNA-data.
Holdet introducerede en proof-of-concept molekylær controller i form af en lille DNA-lagring skrivemekanisme på en chip. Chippen kunne tæt pakke DNA-syntese i 3 størrelsesordener højere end før for at opnå større DNA-skrivegennemstrømning. For at lagre information i DNA i den skala, der er nødvendig til kommerciel brug, krævede to afgørende processer. Først skulle holdet oversætte digitale bits (etler og nuller) til strenge af syntetisk DNA, der repræsenterer bits med kodningssoftware og en DNA-synthesizer. Så skal de være i stand til at læse og afkode informationen tilbage til dens bits for at gendanne den information til digital form igen med en DNA-sequencer og afkodningssoftware.
Udvikling af elektrokemiske arrays til funktioner i nanoskala
Under den traditionelle syntese af DNA-kæder bruger videnskabsmænd en flertrinsmetode kendt som phosphoramidit-kemi, hvor en DNA-kæde kan dyrkes sekventielt ved tilsætning af DNA-baser. Hver DNA-base indeholder en blokerende gruppe for at forhindre flere tilføjelser af DNA-baser til den voksende kæde. Ved binding til en DNA-kæde kan syre leveres i opsætningen for at spalte den blokerende gruppe og prime DNA-kæden for at tilføje den næste base. Under elektrokemisk DNA-syntese indeholder hver plet i arrayet en elektrode, og når der påføres en spænding, genereres syre ved arbejdselektroden (anode) for at afblokere de voksende DNA-kæder, mens en ækvivalent base genereres ved modelektroden (katoden) . Holdet forhindrede syrediffusion i opsætningen ved at designe et elektrodearray, hvor hver arbejdselektrode, omkring hvilken syredannelse fandt sted under DNA-syntese, blev sænket i en brønd og omgivet af fire fælles modelektroder, dvs. katoder, der drev basedannelse, for at begrænse syren til specifikke regioner. Nguyen et al. verificeret effektiviteten af designet ved hjælp af finite element analyse. Under eksperimenterne, når den blev præsenteret i tilstrækkelig koncentration, deblokerede syren de overfladebundne nukleotider for at tillade det næste nukleotid at koble sig. Ved at bruge opsætningen af chips, der indeholder funktionspletter til at begrænse syrer, udviklede de elektrokemiske arrays med fire individuelle elektroder til at regulere DNA-syntese. Holdet udførte derefter eksperimenter med to fluorescerende mærkede baser i grønt og rødt. Som bevis på konceptet viste de enhedens kapacitet til at skrive data ved at syntetisere fire unikke DNA-strenge, hver 100 baser lange med en kodet besked, uden fejl.
-
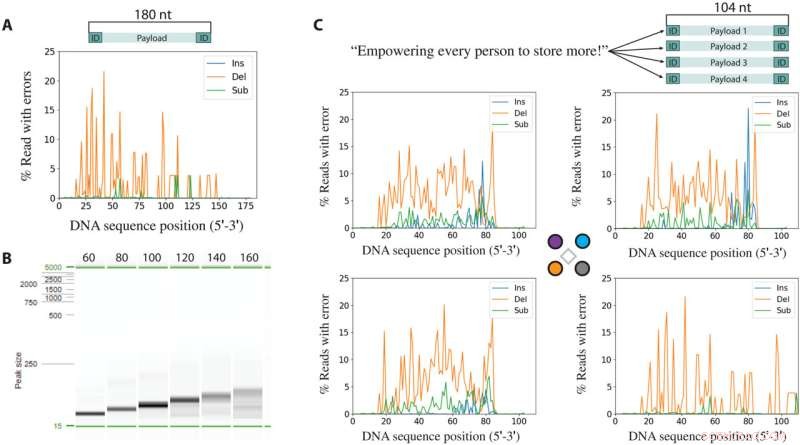
Fejl, der stammer fra syntese efterfulgt af sekventering. (A) Insertioner (Ins), deletioner (Del) og substitutioner (Sub) pr. position for en syntetiseret og PCR-amplificeret 180-baser sekvens. (B) Elektroforesebillede af synteseprodukter efter PCR-amplifikation. (C) Meddelelse kodet i 64 bytes opdelt i fire unikke sekvenser på 104 baser (øverst). Insertioner, deletioner og substitutioner pr. locus af hver af de fire sekvenser i multiplekssyntesekørsel. I hver fejlanalysegraf kommer de terminale 20 baser ved både 3'- og 5'-ender fra de primere, der anvendes i PCR og er ikke repræsentative for de syntetiserede fejl. Kredit:Science Advances , 10.1126/sciadv.abi6714
-
Skalering af DNA-datalagring med elektrodebrønde i nanoskala. Lille DNA-lagring skrivemekanisme på en chip. Kredit:Microsoft Research Blog, Science Advances , 10.1126/sciadv.abi6714
Outlook:Syntetisering af korte oligonukleotider på elektrodearrayet til datalagring
Ved hjælp af opsætningen har Nguyen et al. demonstrerede også rumligt kontrolleret syntese af korte oligonukleotider på elektrodearrayet for at vurdere den maksimale længde af DNA, der kunne dannes. Forskerne skabte en enkelt DNA-sekvens med 180 nukleotider og PCR-amplificerede produkter med forskellige længder fra hele længden af oligonukleotiderne. Efterhånden som amplikonet blev længere, virkede de forventede PCR-produkter svagere og mindre veldefinerede, mens kortere amplikoner viste stærkere og mere veldefinerede bånd, der indikerer højere syntesefejl. Baseret på resultaterne valgte forskerne sekvenslængde svarende til 100 baser for at lette oprensningen for at give en praktisk demonstration af lagring af DNA-data uden yderligere optimering. På denne måde banede proof-of-concept-metoden, som blev demonstreret i dette arbejde af Bichlien H. Nguyen og kolleger, vejen frem for at generere storskala og unikke DNA-sekvenser parallelt til datalagring. Arbejdet overgik tidligere rapporter om tætte syntetiske DNA-sekvenser for at give en første eksperimentel indikation for at opnå den skrivebåndbredde, der kræves til datalagring ved funktionsstørrelser på nanoskala. Forskerne forventer øjeblikkelig anvendelse af enhederne i informationsteknologi og forudser deres praktiske anvendelser inden for materialevidenskab, syntetisk biologi og storskala molekylærbiologiske assays. + Udforsk yderligere
Enzymatisk DNA-syntese ser lyset
© 2021 Science X Network
 Varme artikler
Varme artikler
-
 Ny DNA-baseret chip kan programmeres til at løse komplekse matematiske problemerDNA computing, såsom beregningerne udført af den nye DNA-baserede mikrochip, har potentialet til at udføre komplekse matematiske funktioner lettere end konventionelle elektroniske computere kan. Kredi
Ny DNA-baseret chip kan programmeres til at løse komplekse matematiske problemerDNA computing, såsom beregningerne udført af den nye DNA-baserede mikrochip, har potentialet til at udføre komplekse matematiske funktioner lettere end konventionelle elektroniske computere kan. Kredi -
 Hård carbon nanofiber aerogel bliver superelastiskDug på et edderkoppespind om morgenen. Kredit:Wikipedia/Luc Viatour/Lucnix.be Ledende og komprimerbare carbon aerogeler er nyttige i en række forskellige applikationer. I de seneste årtier har car
Hård carbon nanofiber aerogel bliver superelastiskDug på et edderkoppespind om morgenen. Kredit:Wikipedia/Luc Viatour/Lucnix.be Ledende og komprimerbare carbon aerogeler er nyttige i en række forskellige applikationer. I de seneste årtier har car -
 Forskere skaber nanostrukturer lavet af rent guldNanostruktur lavet af guld. Kredit:Vienna University of Technology Det er Nanoteknologiens De Vises Sten:ved at bruge et teknologisk trick, Det er lykkedes forskere ved TU Wien (Wien) at skabe nan
Forskere skaber nanostrukturer lavet af rent guldNanostruktur lavet af guld. Kredit:Vienna University of Technology Det er Nanoteknologiens De Vises Sten:ved at bruge et teknologisk trick, Det er lykkedes forskere ved TU Wien (Wien) at skabe nan -
 Molekylær beacons skinner lys på, hvordan celler kravlerVores forudsætning er, at mekanik spiller en rolle i næsten alle biologiske processer, og med disse DNA-baserede spændingsprober vil vi afsløre, måle og kortlægge disse kræfter, ” siger biomolekylær k
Molekylær beacons skinner lys på, hvordan celler kravlerVores forudsætning er, at mekanik spiller en rolle i næsten alle biologiske processer, og med disse DNA-baserede spændingsprober vil vi afsløre, måle og kortlægge disse kræfter, ” siger biomolekylær k
- Hvis du tror, at Jupiter kredser om solen,
- Hvad er Math omgruppering?
- Over tusinde kosmiske eksplosioner opdaget på 47 dage
- Kunstig intelligens bruges til bedre at overvåge Maines skove
- Hvad er forskellen mellem en transformator og en ensretter?
- Forskere på forkant med udvikling af maskinlæringsmetoder til kemisk opdagelse


