Fingeraftryksproteiner med kraft åbner en ny vej for enkelt-molekyle proteomics
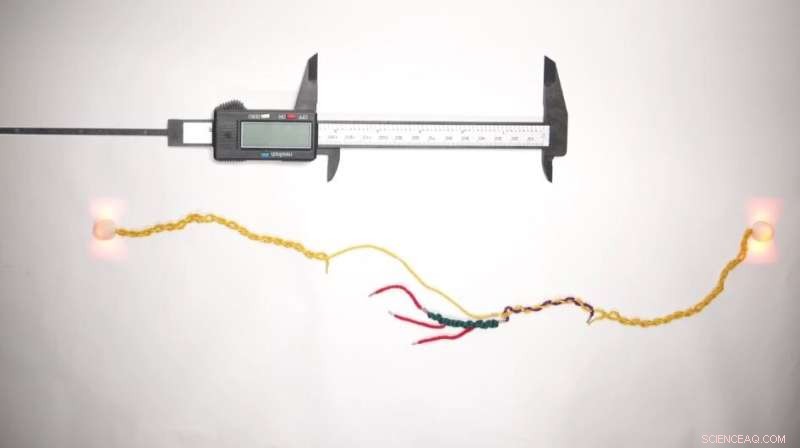
Kredit:Harvard University
Efterhånden som videnskabsmænd har sonderet livets mysterier ned til mindre og mindre skalaer, har de opfundet værktøjer til at hjælpe dem med at forstå, hvad de observerer. Fastlæggelse af identiteten af DNA- og RNA-molekyler er nu blevet almindeligt takket være den kommercielle udvikling af næste generations sekventeringsteknologier, men det samme er endnu ikke tilfældet for proteiner, som er kritisk vigtige spillere i næsten alle biologiske processer. Proteiner er meget mere komplekse end DNA og RNA og er ofte kemisk modificerede, hvilket gør målet om let at identificere enkelte proteiner i en prøve (enkeltmolekyle-proteomik) udfordrende at opnå.
Nu har forskere, der arbejder ved Molecular Robotics Initiative inden for Wyss Institute ved Harvard University, Blavatnik Institute ved Harvard Medical School (HMS) og Boston Children's Hospital (BCH), brugt DNA, det grundlæggende i selve livet, til at skabe, hvad der evt. være verdens mindste lineal til at måle proteiner.
Denne teknologi, der kaldes "DNA Nanoswitch Calipers" (DNC), gør det muligt for forskere at udføre afstandsmålinger på enkelte peptider (proteinernes byggesten) med høj præcision ved at anvende små mængder kraft. Ved hurtigt at lave mange afstandsmålinger på det samme molekyle skaber DNC et unikt "fingeraftryk", som kan bruges til at identificere det i efterfølgende eksperimenter. Præstationen er rapporteret i Nature Nanotechnology .
"Når du forsøger at forstå noget i biologi, er der to hovedmetoder til undersøgelse:du kan observere dit emne i dets naturlige tilstand, eller du kan forstyrre det og se, hvordan det reagerer. Observationer kan give masser af fantastisk biologisk information, men nogle gange er den bedste måde at lære om noget på fysisk at interagere med det," sagde den medkorresponderende forfatter Wesley Wong, Ph.D., Associate Faculty Member ved Wyss Institute og Associate Professor ved HMS, som også er investigator ved BCH . "At bestemme mønsteret af aminosyrer i et peptidmolekyle ved at anvende kraft er et nyt paradigme i den igangværende videnskabelige søgen efter teknikker, der vil gøre os i stand til at sekventere proteiner lige så let, som vi i øjeblikket sekventerer DNA."
Brug kraften
DNC er baseret på DNA-nanoswitchens underliggende teknologi:en enkelt DNA-streng med molekylære "håndtag" knyttet til den på flere punkter langs dens længde. Når to af disse håndtag binder til hinanden, skaber de en løkke i DNA-strengen, og den samlede længde af strengen forkortes. Når der påføres kraft for at trække håndtagene fra hinanden, strækker strengen sig tilbage til sin oprindelige længde. Forskellen mellem længden af strengen i dens løkkede og ikke-løkkede tilstand afspejler løkkens størrelse og dermed afstanden mellem håndtagene.
Forskerholdet indså, at de kunne tage DNA nanoswitches et skridt videre:hvis de i stedet konstruerede håndtagene til at binde til et biomolekyle, kunne håndtagene effektivt "klemme" molekylet mellem sig som de to spidser af en skydelære, i stedet for at binde til hver Andet. Ved at måle, hvordan tilføjelsen af målmolekylet mellem håndtagene ændrede den samlede længde af DNA-nanoswitchen i dens sløjfede vs. ikke-sløjfede tilstande, antog holdet, at de effektivt kunne måle størrelsen af molekylet.
"På nogle måder udnytter DNA-nanoswitches en af de mest klassiske, mekaniske metoder til at måle objekter:bare påfør kraft på noget og se, hvordan det ændrer sig som reaktion," sagde medforfatter Darren Yang, en postdoktor ved Wyss Institute og BCH. "Det er en tilgang, som vi ikke rigtig har set brugt inden for enkelt-molekyle proteomics, fordi det er utroligt udfordrende at anvende kraft på så små objekter. Men vi var op til udfordringen."
For at omsætte deres idé om en ny, kraftbaseret måleteknik til virkelighed, fastgjorde Yang og hans kolleger først to forskellige typer håndtag til et målmolekyle:et "stærkt" håndtag til fast forankring af molekylet til den ene ende af DNC'en, og flere "svage" håndtag, der kunne fastgøres til den anden ende af DNC'en. De bundede derefter begge ender af DNC'en til to "optisk fanget" perler suspenderet i laserstråler. Ved at flytte perlerne tættere på hinanden inducerede de et af målmolekylets svage håndtag til at binde sig til DNC'en, hvilket skabte en sløjfetilstand. Da de derefter øgede kraften ved at flytte perlerne længere fra hinanden, frigjorde det svage håndtag til sidst sin binding, og returnerede DNC'en til sin længere, uløkkede tilstand.
Holdet testede først denne teknik på simple, enkeltstrengede DNA (ssDNA) molekyler og bekræftede, at ændringen i afstandsmålinger mellem DNC's sløjfede og uløkkede tilstand direkte korrelerede med længden af målmolekylet. Disse længdeændringer kunne måles med præcision på angstrom-niveau (det er ti gange mindre end bredden af en DNA-dobbelthelix), hvilket muliggør identifikation af ændringer i længden så små som for et enkelt nukleotid.
Fordi målmolekylet indeholder flere svage håndtag, der kan binde til DNC'en, skaber gentagne cyklusser med binding og brud af disse håndtag en række afstandsmålinger mellem det stærke håndtag og de svage håndtag, der er unikke for hvert målt molekyle. Dette "fingeraftryk" kan bruges til at identificere et kendt molekyle i en prøve eller til at udlede strukturel information om et ukendt molekyle.
Søge proteiner
Efter at have bekræftet, at DNC pålideligt kunne måle størrelsen af DNA-molekyler, flyttede forskerne fokus til deres egentlige mål:proteiner. De designede et syntetisk peptid (en kort kæde af aminosyrer) med en kendt længde og sekvens og gentog eksperimentet og fastgjorde det til den ene ende af DNC via det stærke håndtag og gentagne gange fastgør og bryde bindingerne mellem dets svage håndtag og DNC'en ved at anvende forskellige mængder kraft. De fandt ud af, at alle de afstande, deres værktøj målte mellem de stærke og svage håndtag, matchede de forventede afstande baseret på længden af DNC og længden af aminosyrerne i peptidet. De fik også lignende resultater, da de brugte DNC til at måle et naturligt forekommende lineariseret peptid kaldet NOXA BH3.
Denne proces genererede også unikke målingsfingeraftryk for hvert peptid. Holdet skabte en computermodel til at forudsige, hvor mange humane proteiner der kunne identificeres entydigt ved hjælp af denne metode, og fandt ud af, at over 75 % af proteinerne i en almindeligt anvendt proteindatabase kunne identificeres via fingeraftryk med en sandsynlighed på mindst 90 %.
"Vi var faktisk noget overrasket over, hvor godt denne teknik virkede," sagde medforfatter Prakash Shrestha, Ph.D., en postdoktor ved Wyss Institute og BCH. "Optisk pincet har eksisteret i årtier, og cyklisk DNA mellem en sløjfet og ikke-sløjfet tilstand har eksisteret i omkring 10 år, og vi var ikke sikre på, om vi kunne få tilstrækkelig højopløselige målinger ved at kombinere disse ideer. Men det viste sig, at disse fingeraftryk er meget effektive til at identificere proteiner."
At identificere enkelte proteinmolekyler er en imponerende bedrift i sig selv, men at være i stand til at gøre det for flere proteiner samtidigt er den sande hellige gral for enkelt-molekyle proteomics. Holdet demonstrerede yderligere, at de ved at erstatte de optiske perler med et magnetisk pincetsystem var i stand til at udføre målinger på flere forskellige peptider parallelt, samt bestemme de relative koncentrationer af forskellige molekyler.
"Single-molecule proteomics er stadig i vid udstrækning en drøm på grund af udfordringer i skalering og opløsning. Vores nuværende arbejde viser, at force-baseret sekvens fingerprinting har potentialet til at realisere denne drøm," sagde co-korrespondrende forfatter William Shih, Ph.D. , et kernefakultetsmedlem ved Wyss Institute og professor ved HMS og Dana-Farber Cancer Institute. "Vores ultimative ambition er effektivt at læse ikke kun proteinsekvenser, men også proteinstrukturer på en måde med høj gennemstrømning."
Forskernes næste skridt i retning af dette mål er at validere deres kalibere til strukturelle målinger med lav kraft på foldede proteiner og deres komplekser, og undersøge deres potentielle anvendelse til strukturel biologi og proteomik. De arbejder også på at øge teknologiens gennemløb for yderligere at fremskynde analysen af blandede prøver.
"Denne forskning integrerer molekylær biofysik med banebrydende DNA-nanoteknologi, som er banebrydende her på Wyss Institute for at give os mulighed for at interagere med og analysere biologiske molekyler på en virkelig ny måde. Da William og Wesley først stillede denne idé som en kerneudfordring for de nydannede Molecular Robotics Initiative, det virkede virkelig som science fiction, men det er netop den type projekt, vi ønsker at påtage os på Wyss. Jeg er meget stolt af holdet for at gøre denne teknologi til virkelighed - den har potentialet til at ændre sig totalt. hvordan vi laver videnskab og udvikler terapeutiske midler," sagde Wyss stiftende direktør Don Ingber, M.D., Ph.D., som også er Judah Folkman professor i vaskulær biologi ved Harvard Medical School og BCH, og professor i bioingeniør ved Harvard John A. Paulson School of Engineering and Applied Sciences. + Udforsk yderligere
Demokratisering af high-throughput enkelt molekyle kraftanalyse
Sidste artikelAfmasker superledningsmagien i snoet grafen
Næste artikelEdgy lys på grafen kan bringe nye envejs informationsroutere
 Varme artikler
Varme artikler
-
 Forskere offentliggør en grundlæggende undersøgelse af nanopartiklers indvirkning på levende art…Kredit:University of Plymouth Et internationalt team af forskere har afsluttet den første undersøgelse nogensinde af den potentielle indvirkning af naturligt forekommende og menneskeskabte nanopar
Forskere offentliggør en grundlæggende undersøgelse af nanopartiklers indvirkning på levende art…Kredit:University of Plymouth Et internationalt team af forskere har afsluttet den første undersøgelse nogensinde af den potentielle indvirkning af naturligt forekommende og menneskeskabte nanopar -
 Nogle piezoelektriske materialer kan være falskeKredit:CC0 Public Domain Piezorespons kraftmikroskopi (PFM) er den mest udbredte teknik til karakterisering af piezoelektriske egenskaber på nanoskala, dvs. til bestemmelse af visse materialers ev
Nogle piezoelektriske materialer kan være falskeKredit:CC0 Public Domain Piezorespons kraftmikroskopi (PFM) er den mest udbredte teknik til karakterisering af piezoelektriske egenskaber på nanoskala, dvs. til bestemmelse af visse materialers ev -
 Effektiv, miljøvenlig produktion af finkemikalierDet, der gør katalysatoren så effektiv, er, at palladiumatomet kan bevæge sig inde i hulrummet for at tilpasse sig proceskravene, mens det forbliver fanget inde, forklarer ETH-professor Javier Pérez-R
Effektiv, miljøvenlig produktion af finkemikalierDet, der gør katalysatoren så effektiv, er, at palladiumatomet kan bevæge sig inde i hulrummet for at tilpasse sig proceskravene, mens det forbliver fanget inde, forklarer ETH-professor Javier Pérez-R -
 Udvikling af en 3-D kollagenmodel til at teste magnetisk assisteret osteogenese in vitroa) Mikrostruktur af cellefyldte kollagen stilladser undersøgt under TEM. Celler er omgivet af kollagenmatrixen. Kollagenfibrillerne viste ingen foretrukken retning omkring cellerne under kontrolforhol
Udvikling af en 3-D kollagenmodel til at teste magnetisk assisteret osteogenese in vitroa) Mikrostruktur af cellefyldte kollagen stilladser undersøgt under TEM. Celler er omgivet af kollagenmatrixen. Kollagenfibrillerne viste ingen foretrukken retning omkring cellerne under kontrolforhol
- Thailandske skildpadder plastfyldt mave fremhæver havkrisen
- Martian Moons Exploration rumfartøj til at tage ultra-high definition billeder af Mars via 8K kamer…
- Hvorfor tiltrækker en magnet jern?
- Virksomheder har brug for en bedre tilgang til compliance-programmer, forskningsfund
- Angiv hvilke former for information der kan findes ved at kende sekvensen af en DNA Molecule
- Hvad andre planeters energikredsløb kan fortælle os om klimaændringer på Jorden


