Forskere opdager molekylære tilføjelser, der tilpasser proteingrænseflader
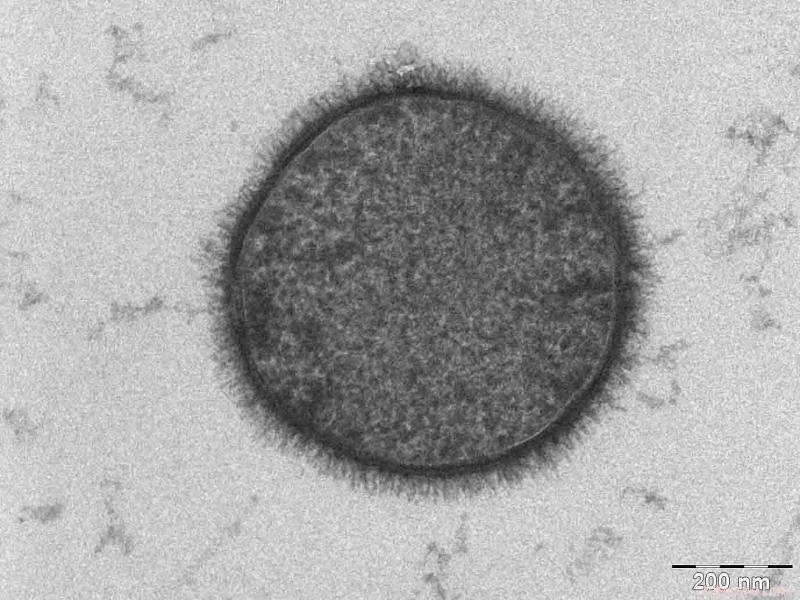
Bacterium Bacillus subtilis taget med en Tecnai T-12 TEM. Taget af Allon Weiner, Weizmann Institute of Science, Rehovot, Israel. 2006. Kredit:Public Domain
Forskere i USA og Tyskland har netop opdaget en tidligere overset del af proteinmolekyler, der kan være nøglen til, hvordan proteiner interagerer med hinanden inde i levende celler for at udføre specialiserede funktioner.
Forskerne opdagede bittesmå stykker molekylært materiale - som de kaldte "add-ons" - på de ydre kanter af proteingrænsefladen, der tilpasser, hvad et protein kan gøre. De valgte navnet, fordi tilføjelserne tilpasser grænsefladen mellem proteiner, som softwaretillæg tilpasser en webgrænseflade med en bruger.
Selvom det længe har været kendt, at proteiner har en grænsefladeregion, hvor de forbinder med andre proteiner, det har ikke været klart præcist, hvordan nøgleproteiner er i stand til at finde hinanden i et overfyldt cellulært miljø, der kan indeholde titusinder af andre proteiner.
Nu, forskere ved Ohio State University og University of Regensburg rapporterer i Proceedings of the National Academy of Sciences at det er tilføjelserne, der gør det muligt for proteiner at forbinde udelukkende med den rigtige dedikerede partner.
Florian Busch, en postdoc forsker i kemi og biokemi ved Ohio State og medforfatter af undersøgelsen, kaldte eksistensen af proteintilsætninger "et hidtil ukendt grundlæggende drivprincip" for at sikre, at proteiner interagerer på bestemte måder.
Forskerne eksperimenterede med levende bakterier, demonstrerer vigtigheden af tilføjelser til normale cellulære funktioner. For eksempel, det bestemte de i organismen Bacillus subtilis , hvor der mangler en unik grænsefladetilføjelse, bakteriekolonier voksede 80 procent mindre under visse forhold. Årsagen til dette var, at den manglende grænseflade-tilføjelse førte til usunde kryds-interaktioner af proteiner i B. subtilis celler.
Det er svært at overvurdere proteinernes betydning for livet, som vi kender det. Enzymer er proteiner, der muliggør kemiske reaktioner i celler. Antistoffer er proteiner, der binder til fremmede angribere i kroppen. Listen fortsætter med at inkludere tusindvis af kritiske funktioner. I de fleste tilfælde, proteiner skal forbindes med hinanden og danne grupper kaldet proteinkomplekser for at udføre så forskellige opgaver.
Men præcis, hvordan proteiner er i stand til at gøre alt, hvad de gør, er et mysterium - et mysterium med rod i matematik og geometri. Der er 20 kendte aminosyrer, som bindes sammen i lange kæder og derefter foldes sammen for at danne proteiner. Det er folden, der bestemmer et proteins generiske form, eller geometri. Selvom der kun er omkring 1, 000 kendte proteingeometrier i naturen, på en eller anden måde er proteiner i stand til at danne komplekser, der udfører hundredtusindvis af meget specifikke funktioner.
Maximilian Plach, hovedforfatter af papiret og biokemiker ved University of Regensburg, forklaret, hvordan forskerne vidste, hvor de skulle lede for at løse mysteriet.
"Meget arbejde er blevet lagt i at analysere, hvordan proteiner interagerer med hinanden, og hvordan grænsefladerne ser ud, hvordan de er opbygget, og hvordan de udviklede sig, " sagde han. "Men de perifere områder af grænseflader har ikke fået så meget opmærksomhed. Jeg tror, at det nye i vores tilgang var at se på regioner, der har været, endnu, betragtes som mindre vigtigt."
Regensburg-holdet, ledet af beregningsbiolog Rainer Merkl og proteinbiokemiker Reinhard Sterner, analyseret proteinsekvenserne afledt af mere end 15, 000 bakterielle og arkæale genomer på en stor computerklynge. De sorterede proteiner, der delte fælles evolutionære forfædre, i en slags stamtræ, og sammenlignede individuelle proteiner med deres protein-"slægtninge". Det var sådan, de opdagede grænsefladestrukturer, der var til stede i nogle proteiner, men mangler i andre - tilføjelserne.
Busch og Vicki Wysocki, Ohio Eminent Scholar af makromolekylær struktur og funktion og direktør for Campus Chemical Instrument Center i Ohio State, brugte derefter naturlig massespektrometri til at detektere, hvordan tilstedeværelsen og fraværet af tilføjelser påvirkede proteiners evne til at interagere med hinanden.
"Vi er virkelig glade for, at vores native massespektrometriteknologi kunne hjælpe med at identificere rollen for disse grænseflade-'add-ons' - en måde for et protein at finde sit kritiske partnerprotein selv i et overfyldt cellulært miljø med lignende strukturer til stede, " sagde Wysocki.
Til Busch, en af de virkelig spændende ting ved undersøgelsen var forskernes brug af "big data" - i dette tilfælde, hele protein- og genomdatabaser.
"Jeg anser vores arbejde for at være et vigtigt eksempel på, hvordan man gør brug af offentligt tilgængelige data for at forstå grundlæggende principper i naturen, og jeg tror, at data mining vil blive stadig vigtigere på det biomedicinske område i fremtiden, " han sagde.
 Varme artikler
Varme artikler
-
 Hvaler og delfiner har rige menneskelignende kulturer og samfundKredit:CC0 Public Domain Hvaler og delfiner (hvaler) lever i tætte sociale grupper, har komplekse forhold, tale med hinanden og endda have regionale dialekter - meget ligesom menneskelige samfund.
Hvaler og delfiner har rige menneskelignende kulturer og samfundKredit:CC0 Public Domain Hvaler og delfiner (hvaler) lever i tætte sociale grupper, har komplekse forhold, tale med hinanden og endda have regionale dialekter - meget ligesom menneskelige samfund. -
 Cellekontakter i embryonal udvikling bestemmer cellulær skæbneKunstnerisk 3D-gengivelse af en zebrafisk prechordal plade, viser stamceller, der enten danner en kompakt klynge og differentierer sig til prekordale pladeceller eller forlader klyngen og differentier
Cellekontakter i embryonal udvikling bestemmer cellulær skæbneKunstnerisk 3D-gengivelse af en zebrafisk prechordal plade, viser stamceller, der enten danner en kompakt klynge og differentierer sig til prekordale pladeceller eller forlader klyngen og differentier -
 Sporing af en parasit, der hærger fiskKredit:EPFL I Schweiz – for ikke at tale om resten af Europa og USA – bliver ferskvandsfisk ofre for en dødelig sygdom, der florerer om sommeren og hviler om vinteren. Det er forårsaget af en pa
Sporing af en parasit, der hærger fiskKredit:EPFL I Schweiz – for ikke at tale om resten af Europa og USA – bliver ferskvandsfisk ofre for en dødelig sygdom, der florerer om sommeren og hviler om vinteren. Det er forårsaget af en pa -
 Kan mad gøre folk glade?Du kommer til at spise det her i din bil, er du ikke? Scott Olson/Getty Images Du kører hjem efter en lang arbejdsdag. Du er ikke i humør til at lave mad, og du stopper ved en drive-through for at he
Kan mad gøre folk glade?Du kommer til at spise det her i din bil, er du ikke? Scott Olson/Getty Images Du kører hjem efter en lang arbejdsdag. Du er ikke i humør til at lave mad, og du stopper ved en drive-through for at he
- Facebook står over for Australiens erstatningskrav for databrud
- Google afsætter 25 millioner dollars til AI-forskning for at hjælpe menneskeheden, jorden
- Sådan fungerer bionedbrydningsadditiver
- Du, også, kan være astrofysiker med dit nye teleskop
- Undersøgelse:Børn fra rigere familier føler mere kontrol over livet
- Sentinel-2B satellit erklæret egnet og klar til idriftsættelse


