Nidovirus udtrykker redundant gener og koder for flere proteiner end tidligere antaget, undersøgelse finder
Kredit:via Georgia State University
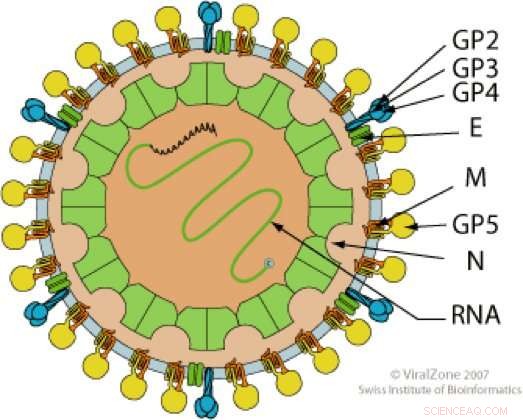
Arterivirus, en familie af enkeltstrengede RNA-vira, der tilhører rækkefølgen Nidovirales, producere flere proteiner og messenger -RNA'er end tidligere rapporteret, et fund, der giver vigtig indsigt om en virus, der potentielt kan udvikle sig til at inficere mennesker i fremtiden, ifølge en ny undersøgelse.
Tidligere har kun ni genom -sekvenser, kendt som transkriptionsregulerende sekvenser (TRS'er), blev rapporteret for arterivirus Simian hæmoragisk febervirus (SHFV), som inficerer aber. Imidlertid, den nye undersøgelse brugte næste generations sekventeringsteknologi og fandt, at 96 TRS'er blev brugt af denne virus til at producere subgenomiske messenger-RNA'er (sg mRNA'er) i både SHFV-inficerede nyreceller og hvide blodlegemer i makak, aber findes primært i Asien. Fire af de tidligere identificerede TRS'er viste sig ikke at være de dominerende, der blev brugt til genekspression.
Resultaterne er offentliggjort i tidsskriftet Procedurer fra National Academy of Sciences .
"Denne virus inficerer i øjeblikket ikke chimpanser eller mennesker, men det er en af de vira, der for nylig er blevet sat på en liste over mulige nye vira, der kan udvikle sig til at inficere chimpanser og/eller mennesker i fremtiden, "sagde Dr. Margo A. Brinton, tilsvarende forfatter til papiret og Regents 'professor i biologi ved Georgia State University. "Ingen forstår, hvad der begrænser værtsområdet for denne virus så præcist. SHFV er i den samme virugruppe som flere vira, der forårsager vigtige landbrugssygdomme og også er relateret til SARS (alvorligt akut respiratorisk syndrom) -virus."
SHFV -infektion påvirker aber af forskellige arter forskelligt. Infektioner hos arter af afrikanske aber er typisk asymptomatiske, men infektioner hos asiatiske makakaber udløser en akut, dødelig hæmoragisk sygdom med dødsfald inden for en til to uger efter infektion. Virussen inficerer makrofager og dendritiske celler, typer hvide blodlegemer, der typisk er væsentlige komponenter i det første værtsforsvar mod en virusinfektion.
Ud over SHFV, arterivirusfamilien omfatter porcin reproduktivt og respiratorisk syndromvirus (PRRSV), som forårsager sygdom hos svin, og Equine arteritis virus (EAV), som forårsager sygdom hos heste.
Nidovirus, den store gruppe af vira, som arterivirus tilhører, også omfatte Coronavirus, Mesonivirus- og Ronivirus -familier. Alle nidovirus har en unik replikationsmekanisme til generering af sg mRNA'er fra højre side af genomet RNA, mens de virale replikative polyproteiner translateres direkte fra venstre side af genomet RNA.
"TRS'er regulerer produktionen af skabeloner til sg mRNA'erne fra genomet RNA, "Brinton sagde." Visdommen var, at der var et primært TRS for hvert strukturgen. Ved hjælp af næste generations sekventering til at opnå en meget dyb analyse af alle de sg mRNA'er, der blev fremstillet i de inficerede celler, vi fandt ud af, at der var flere TRS'er for mange af de strukturelle proteiner (maksimalt 11), og de producerede alle sg mRNA'er. Folk troede, at de få ekstra TRS'er, der tidligere blev fundet, bare var sikkerhedskopier og ikke blev brugt, medmindre den primære blev inaktiveret af mutation, men vores data viser, at de alle altid bruges. "
Forskerne opdagede også TRS'er i det venstre område af genomet, der producerer sg mRNA'er, der giver et alternativt middel for viruset til at forstærke overflod af dets replikative proteiner.
Ud over, de fandt nogle TRS'er, der genererede sg mRNA'er med en anden læseramme end genomet, hvilket betyder, at der produceres hidtil ukendte proteiner. RNA -nukleotidsekvenser læses som trillinger af et translaterende ribosom, med en aminosyre tilsat til det voksende protein pr. trilling. Trillingen kaldes læserammen. Hvis det forskydes med et eller to nukleotider, dette resulterer i en alternativ læseramme, og en anden aminosyresekvens oversættes.
"Dette fund viste, at virussen faktisk kan lave flere proteiner, end man tidligere troede var tilfældet, "Sagde Brinton.
Undersøgelsen opdagede også et antal sg mRNA'er, der kun producerer det terminale fragment af et kendt viralt protein.
"For at teste funktionen af hvert af disse fragmenter, "Sagde Brinton, "vi slog deres produktion ud i inficerede celler en ad gangen ved at mutere hver startkode til translation. Mindre virus blev produceret, da to af disse fragmenter ikke blev produceret, hvilket tyder på, at i det mindste nogle af disse små proteiner er funktionelt vigtige.
"Funktionerne af de nyopdagede virale proteiner er ukendte. Der er stadig meget mere, vi skal lære at forstå, hvordan disse virale proteiner manipulerer den inficerede celle og/eller regulerer viral replikation."
 Varme artikler
Varme artikler
-
 Prag zoo føder sundhed for sjældne malaysiske tigerungerTo seks uger gamle malaysiske tigerunger blev født den 3. oktober, 2017 i zoologisk have i Prag Zoo i Prag fejrede tirsdag sundheden for to sjældne malaysiske tigerunger - en han og en hun - født
Prag zoo føder sundhed for sjældne malaysiske tigerungerTo seks uger gamle malaysiske tigerunger blev født den 3. oktober, 2017 i zoologisk have i Prag Zoo i Prag fejrede tirsdag sundheden for to sjældne malaysiske tigerunger - en han og en hun - født -
 Dovne myrer gør sig nyttige på uventede måderVed at markere myrer med små prikker maling, Daniel Charbonneau og hans samarbejdspartnere var i stand til at holde styr på, hvad individuelle myrer gør i løbet af to eller flere uger. Kredit:Daniel C
Dovne myrer gør sig nyttige på uventede måderVed at markere myrer med små prikker maling, Daniel Charbonneau og hans samarbejdspartnere var i stand til at holde styr på, hvad individuelle myrer gør i løbet af to eller flere uger. Kredit:Daniel C -
 Bekæmpelse af plantesygdomme ved varme temperaturer holder maden på bordetEt spørgsmål af global bekymring er den forventede mangel på landbrugsproduktion for at imødekomme den konstante stigning i den menneskelige befolkning. Forskere fra Michigan State University forstår,
Bekæmpelse af plantesygdomme ved varme temperaturer holder maden på bordetEt spørgsmål af global bekymring er den forventede mangel på landbrugsproduktion for at imødekomme den konstante stigning i den menneskelige befolkning. Forskere fra Michigan State University forstår, -
 Pangolin-handel tvinger Ghana til at se på nye dyrelivslovePangolinen er et føjligt natligt pattedyr, som har en tyk rustning og er også kendt som en skællede myresluger Ghana står over for opfordringer til at opdatere sine love om dyrelivskriminalitet ef
Pangolin-handel tvinger Ghana til at se på nye dyrelivslovePangolinen er et føjligt natligt pattedyr, som har en tyk rustning og er også kendt som en skællede myresluger Ghana står over for opfordringer til at opdatere sine love om dyrelivskriminalitet ef
- Afstemning:Amerikanerne skylder vildt vejr på global opvarmning
- Præcise registreringer af babystjerners vækst fanget ved millimeterbølgelængder
- Når svovl forsvinder sporløst
- Er lavtlønnede job virkelig et springbræt til bedre løn? En ny undersøgelse tyder på, at det ik…
- Ny undersøgelse afslører, at grafener nu går monolag og enkelt krystallinske
- Ligheder og forskelle mellem osmose og diffusion


