Enkeltcelleforskning sætter fokus på rollen som DNA-methylering i beslutninger om celleskæbne
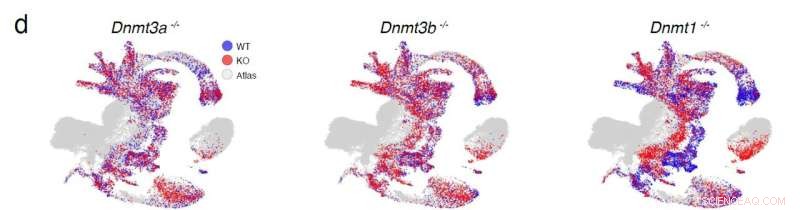
Kortlægning af celler fra DNMT-knock-out-muselinjerne til referenceatlaset (grå) med vildtypeceller angivet med blåt og knock-out-celler angivet med rødt. Kredit:Babraham Institute
Forskning med anvendelse af enkeltcelleanalyseteknikker og sammenligninger med en celleatlasressource gjorde det muligt for forskere ved Babraham Institute at forbinde observerede udviklingsdefekter forårsaget af forstyrrede DNA-methyleringsprocesser med en forståelse af de berørte celletyper. Dette værk, udgivet i Genome Biology , bygger på tidligere arbejde udført med samarbejdspartnere for at etablere et detaljeret celleatlas, der kortlægger celleskæbne gennem tidlig udvikling. Dette arbejde bidrager med vigtig ny viden til at forstå rollen af DNA-methylering under embryogenese, og hjælper med at tyde de regler, der styrer, hvordan forskellige celletyper opstår. En forståelse af disse regler vil være afgørende for, at forskere præcist og sikkert kan styre celleskæbnen til at producere klinisk relevante celletyper til regenerativ medicin.
Forskning fra Reik labat Babraham Institute har fremmet vores forståelse af rollen af DNA-methylering under de tidligere udviklingsstadier. Teknologiske fremskridt på dette område, der giver mulighed for at indsamle parallelle datatyper fra en enkelt celle og eksistensen af celleatlas-referencer og omfattende datasæt, revolutionerer, hvad vi ved om de processer, der bestemmer cellers skæbne. At belyse reglerne for, hvordan forskellige celletyper dannes, har anvendelser i regenerativ medicin såvel som i forståelsen af udviklingsforstyrrelser og sygdom.
Sletningen og genindførelsen af DNA-methylering er kendt for at være afgørende for at etablere celleidentitet, når væv og organer i embryonet dannes. Sletning af vigtige methyleringsenzymer hos mus forårsager i nogle tilfælde alvorlige udviklingsdefekter og embryodødelighed. På trods af vigtigheden af DNA-methylering i udviklingen, er de underliggende mekanismer for, hvordan dette opnås, dårligt forstået. Dette skyldes begrænsninger af den information, forskere tidligere kunne indsamle for at forstå virkningerne af ændringer i de sædvanlige processer af DNA-methylering under udvikling, som var begrænset til analyse af udviklingsdefekter, prøvebilleddannelse og begrænset genom-dækkende analyse ved hjælp af bulkprøver. Disse metoder var ikke tilstrækkelige til at løse virkninger på niveauet af forskellige celletyper.
Ved hjælp af mus, hvor vigtige methyleringsenzymer blev slettet, udførte forskere fra Reik-laboratoriet i Instituttets Epigenetik-program enkeltcelle-genekspressionsanalyse ved starten af organudviklingen, som sker på dag 8.5 efter befrugtning. Ved at udnytte kraften i enkeltcellede tilgange var forskerne i stand til at følge, hvilke celletyper der var påvirket, i form af ikke at være i stand til at dannes i musefosteret, hvilket tyder på mekanismerne bag virkningerne set på en hel-organisme-skala.
Forskningen brugte genetisk modificerede mus, hvor to nøglegrupper af methyleringsenzymer blev slettet:knock-out muselinjer, hvor DNA-methyltransferaser (DNMT 1, 3a og 3b), som introducerer og opretholder DNA-methylering, blev slettet individuelt, og et system til at undersøge virkningerne af en kombineret deletion af alle tre TET-enzymer (ten-elleve translokation (TET) methylcytosindioxygenaser) 1/2/3), som forårsager demethylering.
Dr. Stephen Clark, en seniorforsker i Reik-laboratoriet, da denne forskning blev foretaget, sagde:"Brugen af enkeltcellede tilgange giver virkelig den opløsning, vi har brug for til at studere mekanikken bag DNA-methylering under udvikling. Det billede, vi var i stand til at at opbygge bekræfter den undertrykkende rolle af DNA-methylering på dette udviklingstidspunkt, for det første at opretholdelse af korrekt DNA-methylering er påkrævet for at undertrykke tidligere og alternative celletypeidentiteter, og for det andet at DNA-methylering skal fjernes fra dele af genomet for at tillade visse celler typer til at danne."
En teknik kaldet enkeltcellet RNA-sekventering blev brugt til at måle genekspression på tværs af genomet i hver muselinje. Sammenligning af disse ekspressionsprofiler med et referencedatasæt gjorde det muligt at identificere alle celletyper af embryonet. Efter dette trin kunne effekten af methyleringsforstyrrelser på celleskæbne vurderes ved at sammenligne sammensætningen af knock-out-embryonerne (hvor methyleringsenzymer blev slettet) med vildtype-embryoner på samme udviklingstrin for at fremhæve forskelle i celletype proportioner.
Forskerne var i stand til at korrelere virkninger på celletypedannelse på dag 8.5 af udviklingen, som matchede observerede fænotyper og analysere celletypespecifikke ændringer i genekspression, der kunne være forbundet med defekter i celleskæbneforpligtelse.
Dr. Ricard Argelaguet, en tidligere postdoktor i Reik-laboratoriet ved Instituttet og medførsteforfatter på papiret, sagde:"Evnen til både at have hele organismens perspektiv og granulariteten i at observere ændringer i celletyper og genekspression har givet os muligheden for at skille rollen af DNA-methylering og demethylering i det udviklende embryo på dette bestemte tidspunkt for at skabe ny indsigt. Det vil være lige så interessant at anvende denne tilgang til senere tidspunkter for at forstå mere om rollen af DNA-methylering efterhånden som udviklingen skrider frem."
Forskningen har skabt en interaktiv dataplatform, der giver genekspressionsudlæsninger på enkeltcelleniveau fra Dnmt- og Tet-mutante musembryoner.
Professor Wolf Reik, direktør for Altos Cambridge Institute of Science, som ledede forskningen, mens en gruppeleder i Epigenetics-programmet ved Babraham Institute, sagde:"Denne forskning giver en rig ressource til at undersøge sammenhængen mellem DNA-methylering og etablering af celle skæbne. Denne forskning drage fordel af publicerede datasæt og referenceatlas, og vi håber, at vores arbejde til gengæld er til nytte for andre forskere inden for både udviklings- og epigenetikområdet." + Udforsk yderligere
Ny metode booster studiet af regulering af genaktivitet
 Varme artikler
Varme artikler
-
 Skovnes kulstofoptagelse vil blive kompromitteret af klimaændringer, tyder en undersøgelse af blad…Skovkrone, H.J. Andrews Experimental Forest, Cascade Range, Oregon. Kredit:Theresa Hogue, OSU En ny undersøgelse ledet af Oregon State University tyder på, at blade i skovkroner ikke er i stand til
Skovnes kulstofoptagelse vil blive kompromitteret af klimaændringer, tyder en undersøgelse af blad…Skovkrone, H.J. Andrews Experimental Forest, Cascade Range, Oregon. Kredit:Theresa Hogue, OSU En ny undersøgelse ledet af Oregon State University tyder på, at blade i skovkroner ikke er i stand til -
 Mysteriet,Myte og virkelighed på Plum Island Denne luftfoto af Plum Island Animal Disease Center (PIADC), etableret i 1954 ud for New Yorks kyst, blev taget i oktober, 1971. PIADC blev oprettet af det a
Mysteriet,Myte og virkelighed på Plum Island Denne luftfoto af Plum Island Animal Disease Center (PIADC), etableret i 1954 ud for New Yorks kyst, blev taget i oktober, 1971. PIADC blev oprettet af det a -
 Klimaændringer forkorter den marine fødekæde ud for CalifornienHudceller indsamlet fra almindelige delfiner afslørede skarpe ændringer i det marine fødenet ud for det sydlige Californien. Kredit:Lindsey E. Peavey (NOAA/CINMS) Miljøforstyrrelser som El Niño ry
Klimaændringer forkorter den marine fødekæde ud for CalifornienHudceller indsamlet fra almindelige delfiner afslørede skarpe ændringer i det marine fødenet ud for det sydlige Californien. Kredit:Lindsey E. Peavey (NOAA/CINMS) Miljøforstyrrelser som El Niño ry -
 Batesiansk efterligning:Hvordan Copycats beskytter sig selvEn hornmøl ( Sesia bembeciformis ), ret, efterligner dens udseende, den europæiske hornet ( Vespa crabro ) i et forsøg på at holde rovdyr i skak. Erich Ferdinand/Flickr (CC BY 2.0)/Wikimedia Commo
Batesiansk efterligning:Hvordan Copycats beskytter sig selvEn hornmøl ( Sesia bembeciformis ), ret, efterligner dens udseende, den europæiske hornet ( Vespa crabro ) i et forsøg på at holde rovdyr i skak. Erich Ferdinand/Flickr (CC BY 2.0)/Wikimedia Commo
- 2018 Arktisk sommertid minimumsudbredelse af havis bundet til det sjette laveste i registreringen
- Tilsætning af organisk stof til jorden har en begrænset effekt på vandholdende kapacitet
- Såning af koraller:En ny tilgang baner vejen for storstilet restaurering af koralrev
- Presset til at offentliggøre i topblade kvæler kreativitet inden for økonomisk forskning, undersø…
- Forskere udvikler ny metode til hurtigt, pålideligt overvåge seglcellesygdom
- Forskning peger på kønsforskelle i akademiske præstationer og lederstillinger


