Tavse mutationer hjælper bakterier med at undgå antibiotika
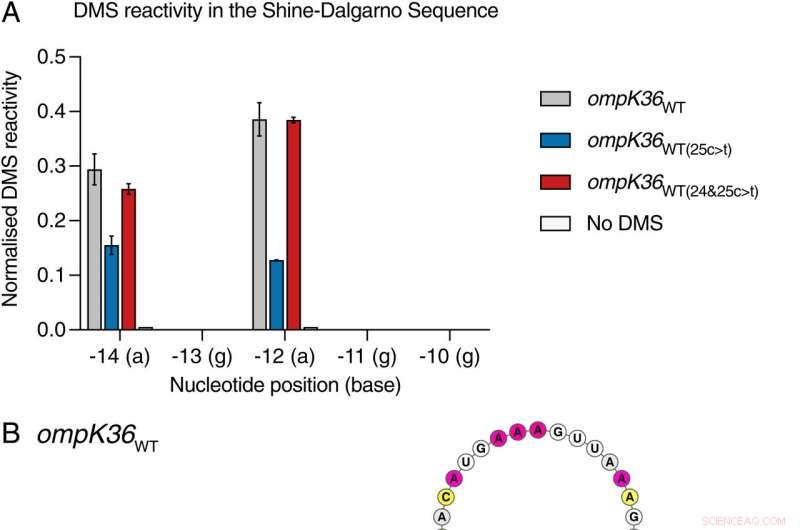
Position 25 i ompK36WT(25c> t) RNA inducerer en stilk, der involverer SDS. (A) Side-by-side normaliseret DMS-signal pr. nukleotid i SDS'et fra fuldlængde in vitro-transskriberet og genfoldet ompK36WT , ompK36WT(25c> t) , ompk36WT(24&25c> t) , og DMS-ubehandlet ompK36WT . Højere værdier svarer til øget basistilgængelighed. DMS-signal (± SD) af 2 biologiske gentagelser for nukleotiderne -14a til -10g er vist. (B-D). DMS-begrænsede strukturmodeller af 5′-enden af ompK36WT (B), ompK36WT(24&25c>t) (C), og ompK36WT(25c> t) (D). Nukleotider er farvet af normaliseret DMS-signal. SDS i RNA-strukturer er fremhævet med gråt. Pile angiver −14-positionen og 25c> t-positionen i ompK36WT(25c> t) . Kredit:Proceedings of the National Academy of Sciences (2022). DOI:10.1073/pnas.2203593119
Forskere har opdaget en ny måde, hvorpå hospitalserhvervede infektioner modstår antibiotika, gennem en "tavs" genetisk mutation.
Bakterier kan opnå resistens over for antibiotika gennem tilfældige mutationer i deres DNA, der giver dem en fordel, der hjælper dem med at overleve. At finde genetiske mutationer og opdage, hvordan de hjælper bakterier med at overleve antibiotikaangreb, er nøglen til at hjælpe os med at kæmpe tilbage med nye lægemidler.
Forskerne har nu opdaget en "tavs" mutation i den genetiske kode, der fører til antibiotikaresistens. Typisk vil mutationer af denne art blive overset, og de kan allerede være til stede i andre smitsomme bakterier.
Holdet, ledet af forskere ved Imperial College London og inklusive internationale samarbejdspartnere, offentliggjorde deres resultater i dag i tidsskriftet Proceedings of the National Academy of Sciences .
Stigende modstand
Forskerne så på bakterien Klebsiella pneumoniae, som forårsager infektioner i lunger, blod og sår hos dem på hospitaler, hvor patienter, der har kompromitteret immunforsvar, såsom dem på intensivafdelinger, er særligt sårbare.
Som mange andre bakterier bliver K. pneumoniae mere og mere resistente over for antibiotika, især en familie af lægemidler kaldet carbapenemer. Disse vigtige lægemidler til sidste udvej bruges på hospitaler, når andre antibiotika allerede har slået fejl.
Da stigende resistens over for carbapenemer dramatisk kan påvirke vores evne til at behandle infektioner, klassificeres carbapenem-resistente K. pneumoniae som "kritiske" World Health Organization Priority 1-organismer.
For at være effektiv skal antibiotika trænge ind i bakterier, og hos K. pneumoniae sker det via en kanal i bakteriens ydre membran, dannet af et protein kaldet OmpK36. Holdet opdagede en genetisk mutation, der får bakterierne til at producere mindre af proteinet, hvilket effektivt lukker nogle af disse kanaler og holder carbapenem-antibiotika ude.
'Tavse' mutationer
Denne mutation virker imidlertid anderledes end standardmutationer, der resulterer i antibiotikaresistens. Normalt ændrer mutationer den genetiske kode, så når den "læses" af ribosomer og omdannes til et protein, producerer den en anden kæde af aminosyrer med forskellige funktioner.
Denne mutation producerer stadig den samme aminosyrekæde, men ændrer strukturen af et vigtigt mRNA-mellemprodukt, hvilket forhindrer ribosomer i at læse koden og producere protein fra den.
Når man leder efter mutationer, søger genomiske teknikker normalt efter ændringer i aminosyresekvensen. Men da denne mutation ændrer en struktur snarere end selve sekvensen, kunne den opfattes som en "tavs" mutation.
Første forfatter Dr. Joshua Wong, fra Institut for Biovidenskab i Imperial, sagde:"I en tidsalder med big data og genomics kan mutationer, som vi har opdaget, blive betragtet som 'tavse', da den genetiske kode resulterer i den samme proteinsekvens .
"Denne opdagelse skulle ændre, hvordan vi ser på den genetiske kode i bakterier og indikerer potentielt, at vi i det videnskabelige samfund har overset andre lignende mutationer, der kan have vigtige virkninger. Vores arbejde fokuserer på en enkelt mutation, men ændrer fundamentalt, hvordan vi fortolker mutationer, især de som man troede var tavse."
Drevet af antibiotikabrug
Holdet hos Imperial, som karakteriserede mutationen, arbejdede med teams ved University of Oxford, University of Florence og Harvard University for at identificere mutationens fordeling globalt, vurdere resistensniveauer og bestemme, hvordan mutationen påvirkede det mellemliggende mRNA struktur.
Ved hjælp af data fra resistente bakterieprøver indsamlet globalt viste holdet, at mutationen var opstået flere gange uafhængigt. Dette tyder på, at det ikke er tilfældigt, og det er i stedet drevet af bakteriernes behov for at forsvare sig selv igen med antibiotika.
Ledende forsker professor Gad Frankel, fra Institut for Biovidenskab ved Imperial, sagde:"Mutationen udviklede sig ved flere lejligheder uafhængigt, og dette fortæller os, at denne nye mekanisme ikke er et engangsindfald, men i stedet drevet af antibiotikaforbrug. tyder på, at mutationen opstår under antibiotikatryk og fremhæver bivirkningerne af overdreven antibiotikaforbrug på hospitaler og andre steder."
Holdet håber nu, at deres fund vil blive inkorporeret i bioinformatiske værktøjer, der analyserer genetiske sekvenser for at identificere tilstedeværelsen af mutationen, som det blev gjort med en tidligere mekanisme, som holdet opdagede.
De vil også fortsætte med at arbejde med deres samarbejdspartnere for at lede efter andre vigtige mutationer i dette nøglepatogen. + Udforsk yderligere
Almindelig lægemiddelresistent superbug udvikler hurtig resistens over for 'sidste udvej'-antibiotikum
 Varme artikler
Varme artikler
-
 Hvordan fungerer muskelsystemet med kredsløbssystemet?Alle kroppens systemer arbejder tæt sammen for at opretholde homeostase - den dynamiske balance mellem indre forhold, som kroppen konstant arbejder for at bevare. Muskelsystemet og kredsløbssystemet h
Hvordan fungerer muskelsystemet med kredsløbssystemet?Alle kroppens systemer arbejder tæt sammen for at opretholde homeostase - den dynamiske balance mellem indre forhold, som kroppen konstant arbejder for at bevare. Muskelsystemet og kredsløbssystemet h -
 Bekymringsvinde:Amerikanske fiskere frygter skove af kraftturbinerI denne mandag, 15. august, 2016, fil foto tre vindmøller fra Deepwater Wind-projektet står i Atlanterhavet ud for Block Island, R.I. Fishermen vender et forsigtigt øje mod en ny opkomling:havvindindu
Bekymringsvinde:Amerikanske fiskere frygter skove af kraftturbinerI denne mandag, 15. august, 2016, fil foto tre vindmøller fra Deepwater Wind-projektet står i Atlanterhavet ud for Block Island, R.I. Fishermen vender et forsigtigt øje mod en ny opkomling:havvindindu -
 Hold giftig cadmium ude af ris på den genetiske mådeI Pokkali optager rødderne på grund af duplikeret OsNramp5 mere Mn og Cd i rodcellerne sammenlignet med andre risvarianter (for eksempel Koshihikari). Det meste Cd optaget gennem OsNramp5 sekvestreres
Hold giftig cadmium ude af ris på den genetiske mådeI Pokkali optager rødderne på grund af duplikeret OsNramp5 mere Mn og Cd i rodcellerne sammenlignet med andre risvarianter (for eksempel Koshihikari). Det meste Cd optaget gennem OsNramp5 sekvestreres -
 Svampene blandt er de store nedbrydereDe mest almindelige forme er mikroskopiske Ascomycetes, eller svampe, der har gengivet sig i det omfang, de kan ses med det blotte menneskelige øje. Jose A. Bernat Bacete/Getty Images Medmindre du er
Svampene blandt er de store nedbrydereDe mest almindelige forme er mikroskopiske Ascomycetes, eller svampe, der har gengivet sig i det omfang, de kan ses med det blotte menneskelige øje. Jose A. Bernat Bacete/Getty Images Medmindre du er
- Yeti Investigation afslører ny information om Himalaya og tibetanske bjørne
- Ny eksotisk stofpartikel, en tetraquark, opdaget
- Fem forskellige typer abiotiske faktorer
- Lidt forbedring i smeltende gletsjer truende Italien resort
- Hvilke tre betingelser er ideelle for bakterier at vokse?
- Robust Bain-forvrængning i premartensitfasen af en platinsubstitueret Ni2MnGa


