Natur vs. laboratorium:Forskellene mellem eksperimentel evolution og naturlig tilpasning

Kredit:CC0 Public Domain
Mennesker har ubevidst udført evolutionsforsøg i årtusinder gennem domesticering af planter, dyr og svampe. Startende med William Dallingers banebrydende eksperimenter i slutningen af det 19. th århundrede er sådanne eksperimenter blevet udført under kontrollerede laboratorieforhold for bedre at forstå evolutionens processer og begrænsninger.
Evolutionære eksperimenter involverer generelt at pålægge en organisme et veldefineret selektivt tryk (såsom ekstrem temperatur, begrænsede næringsstoffer eller tilstedeværelsen af en giftig forbindelse) på en organisme og derefter studere, hvordan den tilpasser sig disse nye forhold. Det længste eksperiment med kontrolleret evolution blev påbegyndt i 1998 af Richard Lenski og fortsætter den dag i dag og involverer over 60.000 generationer af bakterien Escherichia coli.
Mens disse eksperimenter har givet grundlæggende indsigt i evolutionære processer såsom tilpasning, selektion og mutation, er det klart, at naturlig evolution sker under meget mere komplekse begrænsninger. En ny undersøgelse offentliggjort i Genome Biology and Evolution kaster nyt lys over den måde, hvorpå laboratorieudviklingen kan adskille sig fra, hvad der sker i naturen.
Ifølge medforfatter Ruth Hershberg, lektor ved Technion-Israel Institute of Technology, viser deres "resultater, at laboratorietilpasning, som sker som reaktion på ret simple og stærke tryk, ofte kan opstå gennem mutationer, der enten ikke kan forekomme i naturen, eller er meget forbigående, hvis de opstår."
Undersøgelsen, som var medforfatter af Technion Ph.D. studerende Yasmin Cohen, forsøgte at forklare et tilsyneladende paradoks, som forfatterne bemærkede, når de reflekterede over de mutationer, der blev identificeret i deres egne evolutionsforsøg, der involverede bakterier:nemlig at de proteiner, som mutationer oftest forekommer i i laboratoriet, er de samme som dem, der ændrer sig mest. langsomt over lange evolutionære tidsskalaer.
For yderligere at udforske denne observation så Cohen og Hershberg specifikt på to gener, der koder for RNA-polymerase-kerneenzymet (RNAPC), som viste sig at være involveret i tilpasning i mange uafhængige laboratorie-evolutionseksperimenter i E. coli, den art, der oftest anvendes til disse typer af eksperimenter.
Deres litteraturundersøgelse identificerede adaptive mutationer ved 140 aminosyrepositioner på tværs af disse proteiner som reaktion på 12 forskellige laboratoriebetingelser, herunder eksponering for antibiotika, langvarig ressourceudmattelse, vækst ved høje temperaturer og vækst i medier med lavt næringsindhold (minimale). Overraskende nok var der meget lidt overlap i disse adaptive steder, hvor kun fire ud af de 140 optrådte under mere end én betingelse.
Ved at sammenligne disse steder med resten af proteinsekvensen på tværs af bakterieslægter fandt forfatterne desuden, at der ikke kun sker tilpasning i laboratoriet via mutationer til højt konserverede proteiner, men selv inden for RNAPC-proteinerne muterede aminosyrestederne almindeligvis. i laboratorieforsøg havde en tendens til at være mere konserveret i naturen end andre positioner i disse proteiner.
Yderligere analyse identificerede en række spændende mønstre. Positioner, hvor tilpasning fandt sted i laboratorieeksperimenter, havde også en tendens til at falde inden for definerede proteinfunktionelle domæner, at klynge sig tæt på hinanden på proteinstrukturen og at være placeret tæt på det aktive RNAPC-sted oftere end andre steder.
For at se, om lignende dynamik var på spil for andre proteiner, så Cohen og Hershberg på 19 andre proteiner, der indeholdt adaptive mutationer forbundet med ressourceudmattelse. De fandt ud af, at som med RNAPC-proteinerne, havde steder forbundet med tilpasning i laboratorieforsøg en tendens til at være mere konserverede blandt bakterier.
Endnu mere interessant, når man ser på de fire selektive tryk, for hvilke der var tilstrækkelige data, holdt disse mønstre for antibiotikaeksponering, minimale medier og langvarig ressourceudmattelse, men ikke for vækst ved høje temperaturer. Tilpasninger til høje temperaturer udviser således ikke højere bevaring, er ikke klynget tæt på hinanden eller kompleksets aktive sted og beriges ikke inden for funktionelle domæner.
Som Hershberg bemærker, er det uklart, hvor almindeligt dette fund er. "Vi kan i øjeblikket ikke være sikre på, om tilpasninger til de fleste forhold opfører sig som de fleste karakteriserede tilpasninger, hvor høj temperatur er en afviger, eller om der er mange forhold uden tilgængelige data, som mere ligner det, der ses for høj temperatur."
Det, der er klart, er, at dynamikken i laboratorietilpasning adskiller sig meget fra den naturlige tilpasning. Dette skyldes, som forfatterne forklarer, "i laboratorieeksperimenter er bakterier generelt udsat for relativt enkle, stærke og konstante selektive tryk. De selektive pres, der udsættes for i mere naturlige miljøer, er sandsynligvis langt mere komplekse, med flere forskellige faktorer, der udøver modstridende pres. samtidig og/eller med selektive tryk, der ændrer sig med tiden. Tilpasninger af den slags, der opstår så let under laboratorieudvikling, er måske ikke så let tilladt i naturlige miljøer... Desuden, hvis sådanne tilpasninger sker som reaktion på et specifikt sæt af forhold, vil de kan vise sig at være meget forbigående, hurtigt faldende i frekvens, når betingelserne ændrer sig."
For at udforske disse spørgsmål yderligere, mener Hershberg, at det vil være "vigtigt at forsøge at finde ud af, hvad disse tilpasninger gør i den kontekst, hvor de er adaptive, og at måle deres fitnesseffekter under forskellige forhold... Fokus på RNAPC enzymtilpasninger kunne være et nyttigt sted at starte." Det er vigtigt, at sådanne undersøgelser kan give ny indsigt i de mekanismer, hvorved evolution sker, både i laboratoriet og i naturen. Ifølge Hershberg, "at forstå årsagerne til disse forskelle kan gøre det muligt for os at lære vigtige lektioner om naturlig tilpasning." + Udforsk yderligere
En overflod af gavnlige mutationer
 Varme artikler
Varme artikler
-
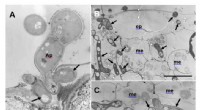 Afvigende hyfer udløst af værtens immunrespons på plantepatogen svampFigur 1:Transmissionselektronmikroskopi (TEM) billeder af modtagelige R108-1 blade 3 dage efter inokulering med M. pinodes. (A) Infektionshyfer (pile), der ligner infektionsvesikler dannet i cellevægg
Afvigende hyfer udløst af værtens immunrespons på plantepatogen svampFigur 1:Transmissionselektronmikroskopi (TEM) billeder af modtagelige R108-1 blade 3 dage efter inokulering med M. pinodes. (A) Infektionshyfer (pile), der ligner infektionsvesikler dannet i cellevægg -
 Afdækning af, hvad genomik kan gøreNy viden om væggelusens genom vil føre til nye bekæmpelsesmetoder. Kredit:Stephen Ausmus Det tog næsten 10, 000 års avl for at tage majs fra en tropisk afgrøde med tommelfingerstore ører til den h
Afdækning af, hvad genomik kan gøreNy viden om væggelusens genom vil føre til nye bekæmpelsesmetoder. Kredit:Stephen Ausmus Det tog næsten 10, 000 års avl for at tage majs fra en tropisk afgrøde med tommelfingerstore ører til den h -
 Earth Microbiome Project:Kortlægning af mikrobiomet af... altingEarth Microbiome Project-samarbejdspartnere indsamler og analyserer prøver fra forskellige miljøer rundt om i verden. Øverst til venstre:Vandretur gennem regnskoven i Puerto Rico for at prøve jord med
Earth Microbiome Project:Kortlægning af mikrobiomet af... altingEarth Microbiome Project-samarbejdspartnere indsamler og analyserer prøver fra forskellige miljøer rundt om i verden. Øverst til venstre:Vandretur gennem regnskoven i Puerto Rico for at prøve jord med -
 Der er en nem måde at forstå mitose og meiose påMitose er opdelingen af en enkelt celle i to celler (som vist her), hver med sin egen kerne og samme antal og slags kromosomer som forældercellen. Meiose opstår, når en enkelt celle deler sig to gan
Der er en nem måde at forstå mitose og meiose påMitose er opdelingen af en enkelt celle i to celler (som vist her), hver med sin egen kerne og samme antal og slags kromosomer som forældercellen. Meiose opstår, når en enkelt celle deler sig to gan
- Doping af atleter kan blive sværere at skjule med ny detektionsmetode
- Sådan styrker man demokratiet ifølge en ny megaundersøgelse
- Forskere skaber en 3D-printet smart gel, der går under vandet, flytter genstande
- Holder astronauter – og Jorden – sikre mod ødelæggende solstorme
- San Francisco kvæler giftig luft, mens skovbrande raser
- Project Sidewalk hjælper brugere med at kortlægge tilgængelighed omkring Seattle og andre byer


