Undersøgelse udvikler en ny måde at identificere kræftceller på
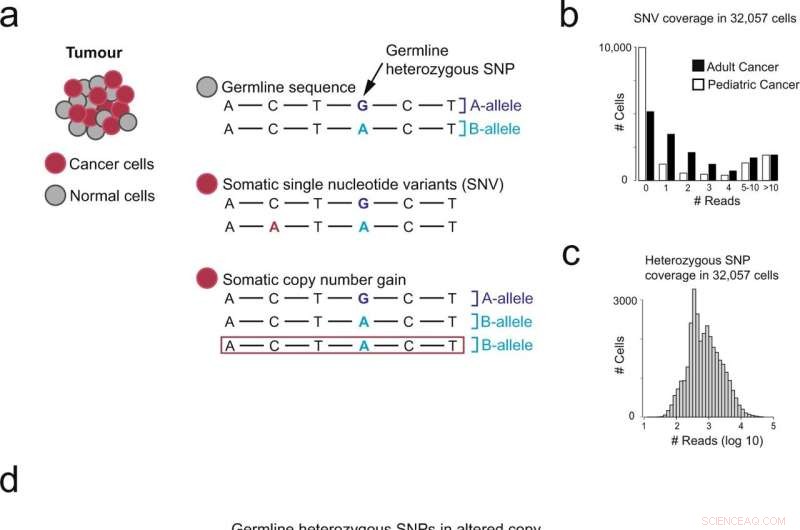
Oversigt over forskellige tilgange til at identificere cancer-afledte celler. a Genomiske ændringer til stede i kræftgenomer. b Antal celler (y-akse) med N-aflæsninger, der dækker punktmutationer (x-akse), adskilt af lav (NB neuroblastom) og høj (RCC nyrecellecarcinom) mutationsbyrde. c Antal celler (y-aksen) med N-aflæsninger, der dækker heterozygote enkeltnukleotidpolymorfier (SNP) (x-aksen). d Oversigt over brug af allelforskydninger, der repræsenterer kopinummerændringer til at påvise cancerceller. Kredit:Kommunikationsbiologi (2022). DOI:10.1038/s42003-022-03808-9. https://www.nature.com/articles/s42003-022-03808-9
En ny metode til at adskille kræftceller fra ikke-kræftceller er blevet udviklet af forskere ved Wellcome Sanger Institute, som et løft for dem, der arbejder på bedre at forstå kræftbiologi ved hjælp af enkeltcellet mRNA-sekventering.
Undersøgelsen, offentliggjort i dag i Communications Biology , forbedrer eksisterende metoder til at identificere, hvilke celler i en prøve der er kræftfremkaldende og giver vigtige data om tumorers mikromiljø. Softwaren er åbent tilgængelig for forskere over hele verden til at anvende på deres egne data, hvilket fremmer effektiviteten af enkeltcellesekvensering for at forstå kræft.
Enkeltcelle-mRNA-analyse af kræftceller er en af de førende teknikker, der bruges til bedre at forstå kræftbiologi. De genererede data kan bruges til at forsøge at forstyrre kræftsygdomme med medicin eller finde ud af, hvordan kræftformer opstår i første omgang.
Et grundlæggende trin i denne proces er at adskille kræftceller og ikke-kræftceller, men det er ikke altid en nem opgave. Ud over de mange kræfttyper vil der også være molekylære forskelle mellem kræftceller af samme type inden for en enkelt tumor.
I øjeblikket er den bedste metode til at gøre dette at måle den gennemsnitlige genekspression af celler i prøven, med højere eller lavere ekspression, der bruges til at skelne kræftceller fra raske celler. Men denne metode kan føre til falske konklusioner.
I denne nye undersøgelse udførte forskere ved Wellcome Sanger Institute helgenomsekventering og enkeltcellet mRNA-sekventering på prøver indsamlet af Great Ormond Street Hospital (GOSH).
Ved at lokalisere ubalancer af alleler i disse data, som indikerer kopiantal ændringer i genomet, var holdet i stand til at identificere kræftceller mere pålideligt end med tidligere metoder. Denne tilgang vil primært være nyttig til at validere nye cancercelletyper og bedre forståelse af mikromiljøet i tumorvæv.
"At være i stand til at vide, hvordan transkriptomet er anderledes i celler med afvigende genomer, såsom dem, der findes i kræftformer, er værdifuld viden og vil udvide de spørgsmål, som vi kan besvare ved hjælp af enkeltcelle-sekventering," siger Dr. Matt Young.
Metoden, kaldet alleleIntegrator, er tilgængelig som en softwarepakke, som forskere over hele verden kan bruge. + Udforsk yderligere
Estimering af tumorspecifikt totalt mRNA-niveau forudsiger cancerudfald
 Varme artikler
Varme artikler
-
 Er mennesker uden visdomstænder mere højt udviklede?Bare rolig, dette vil gøre en stor anekdote en dag. Se flere personlige hygiejnebilleder. Stock4B/Getty Images Hvis du nogensinde sidder fast på en fest uden noget at tale om, du nævner måske, at du
Er mennesker uden visdomstænder mere højt udviklede?Bare rolig, dette vil gøre en stor anekdote en dag. Se flere personlige hygiejnebilleder. Stock4B/Getty Images Hvis du nogensinde sidder fast på en fest uden noget at tale om, du nævner måske, at du -
 Ligheder og forskelle mellem osmose og diffusionOsmose og diffusion spiller væsentlige, men forskellige roller i den menneskelige krop. Diffusion ser molekyler i et område med høj koncentration bevæge sig til områder med en lavere koncentration,
Ligheder og forskelle mellem osmose og diffusionOsmose og diffusion spiller væsentlige, men forskellige roller i den menneskelige krop. Diffusion ser molekyler i et område med høj koncentration bevæge sig til områder med en lavere koncentration, -
 Struktur af primært optogenetisk værktøj afsløretForsøg på at løse strukturen af ChR2 går helt tilbage til tidspunktet for dets opdagelse i 2003. Men på trods af indsatsen fra adskillige forskningsgrupper fra hele verden, strukturen af proteinet
Struktur af primært optogenetisk værktøj afsløretForsøg på at løse strukturen af ChR2 går helt tilbage til tidspunktet for dets opdagelse i 2003. Men på trods af indsatsen fra adskillige forskningsgrupper fra hele verden, strukturen af proteinet -
 Interne kræfter, der styrer cellemigration, afsløres ved levende cellemikroskopiOrientering af integriner i forkant af en migrerende Jurkat T -celle, analyseret ved hjælp af et fluorescenspolariseret lysmikroskop (øjeblikkeligt FluoPolScope) udviklet på Marine Biological Laborato
Interne kræfter, der styrer cellemigration, afsløres ved levende cellemikroskopiOrientering af integriner i forkant af en migrerende Jurkat T -celle, analyseret ved hjælp af et fluorescenspolariseret lysmikroskop (øjeblikkeligt FluoPolScope) udviklet på Marine Biological Laborato
- Det Hvide Hus sigter mod at reducere satellitrod i rummet
- Landsat 9 instrument klar til rumfartøjssamling
- Mindst 40 dræbt i alvorlige oversvømmelser i det nordlige Tyrkiet
- Forskere rapporterer haptisk interface til mobil teknologi
- Mikrobielle samfund har en sæsonbestemt rystelse
- Efter det historiske kvarter, Tesla ser mod Europa, Kina


