AI kan hjælpe forskere med at forstå, hvad vira er i gang med i havene og i din tarm

Virus er en mystisk og dårligt forstået kraft i mikrobielle økosystemer. Forskere ved, at de kan inficere, dræbe og manipulere menneskelige og bakterielle celler i næsten alle miljøer, fra havene til din tarm. Men forskerne har endnu ikke et fuldstændigt billede af, hvordan vira påvirker deres omgivende miljøer i vid udstrækning på grund af deres ekstraordinære mangfoldighed og evne til hurtigt at udvikle sig.
Samfund af mikrober er vanskelige at studere i laboratoriemiljøer. Mange mikrober er udfordrende at dyrke, og deres naturlige miljø har mange flere funktioner, der påvirker deres succes eller fiasko, end forskere kan kopiere i et laboratorium.
Så systembiologer som mig sekventerer ofte alt det DNA, der er til stede i en prøve - for eksempel en fækal prøve fra en patient - udskiller de virale DNA-sekvenser og anmærker derefter de dele af det virale genom, der koder for proteiner. Disse noter om geners placering, struktur og andre egenskaber hjælper forskere med at forstå de funktioner, vira kan udføre i miljøet, og hjælper med at identificere forskellige typer vira. Forskere annoterer vira ved at matche virale sekvenser i en prøve med tidligere annoterede sekvenser, der er tilgængelige i offentlige databaser over virale genetiske sekvenser.
Forskere identificerer dog virale sekvenser i DNA indsamlet fra miljøet i en hastighed, der langt overgår vores evne til at kommentere disse gener. Det betyder, at forskere offentliggør resultater om vira i mikrobielle økosystemer ved hjælp af uacceptabelt små fraktioner af tilgængelige data.
For at forbedre forskernes evne til at studere vira over hele kloden har mit team og jeg udviklet en ny tilgang til at annotere virale sekvenser ved hjælp af kunstig intelligens. Gennem proteinsprogmodeller beslægtet med store sprogmodeller som ChatGPT, men specifikke for proteiner, var vi i stand til at klassificere tidligere usete virale sekvenser. Dette åbner døren for, at forskere ikke kun kan lære mere om vira, men også til at tage fat på biologiske spørgsmål, som er svære at besvare med nuværende teknikker.
Annotering af vira med AI
Store sprogmodeller bruger relationer mellem ord i store datasæt af tekst til at give potentielle svar på spørgsmål, de ikke eksplicit er "lært" svaret på. Når du spørger en chatbot "Hvad er hovedstaden i Frankrig?" for eksempel søger modellen ikke svaret i en tabel over hovedstæder. Det bruger snarere sin træning på enorme datasæt af dokumenter og informationer til at udlede svaret:"Frankrigs hovedstad er Paris."
På samme måde er proteinsprogmodeller AI-algoritmer, der er trænet til at genkende relationer mellem milliarder af proteinsekvenser fra miljøer rundt om i verden. Gennem denne træning kan de muligvis udlede noget om essensen af virale proteiner og deres funktioner.
Vi spekulerede på, om proteinsprogsmodeller kunne besvare dette spørgsmål:"Givet alle annoterede virale genetiske sekvenser, hvad er denne nye sekvenss funktion?"
I vores proof of concept trænede vi neurale netværk på tidligere annoterede virale proteinsekvenser i præ-trænede proteinsprogmodeller og brugte dem derefter til at forudsige annoteringen af nye virale proteinsekvenser. Vores tilgang giver os mulighed for at undersøge, hvad modellen "ser" i en bestemt viral sekvens, der fører til en bestemt annotering. Dette hjælper med at identificere kandidatproteiner af interesse, enten baseret på deres specifikke funktioner eller hvordan deres genom er arrangeret, og vinder ned i søgerummet i enorme datasæt.
Ved at identificere mere fjernt beslægtede virale genfunktioner kan proteinsprogsmodeller komplementere nuværende metoder til at give ny indsigt i mikrobiologi. For eksempel var mit team og jeg i stand til at bruge vores model til at opdage en hidtil ukendt integrase - en type protein, der kan flytte genetisk information ind og ud af celler - i de globalt rigelige marine picocyanobakterier Prochlorococcus og Synechococcus. Især kan denne integrase muligvis flytte gener ind og ud af disse populationer af bakterier i havene og sætte disse mikrober i stand til bedre at tilpasse sig skiftende miljøer.
Vores sprogmodel identificerede også et nyt viralt capsidprotein, der er udbredt i de globale oceaner. Vi producerede det første billede af, hvordan dets gener er arrangeret, hvilket viser, at det kan indeholde forskellige sæt gener, som vi mener indikerer, at denne virus tjener forskellige funktioner i sit miljø.
Disse foreløbige resultater repræsenterer kun to af tusindvis af annoteringer, som vores tilgang har givet.
Analyse af det ukendte
De fleste af de hundredtusindvis af nyopdagede vira forbliver uklassificerede. Mange virale genetiske sekvenser matcher proteinfamilier uden kendt funktion eller er aldrig set før. Vores arbejde viser, at lignende proteinsprogsmodeller kan hjælpe med at studere truslen og løftet om vores planets mange ukarakteriserede vira.
Mens vores undersøgelse fokuserede på vira i de globale oceaner, er forbedret annotering af virale proteiner afgørende for bedre at forstå den rolle, vira spiller i sundhed og sygdom i den menneskelige krop. Vi og andre forskere har antaget, at viral aktivitet i det menneskelige tarmmikrobiom kan blive ændret, når du er syg. Det betyder, at vira kan hjælpe med at identificere stress i mikrobielle samfund.
Vores tilgang er dog også begrænset, fordi den kræver annoteringer af høj kvalitet. Forskere er ved at udvikle nyere proteinsprogmodeller, der inkorporerer andre "opgaver" som en del af deres træning, især forudsige proteinstrukturer for at opdage lignende proteiner, for at gøre dem mere kraftfulde.
At gøre alle AI-værktøjer tilgængelige via FAIR Data Principles – data, der er tilgængelige, tilgængelige, interoperable og genbrugelige – kan hjælpe forskere som helhed med at indse potentialet i disse nye måder at annotere proteinsekvenser på, hvilket fører til opdagelser, der gavner menneskers sundhed.
Leveret af The Conversation
Denne artikel er genudgivet fra The Conversation under en Creative Commons-licens. Læs den originale artikel. 
 Varme artikler
Varme artikler
-
 Sådan behandles Poison SumacGift sumac ( Toxicodendron vernix ) har typisk mellem syv og 13 blade, alle arrangeret i par, og sport små cremefarvede eller gule bær. Joshua Mayer/Flicker (CC By-SA 2.0) Poison ivy ser ud til at
Sådan behandles Poison SumacGift sumac ( Toxicodendron vernix ) har typisk mellem syv og 13 blade, alle arrangeret i par, og sport små cremefarvede eller gule bær. Joshua Mayer/Flicker (CC By-SA 2.0) Poison ivy ser ud til at -
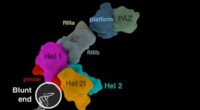 Mød de små maskiner i celler, der massakrerer viraNår vira inficerer kroppens celler, disse celler står over for et vanskeligt problem. Hvordan kan de ødelægge vira uden at skade sig selv? Forskere ved University of Utah Health har fundet et svar ved
Mød de små maskiner i celler, der massakrerer viraNår vira inficerer kroppens celler, disse celler står over for et vanskeligt problem. Hvordan kan de ødelægge vira uden at skade sig selv? Forskere ved University of Utah Health har fundet et svar ved -
 Disse spidser har hoveder, der krymper med sæsonenFoto af den almindelige spidsmus Sorex araneus . Kredit:Karol Zub Hvis en del af kroppen ser ud til at være dårligt udstyret til at krympe, det ville sandsynligvis være hoved og kranium. Og, end
Disse spidser har hoveder, der krymper med sæsonenFoto af den almindelige spidsmus Sorex araneus . Kredit:Karol Zub Hvis en del af kroppen ser ud til at være dårligt udstyret til at krympe, det ville sandsynligvis være hoved og kranium. Og, end -
 Antidepressiva fra urin gør fisk mindre bange for rovdyrKredit:Freshscience Antidepressiva er på vej ind i vores søer og floder - og de gør ferskvandsfisk mindre forvirret af rovdyr. Jake Martin, en ph.d. -kandidat ved Monash University, kiggede på, h
Antidepressiva fra urin gør fisk mindre bange for rovdyrKredit:Freshscience Antidepressiva er på vej ind i vores søer og floder - og de gør ferskvandsfisk mindre forvirret af rovdyr. Jake Martin, en ph.d. -kandidat ved Monash University, kiggede på, h
- At inddrive tabt løn er næsten umuligt for Australiens underbetalte migrantarbejdere. Sådan løse…
- Giver det at slå nummer to i T20 verdensmesterskab i cricket en afgørende fordel? En statistikprof…
- Google Assistant vil snart være på næsten 1 milliard enheder, siger selskabet på CES 2019
- Hvor lang tid tager det at komme til Mars?
- Sojamikroperler er miljøvenlige alternativer til plastmikroperler, der bruges i kosmetik, sæbe pro…
- Verdens tørst efter olie vokser, med SUV'er en vigtig synder


