Sweetpotato genetik:En omfattende opdatering til Taizhong 6-genomannoteringen
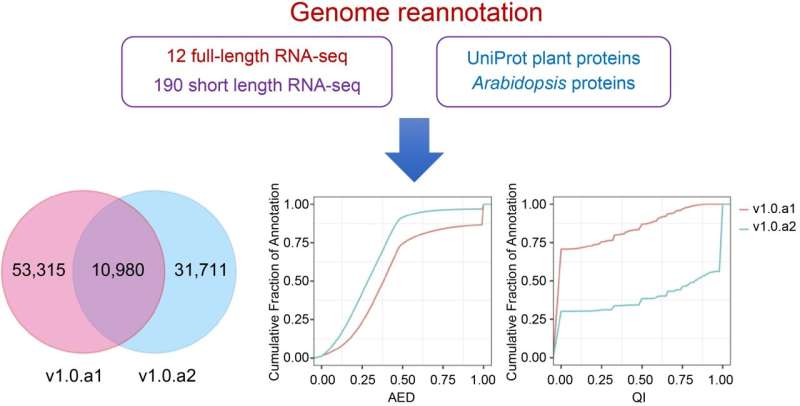
Et forskerhold har forbedret annoteringen af sødkartoffelgenomet "Taizhong 6" væsentligt, og introducerer en mere omfattende og detaljeret version, v1.0.a2. Denne opdatering bruger 12 Nanopore RNA-biblioteker i fuld længde og 190 Illumina RNA-seq-biblioteker, hvilket resulterer i identifikation af 360 nye gener, modifikation eller tilføjelse af 31.771 genmodeller og raffineret gennomenklatur.
Den forbedrede annotering inkluderer detaljerede miRNA-ekspressionsprofiler, som gavner genfunktionelle undersøgelser i sødkartoffel og avancerede genomiske analyser på tværs af Convolvulaceae-familien, hvilket giver en kritisk ressource for fremtidig landbrugs- og genetisk forskning.
Sødkartoffel (Ipomoea batatas) er en vital afgrøde i udviklingslande, der giver essentielle næringsstoffer og bekæmper A-vitaminmangel i Afrika som en hexaploid art. Den første genomsekvens, cultivar Taizhong 6, brugte Illumina-sekventering, senere forbedret med tredje generations sekventeringsteknologier, hvilket førte til en kromosomskaleret genomsamling af høj kvalitet. Der er dog stadig udfordringer med at opnå præcise genomannoteringer på grund af begrænsningerne ved kortlæst sekventering.
For nylig har fremskridt inden for langlæst sekventering, som Oxford Nanopore Technologies, lettet mere nøjagtige annoteringer og givet dybere indsigt i genstruktur og alternativ splejsning.
En undersøgelse offentliggjort i Tropical Plants den 21. marts 2024, bygger på dette fremskridt ved hjælp af en raffineret annotationspipeline, der inkorporerede både Nanopore fuldlængde og omfattende RNA-seq-datasæt, hvilket forbedrer den nuværende genom-annotering for sweetpotato.
I denne undersøgelse forbedrede forskere annoteringen af I. batata-genomet til version 1.0.a2 ved at bruge en omfattende tilgang, der integrerer Nanopore fuldlængde transkriptomer og Illumina RNA-seq-data på tværs af forskellige udviklingsstadier og væv af sød kartoffel.
Deres metode brugte BRAKER til indledende genforudsigelser, beriget med forskellige genomiske hints, efterfulgt af generering af konsensusmodel gennem EVidenceModeler (EVM). Navnlig indeholder den opdaterede annotation nu 42.751 proteinkodende gener, hvilket forbedrer modellen med 3' og 5' UTR'er og øger det gennemsnitlige exonantal pr. gen.
Det er væsentligt, at denne revision tilføjede eller modificerede 31.771 genmodeller, der inkorporerede 8.736 alternative splejsningsisoformer, og introducerede en ny gennomenklatur til klarere reference. Denne mere detaljerede annotering hjælper med præcise genomiske undersøgelser og understøtter avanceret funktionel genomik i søde kartofler.
Desuden giver integration af miRNA-data og deres mål ny indsigt i genregulering, især under forskellige udviklingsstadier af lagerrødder, hvilket forbedrer vores forståelse af sødkartoffelbiologi og hjælper med målrettede avlsindsatser. De omfattende genfunktionsforudsigelser blev udført ved hjælp af InterProScan og eggNOG-mapper, hvilket giver en rigere annotation, der er afgørende for igangværende forsknings- og avlsprogrammer fokuseret på at forbedre sødkartoffelkultivarer til globalt landbrug.
Ifølge undersøgelsens ledende forsker, prof. Guopeng Zhu, "Vores undersøgelse bidrager til en opdateret genomannotering for sødkartoffelgenomet, som væsentligt vil lette genfunktionelle undersøgelser i sødkartoffel og fremme genomiske analyser på tværs af Convolvulaceae-familien."
Samlet set letter denne forbedrede genomiske ramme en dybere funktionel genomik i søde kartofler og understøtter avancerede avlsprogrammer ved at integrere detaljerede miRNA-data og genfunktionsforudsigelser for at forbedre kultivaregenskaber.
Flere oplysninger: Bei Liang et al., Genom reannotation af søde kartofler (Ipomoea batatas (L.) Lam.) ved hjælp af omfattende Nanopore og Illumina-baserede RNA-seq datasæt, Tropical Plants (2024). DOI:10.48130/tp-0024-0009
Leveret af Chinese Academy of Sciences
 Varme artikler
Varme artikler
-
 Forskere afslører, hvordan et vigtigt kræftmål kunne standse kræftspredningNuklear fascin bidrager til F-aktin bundtning. (A) Repræsentativt western blot af fascin knockdown (KD) HeLa celler, der udtrykker specificerede GFP-fascin konstruktioner udsat for biokemisk fraktione
Forskere afslører, hvordan et vigtigt kræftmål kunne standse kræftspredningNuklear fascin bidrager til F-aktin bundtning. (A) Repræsentativt western blot af fascin knockdown (KD) HeLa celler, der udtrykker specificerede GFP-fascin konstruktioner udsat for biokemisk fraktione -
 Skal vi banke gener for ekstraordinære mennesker til kloning?Vores gener giver en delvis blueprint for, hvem vi er. Skal vi redde generne fra store lærde, kunstnere, ledere og atleter til fremtidig kloning? Tim Flach/Stone/Getty Images Fiktive karakterer har f
Skal vi banke gener for ekstraordinære mennesker til kloning?Vores gener giver en delvis blueprint for, hvem vi er. Skal vi redde generne fra store lærde, kunstnere, ledere og atleter til fremtidig kloning? Tim Flach/Stone/Getty Images Fiktive karakterer har f -
 Fiskenes morgengry:Tidlige silurkæbede hvirveldyr afslørede hoved til haleLivsrekonstruktion af Xiushanosteus mirabilis. Kredit:ZHANG Heming En nyligt opdaget fossil skattehob, der går tilbage omkring 436 millioner år tilbage til den tidlige siluriske periode, afslører f
Fiskenes morgengry:Tidlige silurkæbede hvirveldyr afslørede hoved til haleLivsrekonstruktion af Xiushanosteus mirabilis. Kredit:ZHANG Heming En nyligt opdaget fossil skattehob, der går tilbage omkring 436 millioner år tilbage til den tidlige siluriske periode, afslører f -
 Ældste opdagelse til dato af fysogastriske insekterFossile snørevingelarver med forstørret mave. Kredit:J. Haug Fanget i træharpiks og bevaret som i en tidskapsel:fossiler indesluttet i rav giver detaljeret indblik i anatomien af lange uddøde art
Ældste opdagelse til dato af fysogastriske insekterFossile snørevingelarver med forstørret mave. Kredit:J. Haug Fanget i træharpiks og bevaret som i en tidskapsel:fossiler indesluttet i rav giver detaljeret indblik i anatomien af lange uddøde art
- Groupie-galakser, der kredser om Mælkevejen, fortæller os om mørkt stof, hvordan galaksen blev da…
- Et afgørende enzym blev endelig afsløret
- Astronomer opdager to lyse højrøde forskydningskvasarer
- Hvordan storskala enkeltcelle-genomik komplementerer metagenomiske undersøgelser
- EU's husholdninger spilder over 17 milliarder kg frisk frugt og grøntsager om året
- En ny æra af dinosaurer


