Minimalistiske maskinlæringsalgoritmer analyserer billeder fra meget få data
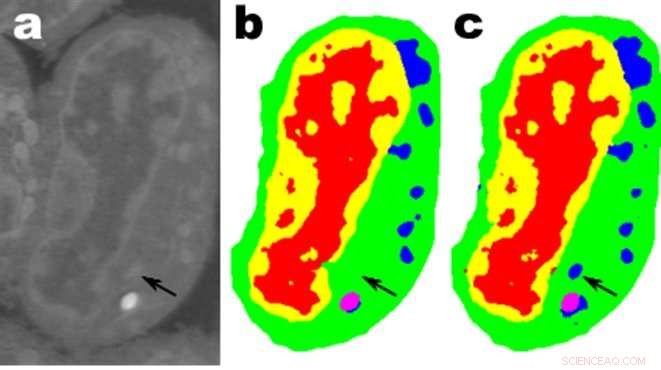
Billeder af et udsnit af muselymfblastoidceller; en. er rådata, b er den tilsvarende manuelle segmentering og c er output fra et MS-D-netværk med 100 lag. Kredit:Data fra A. Ekman og C. Larabell, Nationalt center for røntgentomografi.
Matematikere ved Department of Energy's Lawrence Berkeley National Laboratory (Berkeley Lab) har udviklet en ny tilgang til maskinlæring rettet mod eksperimentelle billeddannelsesdata. I stedet for at stole på de titusinder eller hundredtusinder af billeder, der bruges ved typiske maskinlæringsmetoder, denne nye tilgang "lærer" meget hurtigere og kræver langt færre billeder.
Daniël Pelt og James Sethian fra Berkeley Labs Center for Advanced Mathematics for Energy Research Applications (CAMERA) vendte det sædvanlige maskinlæringsperspektiv på hovedet ved at udvikle det, de kalder et "Mixed-Scale Dense Convolution Neural Network (MS-D)", der kræver langt færre parametre end traditionelle metoder, konvergerer hurtigt, og har evnen til at "lære" fra et bemærkelsesværdigt lille træningssæt. Deres tilgang bruges allerede til at udtrække biologisk struktur fra cellebilleder, og er klar til at levere et stort nyt beregningsværktøj til at analysere data på tværs af en lang række forskningsområder.
Da eksperimentelle faciliteter genererer billeder med højere opløsning ved højere hastigheder, forskere kan kæmpe med at styre og analysere de resulterende data, som ofte udføres omhyggeligt i hånden. I 2014, Sethian etablerede CAMERA på Berkeley Lab som en integreret, tværfagligt center for at udvikle og levere grundlæggende ny matematik, der kræves for at udnytte eksperimentelle undersøgelser på DOE Office of Science-brugerfaciliteter. CAMERA er en del af laboratoriets afdeling for beregning af forskning.
"I mange videnskabelige anvendelser, enormt manuelt arbejde er påkrævet for at kommentere og mærke billeder - det kan tage uger at producere en håndfuld omhyggeligt afgrænsede billeder, "sagde Sethian, som også er matematikprofessor ved University of California, Berkeley. "Vores mål var at udvikle en teknik, der lærer af et meget lille datasæt."
Detaljer om algoritmen blev offentliggjort 26. december, 2017 i et papir i Procedurer fra National Academy of Sciences .
"Gennembruddet skyldtes at indse, at den sædvanlige ned- og opskalering, der fanger funktioner på forskellige billedskalaer, kunne erstattes af matematiske konvolutions, der håndterer flere skalaer i et enkelt lag, sagde Pelt, som også er medlem af Computational Imaging Group på Centrum Wiskunde &Informatica, det nationale forskningsinstitut for matematik og datalogi i Holland.
For at gøre algoritmen tilgængelig for et bredt sæt forskere, et Berkeley -team ledet af Olivia Jain og Simon Mo byggede en webportal "Segmenting Labeled Image Data Engine (SlideCAM)" som en del af CAMERA -pakken med værktøjer til DOE -eksperimentelle faciliteter.

Tomografiske billeder af en fiberforstærket minikomposit, rekonstrueret ved hjælp af 1024 fremskrivninger (a) og 120 fremskrivninger (b). I (c), output fra et MS-D-netværk med billede (b) som input vises. Et lille område angivet med en rød firkant vises forstørret i nederste højre hjørne af hvert billede. Kredit:Daniël Pelt og James Sethian, Berkeley Lab
En lovende anvendelse er at forstå den indre struktur af biologiske celler og et projekt, hvor Pelts og Sethians MS-D-metode kun behøvede data fra syv celler for at bestemme cellestrukturen.
"I vores laboratorium, vi arbejder på at forstå, hvordan cellestruktur og morfologi påvirker eller styrer celleadfærd. Vi bruger utallige timer på at håndsegmentere celler for at udtrække struktur, og identificere, for eksempel, forskelle mellem raske vs syge celler, "sagde Carolyn Larabell, Direktør for National Center for X-ray Tomography og professor ved University of California San Francisco School of Medicine. "Denne nye tilgang har potentiale til radikalt at ændre vores evne til at forstå sygdom, og er et centralt værktøj i vores nye Chan-Zuckerberg-sponsorerede projekt til etablering af et menneskeligt celleatlas, et globalt samarbejde om at kortlægge og karakterisere alle celler i en sund menneskekrop. "
Få mere videnskab fra færre data
Billeder er overalt. Smart telefoner og sensorer har produceret en skattekiste af billeder, mange mærket med relevant information, der identificerer indhold. Ved hjælp af denne enorme database med krydsrefererede billeder, konvolutionelle neurale netværk og andre metoder til maskinlæring har revolutioneret vores evne til hurtigt at identificere naturlige billeder, der ligner dem, der tidligere er set og katalogiseret.
Disse metoder "lærer" ved at indstille et forbløffende stort sæt skjulte interne parametre, styret af millioner af taggede billeder, og kræver store mængder supercomputer tid. Men hvad nu hvis du ikke har så mange taggede billeder? På mange områder, sådan en database er en uopnåelig luksus. Biologer optager cellebilleder og skitserer omhyggeligt grænserne og strukturen i hånden:det er ikke usædvanligt, at en person bruger uger på at komme med et enkelt fuldt tredimensionelt billede. Materialeforskere bruger tomografisk rekonstruktion til at kigge inde i sten og materialer, og derefter rulle ærmerne op for at mærke forskellige regioner, identificere revner, brud, og hulrum i hånden. Kontraster mellem forskellige, men vigtige strukturer er ofte meget små, og "støj" i dataene kan maskere funktioner og forvirre de bedste algoritmer (og mennesker).
Disse dyrebare håndkurerede billeder er ikke nær nok til traditionelle maskinlæringsmetoder. For at imødekomme denne udfordring, matematikere på CAMERA angreb problemet med maskinlæring fra meget begrænsede datamængder. Forsøger at gøre "mere med mindre, "deres mål var at finde ud af, hvordan man opbygger et effektivt sæt matematiske" operatører ", der i høj grad kan reducere antallet af parametre. Disse matematiske operatører kan naturligvis inkorporere centrale begrænsninger for at hjælpe med identifikation, såsom ved at inkludere krav til videnskabeligt sandsynlige former og mønstre.
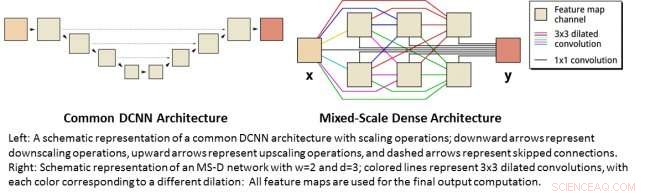
Til venstre:En skematisk fremstilling af en fælles DCNN -arkitektur med skaleringsoperationer; nedadgående pile repræsenterer nedskaleringsoperationer, opadgående pile repræsenterer opskalering, og stiplede pile repræsenterer overspringede forbindelser. Til højre:Skematisk fremstilling af et MS-D-netværk med w =2 og d =3; farvede linjer repræsenterer 3x3 dilaterede konvolutter, med hver farve svarende til en anden udvidelse:Alle funktionskort bruges til den endelige outputberegning. Kredit:Daniël Pelt og James Sethian, Berkeley Lab
Mixed-skala tætte konvolutionsneurale netværk
Mange anvendelser af maskinlæring til billeddannelsesproblemer bruger dybe konvolutonære neurale netværk (DCNN'er), hvor inputbilledet og mellembillederne er indviklet i et stort antal successive lag, gør det muligt for netværket at lære meget ikke -lineære funktioner. For at opnå nøjagtige resultater ved vanskelige billedbehandlingsproblemer, DCNN'er er typisk afhængige af kombinationer af yderligere operationer og forbindelser, herunder, for eksempel, nedskalering og opskalering af operationer for at fange funktioner på forskellige billedskalaer. For at træne dybere og mere kraftfulde netværk, yderligere lagtyper og forbindelser er ofte nødvendige. Endelig, DCNN'er bruger typisk et stort antal mellembilleder og træningsbare parametre, ofte mere end 100 mio. at opnå resultater for vanskelige problemer.
I stedet, den nye "Mixed-Scale Dense" netværksarkitektur undgår mange af disse komplikationer og beregner udvidede konvolutions som en erstatning for skaleringsoperationer for at fange funktioner på forskellige rumlige områder, anvender flere skalaer i et enkelt lag, og tæt forbinder alle mellemliggende billeder. Den nye algoritme opnår nøjagtige resultater med få mellemliggende billeder og parametre, eliminerer både behovet for at indstille hyperparametre og yderligere lag eller forbindelser for at muliggøre træning.
Få videnskab med høj opløsning fra data med lav opløsning
En anden udfordring er at producere billeder i høj opløsning fra input med lav opløsning. Som enhver, der har forsøgt at forstørre et lille foto og fundet det kun bliver værre, efterhånden som det bliver større, det lyder næsten umuligt. Men et lille sæt træningsbilleder behandlet med et blandet-skala tæt netværk kan give virkelig fremgang. Som et eksempel, forestil dig at forsøge at denomisere tomografiske rekonstruktioner af et fiberforstærket minikompositmateriale. I et forsøg beskrevet i avisen, billeder blev rekonstrueret ved hjælp af 1, 024 erhvervede røntgenprojektioner for at opnå billeder med relativt lave støjmængder. Støjende billeder af det samme objekt blev derefter opnået ved at rekonstruere ved hjælp af 128 projektioner. Træningsinput var støjende billeder, med tilsvarende lydløse billeder, der bruges som måloutput under træning. Det uddannede netværk kunne derefter effektivt tage støjende inputdata og rekonstruere billeder med højere opløsning.
Nye applikationer
Pelt og Sethian tager deres tilgang til en lang række nye områder, såsom hurtig realtidsanalyse af billeder, der kommer fra synkrotronlyskilder og genopbygningsproblemer i biologisk rekonstruktion, f.eks. for celler og hjernekortlægning.
"Disse nye tilgange er virkelig spændende, da de vil muliggøre anvendelse af maskinlæring på en langt større række billeddannelsesproblemer end i øjeblikket muligt, "Sagde Pelt." Ved at reducere mængden af nødvendige træningsbilleder og øge størrelsen på billeder, der kan behandles, den nye arkitektur kan bruges til at besvare vigtige spørgsmål inden for mange forskningsområder. "
 Varme artikler
Varme artikler
-
 Under pres, Apple viser det annekterede Krim som Rusland på appsDen amerikanske teknologigigant Apple holdt samtaler med Rusland, før de viste den annekterede halvø Krim som russisk territorium på sine apps Den amerikanske teknologigigant Apple har efterkommet
Under pres, Apple viser det annekterede Krim som Rusland på appsDen amerikanske teknologigigant Apple holdt samtaler med Rusland, før de viste den annekterede halvø Krim som russisk territorium på sine apps Den amerikanske teknologigigant Apple har efterkommet -
 Byggeforsinkelser gør nye atomkraftværker dyrere end nogensindeKredit:Imperial College London Omkostningerne ved at bygge nye atomkraftværker er næsten 20 procent højere end forventet på grund af forsinkelser, en ny analyse har fundet. En ny analyse af histo
Byggeforsinkelser gør nye atomkraftværker dyrere end nogensindeKredit:Imperial College London Omkostningerne ved at bygge nye atomkraftværker er næsten 20 procent højere end forventet på grund af forsinkelser, en ny analyse har fundet. En ny analyse af histo -
 I Paris, Airbnb får skylden for alle former for dårligdommeI denne torsdag, 20. september, 2018 foto, en rengøringsdame arbejder i en lejlighed beliggende på Airbnb i Paris. Airbnbs spektakulære vækst i Paris, den bedste verdensomspændende placering for inter
I Paris, Airbnb får skylden for alle former for dårligdommeI denne torsdag, 20. september, 2018 foto, en rengøringsdame arbejder i en lejlighed beliggende på Airbnb i Paris. Airbnbs spektakulære vækst i Paris, den bedste verdensomspændende placering for inter -
 Ny AI dyb læringsmodel tillader tidligere, mere præcise ozonadvarslerYunsoo Choi, venstre, lektor ved Institut for Jord- og Atmosfæriske Videnskaber ved UH, og ph.d. studerende Alqamah Sayeed forklare en ny model til bedre at forudsige ozonniveauer. Kredit:University o
Ny AI dyb læringsmodel tillader tidligere, mere præcise ozonadvarslerYunsoo Choi, venstre, lektor ved Institut for Jord- og Atmosfæriske Videnskaber ved UH, og ph.d. studerende Alqamah Sayeed forklare en ny model til bedre at forudsige ozonniveauer. Kredit:University o
- Livscyklus af en Turtle
- Hot ring producerer mikrobølge-drevne ultralydspulser trådløst
- Vedvarende energi og kampen om guatemalanske floder
- Lufthansa reducerer flådestørrelsen, Luk Germanwings, når virus rammer
- Fra Bigfoot til Nessie:7 legendariske kryptider, der holder dig oppe om natten
- Hubble får øje på en grøn kosmisk bue


