Der er desperat brug for nye antibiotika - maskinlæring kan hjælpe
Kredit:CC0 Public Domain
Forskere ved Stanford har skabt en algoritme, der styret af tidligere forskning, opstiller de DNA-sekvenser, der med størst sandsynlighed er på linje med antimikrobielle egenskaber.
Da truslen om antibiotikaresistens truer, mikrobiologer er ikke de eneste, der tænker på nye løsninger. James Zou, Ph.d., assisterende professor i biomedicinsk datavidenskab ved Stanford, har anvendt maskinlæring til at skabe en algoritme, der genererer tusindvis af helt nye virtuelle DNA-sekvenser med den hensigt at en dag skabe antimikrobielle proteiner.
Algoritmen, kaldet Feedback GAN, fungerer i det væsentlige som en masseproducent af forskellige DNA-fragmenter. Og selvom disse sekvensforsøg er noget tilfældige, Algoritmen virker ikke blindt. Det baserer de nye mulige peptider, eller små grupper af aminosyrer, på tidligere forskning, der udstikker de DNA-sekvenser, der mest sandsynligt stemmer overens med antimikrobielle egenskaber.
For nu, disse skabeloner, som ikke findes i naturen, er teoretiske, genereret på en computer. Men i lyset af stigende bekymringer om mikroberesistens, Zou sagde, at det er vigtigt at tænke på løsninger, der ikke allerede eksisterer.
"Vi valgte at forfølge antimikrobielle proteiner, fordi det er et meget vigtigt, problem med stor indvirkning, som også er et relativt løseligt problem for algoritmen, " sagde Zou. "Der er eksisterende værktøjer, som vi inkorporerer i vores system, der evaluerer, om en ny sekvens sandsynligvis har egenskaberne af et vellykket antimikrobielt protein."
Feedback GAN bygger på det, arbejder på at inkorporere den helt rigtige balance mellem tilfældige tilfældigheder og præcision.
Et papir, der beskriver algoritmen, blev offentliggjort online 11. februar i Nature Machine Learning . Anvita Gupta, en studerende i datalogi, er den første forfatter; Zou er ledende forfatter.
Selvforfinende
Gupta og Zous algoritme udløser ikke kun nye kombinationer af DNA. Den forfiner sig også aktivt, lære, hvad der virker, og hvad der ikke virker gennem en feedback-loop:Efter at algoritmen spytter en lang række DNA-sekvenser ud, det kører en trial-and-error-læringsproces, der gennemskuer peptidforslagene. Baseret på deres lighed med andre kendte antimikrobielle peptider, de "gode" bliver ført tilbage til algoritmen for at informere fremtidige DNA-sekvenser genereret fra koden, og for at blive raffineret selv.
"Der er en indbygget dommer og, ved at have denne feedback loop, systemet lærer at modellere nygenererede sekvenser efter dem, der anses for at have antimikrobielle egenskaber, "Så sagde Zou. "Så ideen er både individuelle peptidsekvenser og genereringen af sekvenserne bliver bedre og bedre."
Zou har også overvejet en anden kernekomponent i hypotetiske proteiner:proteinfoldning. Proteiner forvrænges til meget specifikke strukturer knyttet til deres funktioner. En algoritme kunne skabe den perfekte sekvens, men medmindre den kan foldes sammen, det er ubrugeligt - som tandhjulene på et ur, der er strøet på et bord.
Zou kan justere algoritmen, så i stedet for at analysere en tilbøjelighed til antimikrobielle egenskaber, det bestemmer sandsynligheden for korrekt foldning.
"Vi kan faktisk gøre disse to ting parallelt, hvor vi ser på antimikrobielle egenskaber af en sekvens og folde sandsynligheden for en anden, " sagde Zou. "Vi kører begge, så vi optimerer enten de antimikrobielle egenskaber eller dens evne til at folde."
Næste, Zou håber at fusionere de to variationer af algoritmen for at skabe peptidsekvenser, der er optimeret til både deres mikrobe-dræbende evner og deres evne til at folde til et ægte protein.
 Varme artikler
Varme artikler
-
 Amazon leverer rekordoverskud på gevinster i skyen, annonceringPå trods af aftagende vækst i online detailsalg, Amazon leverede sit bedste overskud nogensinde i første kvartal takket være store gevinster i cloud computing og abonnementer Amazon leverede torsd
Amazon leverer rekordoverskud på gevinster i skyen, annonceringPå trods af aftagende vækst i online detailsalg, Amazon leverede sit bedste overskud nogensinde i første kvartal takket være store gevinster i cloud computing og abonnementer Amazon leverede torsd -
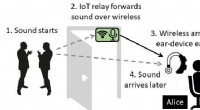 Forskere udvikler metode til at annullere støj uden øreblokerende hovedtelefonerDette diagram viser, hvordan det nye støjreduceringssystem ville fungere. Overførsel af lyd via et trådløst netværk hurtigere end lyd rejser alene. Kredit:Sheng Shen Forstyrrende støj er næsten ov
Forskere udvikler metode til at annullere støj uden øreblokerende hovedtelefonerDette diagram viser, hvordan det nye støjreduceringssystem ville fungere. Overførsel af lyd via et trådløst netværk hurtigere end lyd rejser alene. Kredit:Sheng Shen Forstyrrende støj er næsten ov -
 Snapchat lancerer egen multi-player gaming platformSnapchat – den populære beskedapp – lancerer sin egen multiplayer-spilplatform Messaging app Snapchat, som er meget populær blandt yngre brugere, men har kæmpet for at tjene penge siden oprettelse
Snapchat lancerer egen multi-player gaming platformSnapchat – den populære beskedapp – lancerer sin egen multiplayer-spilplatform Messaging app Snapchat, som er meget populær blandt yngre brugere, men har kæmpet for at tjene penge siden oprettelse -
 Russisk regulator flytter for at blokere Telegram-beskedappTelegrams russiske grundlægger har lovet at afvise ethvert forsøg fra sikkerhedstjenesterne på at få bagdørsadgang til appen Ruslands telekommunikationsvagthund bad fredag en Moskva-domstol om a
Russisk regulator flytter for at blokere Telegram-beskedappTelegrams russiske grundlægger har lovet at afvise ethvert forsøg fra sikkerhedstjenesterne på at få bagdørsadgang til appen Ruslands telekommunikationsvagthund bad fredag en Moskva-domstol om a
- Hvordan man opløser stål
- Billede:Satellitudsigter brande i Californien
- AI er endnu ikke smart nok til at redde os fra falske nyheder:Facebook -brugere (og deres bias) er n…
- NASA vil lancere en delikat opbevaring af OSIRIS-REx asteroideprøver
- Brug af lys til at identificere chirale molekyler til lægemidler
- Sådan fungerer lufthavne


