En webapplikation til at udtrække nøgleinformation fra tidsskriftsartikler

Et skærmbillede af DIVE-webstedet. Kredit:Gupta et al.
Akademiske artikler indeholder ofte beretninger om nye gennembrud og interessante teorier relateret til en række forskellige områder. Imidlertid, de fleste af disse artikler er skrevet ved hjælp af jargon og teknisk sprog, der kun kan forstås af læsere, der er fortrolige med det pågældende studieområde.
Ikke-ekspert læsere er derfor typisk ude af stand til at forstå videnskabelige artikler, medmindre de er kurateret og gjort mere tilgængelige af tredjeparter, som forstår de begreber og ideer, der er indeholdt i dem. Med det i tankerne, et team af forskere ved Texas Advanced Computing Center på University of Texas i Austin (TACC), Oregon State University (OSU) og American Society of Plant Biologists (ASPB) har sat sig for at udvikle et værktøj, der automatisk kan udtrække vigtige sætninger og terminologi fra forskningsartikler for at give nyttige definitioner og forbedre deres læsbarhed.
"Vores projekt er motiveret af behovet for at forbedre læsbarheden af tidsskriftsartikler, "Weijia Xu, som leder teamet hos TACC, fortalte TechXplore. "Det er en fælles indsats mellem biologiske kuratorer, tidsskriftsudgivere og dataloger, der sigter mod at udvikle en webservice, der kan genkende og muliggøre forfatterkuration af vigtig terminologi, der bruges i tidsskriftspublikationer. Terminologien og ordene er derefter knyttet til slutningen af tidsskriftsartiklen for at øge dens tilgængelighed for læserne."
Xu og hans kolleger udviklede en udvidelsesbar ramme, der kan bruges til at udtrække information fra dokumenter. De implementerede derefter denne ramme i en webservice kaldet DIVE (Domain Information Vocabulary Extraction), integrere den med ASPB's pipeline for tidsskriftspublikationer. I modsætning til eksisterende værktøjer til at udtrække domæneoplysninger, deres rammer kombinerer flere tilgange, herunder ontologi-guidet udvinding, regelbaseret udvinding, naturlig sprogbehandling (NLP) og deep learning -teknikker.

Arkitekturoversigten over systemet foreslået af forskerne. Kredit:Gupta et al.
"Resultaterne opnået af forskellige modeller gemmes derefter i en centraliseret database, " Xu forklarede. "Vi har også designet en webservice, der giver brugerne mulighed for at kurere ekstraktionsresultater. Webservicen er integreret med produktionspublikationspipeline hos ASPB."
Når preview -versionen af en journalartikel er indsendt og kommer ind i ASPB's pipeline, manuskriptet fodres automatisk til DIVE, som behandler det og producerer en URL, hvormed forfatteren vil kunne få adgang til behandlingsresultaterne af DIVE. Forfatteren af papiret bliver bedt om at besøge det angivne link og gennemgå den udtrukne information, før han/hun er i stand til officielt at indsende papiret.
"Forfatteren er nødt til at besøge DIVE-stedet for at gennemgå ekstraktionsresultaterne og foretage den endelige godkendelse af listen over oplysninger, der skal inkluderes i slutningen af deres artikel, " sagde Xu. "DIVE sporer også forfatterrettelser for at forbedre fremtidige ekstraktionsopgaver. I øjeblikket, ingen anden tidsskriftsudgiver har valgt en lignende tilgang og integreret den med deres publikationspipeline."
Under sine analyser og ved udtrækning af nøgledata fra dokumenter, rammerne udviklet af forskerne bruger flere teknikker. Dette gør det muligt for den at fange mere information end andre metoder, f.eks. ABNER (A Biomedical Named Entity Recognizer), som er et open source-softwareværktøj til molekylærbiologisk tekstmining, der kun kan udtrække generelle termer (f.eks. gener og proteiner). I modsætning til DIVE, ABNER er kun baseret på betingede tilfældige felter (CRF'er), en statistisk modelleringsmetode, der almindeligvis bruges i mønstergenkendelse og maskinlæringsapplikationer.
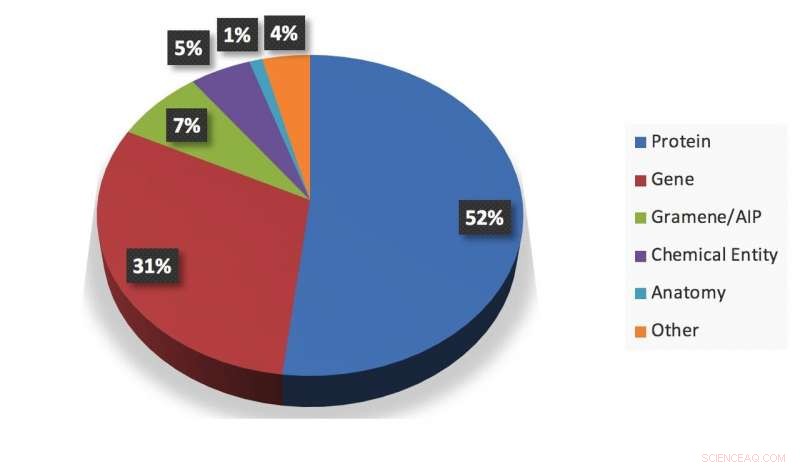
En visuel oversigt over et øjebliksbillede af information udtrukket af systemet. Kredit:Gupta et al.
"Et væsentligt bidrag fra vores projekt er, at det hjælper med at bygge datasæt og modeller, der kan udlede forfatternes forskningsinteresser ud fra deres publikationer, " Xu sagde. "Vores projekt kan gavne bredere samfund af biologiske forskere. For forfattere, udtræk og inddragelse af nøgleinformation kan øge tilgængeligheden af deres artikler. "
Xu og hans kollega Amit Gupta evaluerede deres rammer og sammenlignede dens ydeevne med andre informationsudvindingsværktøjer, inklusive ABNER. Deres resultater afslørede, at brug af flere tilgange, herunder dyb læring, DIVE opnår højere præcisionsscore end andre præ-trænede modeller udelukkende baseret på CRF'er. Interessant nok, DIVE-rammen kan også løbende opdateres, da der til enhver tid kan tilføjes ekstra ekstraktionsmodeller.
DIVE-webapplikationen giver ikke kun ikke-ekspert læsere mulighed for bedre at forstå akademiske artikler, det kan også hjælpe dem med at identificere papirer, der er tilpasset deres interesser. Forskere, på den anden side, kan bruge DIVE til at holde sig orienteret om bestemte forskningsområder, samt at lære om ny terminologi og tendenser relateret til deres interessefelt. Endelig, de oplysninger, der genereres af applikationen, kan også vejlede biologikuratorer i deres beslutninger og dataindsamlingsprocesser.
"Vi fortsætter vores projekt ved at udforske to retninger, " sagde Xu. "På den ene side, vi undersøger nye metoder til at inkorporere med vores informationsekstraktionsmodeller for at forbedre ydeevnen. På den anden side, vi forsøger også at udvide vores service ved at tilbyde den til flere brugersamfund og tidsskriftsudgivere. "
© 2019 Science X Network
 Varme artikler
Varme artikler
-
 Model hjælper robotter med at navigere mere som mennesker gørMIT-forskere har udtænkt en måde at hjælpe robotter med at navigere i miljøer mere som mennesker gør. Kredit:Massachusetts Institute of Technology Når man bevæger sig gennem en menneskemængde for
Model hjælper robotter med at navigere mere som mennesker gørMIT-forskere har udtænkt en måde at hjælpe robotter med at navigere i miljøer mere som mennesker gør. Kredit:Massachusetts Institute of Technology Når man bevæger sig gennem en menneskemængde for -
 Twitter forbereder flygtige tweets, begynder at teste i BrasilienDenne 26. april, 2017, filbillede viser Twitter-app-ikonet på en mobiltelefon i Philadelphia. Twitter starter onsdag d. 4. marts 2020, at teste tweets, der forsvinder efter 24 timer, selvom den nye fu
Twitter forbereder flygtige tweets, begynder at teste i BrasilienDenne 26. april, 2017, filbillede viser Twitter-app-ikonet på en mobiltelefon i Philadelphia. Twitter starter onsdag d. 4. marts 2020, at teste tweets, der forsvinder efter 24 timer, selvom den nye fu -
 Air France siger at reducere kapaciteten mellem 70 og 90% i løbet af de næste to månederKredit:CC0 Public Domain Air France sagde mandag, at det vil reducere flykapaciteten med 70-90 procent i løbet af de næste to måneder på grund af coronavirusudbruddet, slutter sig til en lang række
Air France siger at reducere kapaciteten mellem 70 og 90% i løbet af de næste to månederKredit:CC0 Public Domain Air France sagde mandag, at det vil reducere flykapaciteten med 70-90 procent i løbet af de næste to måneder på grund af coronavirusudbruddet, slutter sig til en lang række -
 NOAA-satellitter hjalp med at redde 340 mennesker i 2018Kredit:NOAA Piloten på robåden Alba havde et ædelt mål - at skabe opmærksomhed og midler til Scottish Association for Mental Health. Og han skulle til række 3, 400 sømil, fra Norfolk, Va., til sit
NOAA-satellitter hjalp med at redde 340 mennesker i 2018Kredit:NOAA Piloten på robåden Alba havde et ædelt mål - at skabe opmærksomhed og midler til Scottish Association for Mental Health. Og han skulle til række 3, 400 sømil, fra Norfolk, Va., til sit
- Huawei inviterer udenlandske medier til selv at se på spionpåstande
- Genbrug af saltvand kan hjælpe med at kontrollere skadelige mikrober på boresteder i Alberta
- Til støtte for økologisk forvaltning, mere levende end digitalt
- Robotter kører for at redde, da leveringsrisici stiger
- Kina afslutter opførelsen af et stabilt højt magnetfelt anlæg
- Stjerne-skælvsvibrationer fører til et nyt skøn for Mælkevejens alder


