Forskere konverterer 2-D-billeder til 3-D ved hjælp af dyb læring
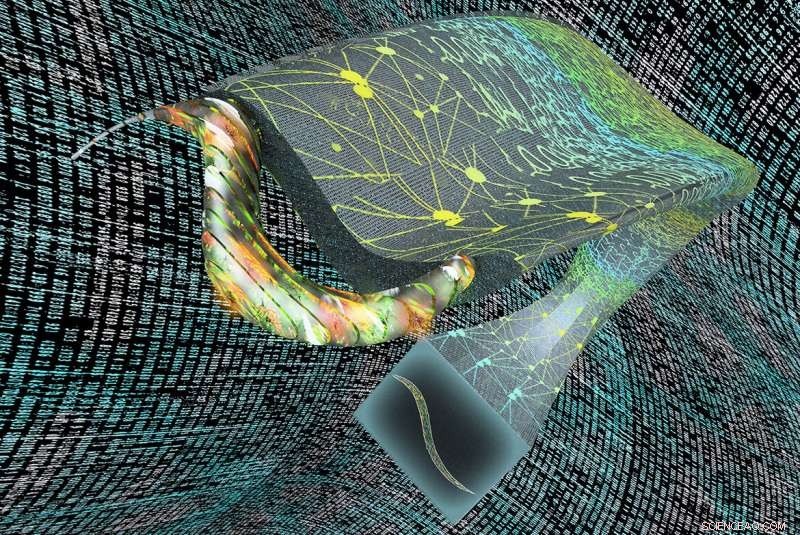
Illustrationen repræsenterer Deep-Z, en kunstig intelligens-baseret ramme, der digitalt kan omfokusere et 2D-fluorescensmikroskopbillede (nederst) for at producere 3D-skiver (til venstre). Kredit:Ozcan Lab/UCLA
Et UCLA -forskerhold har udtænkt en teknik, der udvider fluorescensmikroskopiens muligheder, som giver forskerne mulighed for præcist at mærke dele af levende celler og væv med farvestoffer, der lyser under speciel belysning. Forskerne bruger kunstig intelligens til at omdanne todimensionelle billeder til stakke af virtuelle tredimensionelle skiver, der viser aktivitet inde i organismer.
I en undersøgelse offentliggjort i Naturens metoder , forskerne rapporterede også, at deres rammer, kaldet "Deep-Z, " var i stand til at rette fejl eller aberrationer i billeder, som når en prøve er vippet eller buet. Yderligere, de demonstrerede, at systemet kunne tage 2-D-billeder fra én type mikroskop og virtuelt skabe 3-D-billeder af prøven, som om de blev taget af en anden, mere avanceret mikroskop.
"Dette er en meget kraftfuld ny metode, der er muliggjort ved dyb læring til at udføre 3-D-billeddannelse af levende prøver, med den mindste eksponering for lys, som kan være giftig for prøver, " sagde seniorforfatter Aydogan Ozcan, UCLA-kanslerens professor i elektro- og computerteknik og associeret direktør for California NanoSystems Institute ved UCLA.
Ud over at skåne prøver fra potentielt skadelige doser af lys, dette system kunne tilbyde biologer og life science-forskere et nyt værktøj til 3D-billeddannelse, der er enklere, hurtigere og meget billigere end de nuværende metoder. Muligheden for at korrigere for aberrationer kan give forskere, der studerer levende organismer, mulighed for at indsamle data fra billeder, som ellers ville være ubrugelige. Efterforskere kunne også få virtuel adgang til dyrt og kompliceret udstyr.
Denne forskning bygger på en tidligere teknik, Ozcan og hans kolleger udviklede, som gjorde det muligt for dem at gengive 2-D fluorescensmikroskopbilleder i superopløsning. Begge teknikker fremmer mikroskopi ved at stole på dyb læring - ved at bruge data til at "træne" et neuralt netværk, et computersystem inspireret af den menneskelige hjerne.
Deep-Z blev undervist ved hjælp af eksperimentelle billeder fra et scannende fluorescensmikroskop, som tager billeder fokuseret på flere dybder for at opnå 3-D-billeddannelse af prøver. I tusindvis af træningsløb, det neurale netværk lærte, hvordan man tager et 2-D-billede og udleder nøjagtige 3-D-snit i forskellige dybder i en prøve. Derefter, rammen blev testet blindt - fodret med billeder, der ikke var en del af dens træning, med de virtuelle billeder sammenlignet med de faktiske 3D-skiver opnået fra et scanningsmikroskop, giver et fremragende match.
Ozcan og hans kolleger anvendte Deep-Z på billeder af C. elegans, en rundorm, der er en almindelig model inden for neurovidenskab på grund af dets enkle og velforståede nervesystem. Konvertering af en 2-D-film af en orm til 3-D, ramme for ramme, forskerne var i stand til at spore aktiviteten af individuelle neuroner i ormens krop. Og startende med et eller to 2-D billeder af C. elegans taget i forskellige dybder, Deep-Z producerede virtuelle 3-D billeder, der gjorde det muligt for holdet at identificere individuelle neuroner i ormen, matcher et scanningsmikroskops 3-D output, undtagen med meget mindre lyseksponering for den levende organisme.
Forskerne fandt også ud af, at Deep-Z kunne producere 3-D-billeder fra 2-D-overflader, hvor prøver var vippede eller buede - selvom det neurale netværk kun blev trænet med 3-D-skiver, der var perfekt parallelle med prøvens overflade.
"Denne funktion var faktisk meget overraskende, " sagde Yichen Wu, en UCLA-kandidatstuderende, der er medførsteforfatter til publikationen. "Med det, du kan se gennem krumning eller anden kompleks topologi, som er meget udfordrende at afbilde."
I andre forsøg, Deep-Z blev trænet med billeder fra to typer fluorescensmikroskoper:wide-field, som udsætter hele prøven for en lyskilde; og konfokal, som bruger en laser til at scanne en prøve del for del. Ozcan og hans team viste, at deres ramme derefter kunne bruge 2-D vidfeltmikroskopbilleder af prøver til at producere 3D-billeder, der næsten er identiske med dem, der er taget med et konfokalt mikroskop.
Denne konvertering er værdifuld, fordi det konfokale mikroskop skaber billeder, der er skarpere, med mere kontrast, sammenlignet med det brede felt. På den anden side, bredfeltsmikroskopet tager billeder med færre omkostninger og med færre tekniske krav.
"Dette er en platform, der generelt er anvendelig til forskellige par mikroskoper, ikke kun bredfelt-til-konfokal konvertering, " sagde co-first forfatter Yair Rivenson, UCLA assisterende adjungeret professor i elektro- og computerteknik. "Hvert mikroskop har sine egne fordele og ulemper. Med denne ramme, du kan få det bedste fra begge verdener ved at bruge kunstig intelligens til at forbinde forskellige typer mikroskoper digitalt."
Andre forfattere er kandidatstuderende Hongda Wang og Yilin Luo, postdoc-stipendiat Eyal Ben-David og Laurent Bentolila, videnskabelig direktør for California NanoSystems Institute's Advanced Light Microscopy and Spectroscopy Laboratory, hele UCLA; og Christian Pritz fra hebraisk universitet i Jerusalem i Israel.
 Varme artikler
Varme artikler
-
 Hvordan virker en fløjte?Whistles gennembler ofte det daglige livs stof: en dommer foretager et afgørende opkald i de sidste øjeblikke i et spil; en krydsningsvagt signalerer børn, at det er okay at krydse gaden; og en kæledy
Hvordan virker en fløjte?Whistles gennembler ofte det daglige livs stof: en dommer foretager et afgørende opkald i de sidste øjeblikke i et spil; en krydsningsvagt signalerer børn, at det er okay at krydse gaden; og en kæledy -
 Forskere forvandler atomkraftmikroskopmålinger til farvebillederSkematisk af farven atomkraftmikroskop. Kredit:2017 Hideki Kawakatsu, Kawakatsu Laboratory, Institute of Industrial Science, University of Tokyo. En fransk og japansk forskergruppe har udviklet en
Forskere forvandler atomkraftmikroskopmålinger til farvebillederSkematisk af farven atomkraftmikroskop. Kredit:2017 Hideki Kawakatsu, Kawakatsu Laboratory, Institute of Industrial Science, University of Tokyo. En fransk og japansk forskergruppe har udviklet en -
 Ingeniører gør klare dråber producerer iriserende farverEn petriskål med transparente dråbeemulsioner fremstillet af en blanding af olier med forskellig densitet og vandbaserede overfladeaktive stoffer fotograferet fra forskellige vinkler. Når de belyses m
Ingeniører gør klare dråber producerer iriserende farverEn petriskål med transparente dråbeemulsioner fremstillet af en blanding af olier med forskellig densitet og vandbaserede overfladeaktive stoffer fotograferet fra forskellige vinkler. Når de belyses m -
 Kernen – kommer snart i 3DDet radiale tidsprojektionskammer, vist installeret i Jefferson Labs forsøgshal B, blev bygget til at måle kernerne af helium-4 i eksperimentet. Denne måling viser, at 3D-billeddannelse af kernens ind
Kernen – kommer snart i 3DDet radiale tidsprojektionskammer, vist installeret i Jefferson Labs forsøgshal B, blev bygget til at måle kernerne af helium-4 i eksperimentet. Denne måling viser, at 3D-billeddannelse af kernens ind
- Ny model forudsiger ændringer i træ
- Paris bremser for delebiler
- Invasiv kalve breder sig nordpå, truer Californias håndflader
- Agrivoltaik viser gensidig fordel på tværs af fødevarer, vand, energiforbindelse
- Netflix samarbejder med Italiens undersøgelse af skatteunddragelse
- Ny forskning identificerer New Bedford Harbor som en vigtig kilde til luftbårne PCB'er


