Feedback fra tusindvis af designs kunne transformere proteinteknologi
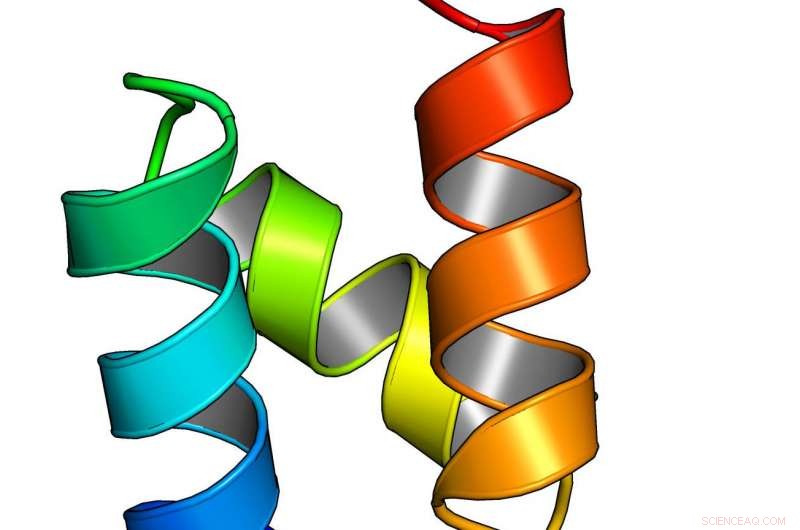
En model af et beregningsmæssigt designet miniprotein fra en storstilet undersøgelse fra UW Medicine Institute for Protein Design. Kredit:UW Medicine Institute for Protein Design
Scenen er sat til en ny æra af datadrevet proteinmolekylær konstruktion, da fremskridt inden for DNA-synteseteknologi smelter sammen med forbedringer i beregningsmæssigt design af nye proteiner.
Denne uges Videnskab rapporterer den største test af foldningsstabilitet for computerdesignede proteiner, muliggjort af en ny high-throughput tilgang.
Forskerne er fra UW Medicine Institute for Protein Design ved University of Washington i Seattle og University of Toronto i Ontario.
Hovedforfatteren af papiret er Gabriel Rocklin, en postdoc i biokemi ved University of Washington School of Medicine. Seniorforfatterne er Cheryl Arrowsmith, af Princess Margaret Cancer Center, Structural Genomics Consortium og Institut for Medicinsk Biofysik ved University of Toronto, og David Baker, UW professor i biokemi og en Howard Hughes Medical Institute-forsker.
Proteiner er biologiske arbejdsheste. Forskere vil bygge nye molekyler, ikke findes naturligt, der kan udføre opgaver med at forebygge eller behandle sygdom, i industrielle applikationer, i energiproduktion, og i miljøoprydning.
"Imidlertid, computerdesignede proteiner formår ofte ikke at danne de foldede strukturer, som de er designet til at have, når de rent faktisk testes i laboratoriet, " sagde Rocklin.
I den seneste undersøgelse, forskerne testede mere end 15, 000 nydesignede miniproteiner, der ikke findes i naturen for at se, om de danner foldede strukturer. Selv store proteindesignundersøgelser i de sidste par år har generelt kun undersøgt 50 til 100 designs.
"Vi lærte enormt meget på denne nye skala, men smagen har givet os en endnu større appetit, " sagde Rocklin. "Vi er ivrige efter at teste hundredtusindvis af designs i de næste par år."
Den seneste test førte til designet af 2, 788 stabile proteinstrukturer og kunne have mange bioteknologiske og syntetiske biologiapplikationer. Deres lille størrelse kan være fordelagtig til behandling af sygdomme, når lægemidlet skal nå ind i en celle.
Proteiner er lavet af aminosyrekæder med specifikke sekvenser, og naturlige proteinsekvenser er kodet i cellulært DNA. Disse kæder foldes til 3-dimensionelle konformationer. Rækkefølgen af aminosyrerne i kædeguiden, hvor den vil bøje og vride sig, og hvordan dele vil interagere for at holde strukturen sammen.
I årtier, forskere har studeret disse interaktioner ved at undersøge strukturerne af naturligt forekommende proteiner. Imidlertid, naturlige proteinstrukturer er typisk store og komplekse, med tusindvis af interaktioner, der tilsammen holder proteinet i dets foldede form. Det bliver meget vanskeligt at måle bidraget fra hver interaktion.
Forskerne adresserede dette problem ved beregningsmæssigt at designe deres egne, meget simplere proteiner. Disse simplere proteiner gjorde det lettere at analysere de forskellige typer af interaktioner, der holder alle proteiner i deres foldede strukturer.
"Stadig, selv simple proteiner er så komplicerede, at det var vigtigt at studere tusindvis af dem for at finde ud af, hvorfor de folder, " sagde Rocklin. "Dette havde været umuligt indtil for nylig, på grund af omkostningerne til DNA. Hvert designet protein kræver sit eget tilpassede stykke DNA, så det kan laves inde i en celle. Dette har begrænset tidligere undersøgelser til kun at teste snesevis af designs."
For at kode deres design af korte proteiner i dette projekt, forskerne brugte det, der kaldes DNA-oligo-biblioteksynteseteknologi. Det blev oprindeligt udviklet til andre laboratorieprotokoller, såsom stor gensamling. En af de virksomheder, der har leveret deres DNA, er CustomArray i Bothell, Wash. De brugte også DNA-biblioteker lavet af Agilent i Santa Clara, Californien, og Twist Bioscience i San Francisco.
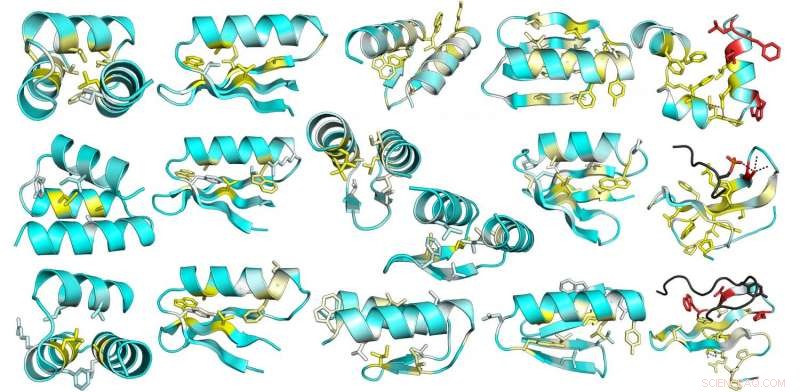
Dette billede er fra en omfattende mutationsanalyse af stabilitet i designet og naturlige proteiner. Den gennemsnitlige ændring i stabilitet på grund af mutation af hver position i 13 designede proteiner er afbildet på designmodelstrukturerne. Gul angiver positioner, hvor mutationer er mest destabiliserende; positioner, hvor der er ringe effekt, er blå Kredit:UW Medicine Institute for Protein Design
Ved at gentage beregningscyklussen og eksperimentel test over flere iterationer, forskerne lærte af deres designfejl og forbedrede gradvist deres modellering. Deres succesrate for design steg fra 6 procent til 47 procent. De producerede også stabile proteiner i former, hvor alle deres første design mislykkedes.
Deres store sæt af stabile og ustabile miniproteiner gjorde dem i stand til kvantitativt at analysere, hvilke proteinegenskaber der korrelerede med foldning. De sammenlignede også stabiliteten af deres designede proteiner med tilsvarende størrelse, naturligt forekommende proteiner.
Det mest stabile naturlige protein, forskerne identificerede, var et meget undersøgt protein fra bakterien Bacillus stearothermophilus. Denne organisme soler sig i høje temperaturer, som dem i varme kilder og havets termiske ventilationsåbninger. De fleste proteiner mister deres foldede strukturer under så høje temperaturforhold. Organismer, der trives der, har udviklet meget stabile proteiner, der forbliver foldede, selv når de er varme.
"I alt 774 designet proteiner havde en højere stabilitetsscore end dette mest protease-resistente monomere protein, " bemærkede forskerne. Proteaser er enzymer, der nedbryder proteiner, og var væsentlige værktøjer, forskerne brugte til at måle stabiliteten for deres tusindvis af proteiner.
Forskerne forudser, at i takt med at DNA-synteseteknologien bliver ved med at blive bedre, high-throughput proteindesign bliver muligt for større, mere komplekse proteinstrukturer.
"Vi bevæger os væk fra den gamle stil med proteindesign, som var en blanding af computermodellering, menneskelig intuition, og små bidder af beviser om, hvad der virkede før." Rocklin sagde. "Proteindesignere var som mesterhåndværkere, der brugte deres erfaring til at håndskulpturere hvert stykke i deres værksted. Nogle gange virkede tingene, men da de fejlede, var det svært at sige hvorfor. Vores nye tilgang lader os indsamle en enorm mængde data om, hvad der gør proteiner stabile. Disse data kan nu drive designprocessen."
 Varme artikler
Varme artikler
-
 Kemikere udvikler MRI-lignende teknik til at opdage, hvad der fejler batterierEt team af kemikere har udviklet en MRI-baseret teknik, der hurtigt kan diagnosticere, hvad der fejler visse typer batterier - fra at bestemme, hvor meget ladning der er tilbage til at opdage interne
Kemikere udvikler MRI-lignende teknik til at opdage, hvad der fejler batterierEt team af kemikere har udviklet en MRI-baseret teknik, der hurtigt kan diagnosticere, hvad der fejler visse typer batterier - fra at bestemme, hvor meget ladning der er tilbage til at opdage interne -
 Hvordan man laver tør is til FogTør is kan være et meget interessant stof. Det kan ikke kun bruges til at afkøle varer i et iskiste i lange perioder, det kan også bruges til at skabe tåge på grund af den ekstremt lave temperatur på
Hvordan man laver tør is til FogTør is kan være et meget interessant stof. Det kan ikke kun bruges til at afkøle varer i et iskiste i lange perioder, det kan også bruges til at skabe tåge på grund af den ekstremt lave temperatur på -
 Manipulerede enzymer – forskere sætter milepæl i biokatalyseforskningKathrin Heckenbichler og Rolf Breinbauer og deres team formåede for første gang nogensinde at genoplære et enzym til at bygge ringformede molekylære strukturer i stedet for at udføre sin naturlige opg
Manipulerede enzymer – forskere sætter milepæl i biokatalyseforskningKathrin Heckenbichler og Rolf Breinbauer og deres team formåede for første gang nogensinde at genoplære et enzym til at bygge ringformede molekylære strukturer i stedet for at udføre sin naturlige opg -
 Varmehærdet magnesiumlegering et stærkt valg til implantaterBillede fra elektronmikroskop, der viser et stort bundfald i magnesiumlegering. Disse bundfald opløses ved varmebehandling, hvilket reducerer korrosionshastigheden væsentligt og gør legeringen meget s
Varmehærdet magnesiumlegering et stærkt valg til implantaterBillede fra elektronmikroskop, der viser et stort bundfald i magnesiumlegering. Disse bundfald opløses ved varmebehandling, hvilket reducerer korrosionshastigheden væsentligt og gør legeringen meget s
- Nestende vaner af vilde kaniner
- Dyrkning af en dinosaur -middag
- UV-lys: Positive & Negative Effects
- Virkningen af orkaner på Puerto Ricos tørre skove
- Et skridt i retning af at kvitte register kvitteringer af BPA
- Høje energiregninger kan føre familier ud i fattigdom, viser en landsdækkende undersøgelse


