Effektiv og high-throughput teknik til at studere strukturen af DNA

Fra venstre mod højre:SEM-billede af DNA-bundter homogent suspenderet over en superhydrofob enhed. I det relaterede HRTEM-billede har frynserne en afstand på 2,7 Å, i god overensstemmelse med diffraktionsmønsteret:begge tilfælde viser afstanden mellem to baser i DNA-dobbelthelixen. Kredit:Monica Marini
Det kan være den mest berømte struktur i biologi, men det var først for et par år siden, at biofysiker Enzo di Fabrizio og hans kolleger tog de første direkte billeder af DNA-dobbelthelixen med et elektronmikroskop.
Nu, di Fabrizio og hans laboratoriegruppe på KAUST har forbedret deres banebrydende teknik, tweaking protokollen for at gøre den enklere og hurtigere.
"Det er et effektivt alternativ med høj gennemstrømning til mere konventionelle teknikker, " siger Monica Marini, postdoc i di Fabrizios laboratorium og førsteforfatter til det nye studie.
Billedet, der hjalp Watson og Crick med at tyde den proptrækkerlignende struktur af DNA for 65 år siden, blev taget ved hjælp af en teknik kaldet røntgenkrystallografi, som involverer spredning af elektromagnetisk stråling fra atomer i en krystalliseret form af DNA. I årtier, dette var den eneste måde at få 3D-gengivelser af livets byggesten.
Men de billeder var kun abstraktioner, baseret på fortolkninger af diffrakterede røntgenstråler. Det var ikke rigtige fotografier. Og det var først i 2012 - da di Fabrizio, tilbage i sit hjemland Italien, producerede det første direkte billede af DNA - at forskerne fik et trofast billede af den dobbelte helix.
Di Fabrizio flyttede til KAUST i 2013, og i løbet af de sidste fem år har hans gruppe støt forbedret og bygget videre på den originale billeddannelsesprotokol, som involverede transmissionselektronmikroskopi (TEM), en teknik, hvor elektroner udstråles på fotografisk film.
Deres metode går ud på at sprede små dråber af væske indeholdende DNA på siliciumwafers ætset med små cylindriske søjler og huller. Når dråberne tørrer ud, DNA'et strækker sig ud over det mikroskopiske leje af søjler, skabe spoler af indbyrdes forbundne tråde.
Tidligere, Di Fabrizios team havde anvendt TEM til at fange billeder af DNA-strengene direkte. Men nu, de har også udført en mere ligetil diffraktionsanalyse af TEM-strålerne - og dermed "skabt et eksperiment, der er ret ens, i form af fysiske principper, til det, der blev gjort af de videnskabsmænd, der først opdagede DNA-strukturen, " siger Andrea Falqui, et andet KAUST-fakultetsmedlem, der har samarbejdet med di Fabrizio i tidligere værker.
Som di Fabrizio og Marini viser, denne diffraktionsbaserede tilgang producerede billeder, der målte afstanden mellem trinene på DNA-stigen lige så præcist som direkte TEM-billeddannelse. Nu, forskerne planlægger at bruge denne teknik til at afbilde mere komplekse arrangementer af DNA. For eksempel, de vil se på DNA, når det interagerer med proteiner, stoffer eller tungmetaller.
Alle disse interaktioner "kan forårsage variationer i den uberørte konformation af den dobbelte helix, Marini siger, og snart skulle de have det fotografiske bevis for at se disse ændringer i detaljer.
Sidste artikelHurtigsporende endokrine analyser
Næste artikelEnerginiveaujustering for molekylær elektronik
 Varme artikler
Varme artikler
-
 En bedre kulstoffælde vil tage drivhusgasser ud af luften og tage dem i brugKredit:Rensselaer Polytekniske Institut Kulstoffangstteknologier spiller en afgørende rolle i at reducere drivhusgasemissioner fra kraftværker og fabrikker, mens du udnytter kuldioxid (CO 2 ) ti
En bedre kulstoffælde vil tage drivhusgasser ud af luften og tage dem i brugKredit:Rensselaer Polytekniske Institut Kulstoffangstteknologier spiller en afgørende rolle i at reducere drivhusgasemissioner fra kraftværker og fabrikker, mens du udnytter kuldioxid (CO 2 ) ti -
 Vand skaber fælder i organisk elektronikTanvi Upreti, ph.d.-studerende ved afdelingen for komplekse materialer og anordninger, som udførte eksperimenterne sammen med den første forfatter, Guangzheng Zuo. Kredit:Charlotte Perhammar Organ
Vand skaber fælder i organisk elektronikTanvi Upreti, ph.d.-studerende ved afdelingen for komplekse materialer og anordninger, som udførte eksperimenterne sammen med den første forfatter, Guangzheng Zuo. Kredit:Charlotte Perhammar Organ -
 Neutroner kigger ind i en kørende motorForskere brugte neutroner til at sondere en kørende motor ved ORNLs Spallation Neutron Source, giver dem mulighed for at teste en aluminium-cerium-legering under driftsforhold. Fra venstre, forskere O
Neutroner kigger ind i en kørende motorForskere brugte neutroner til at sondere en kørende motor ved ORNLs Spallation Neutron Source, giver dem mulighed for at teste en aluminium-cerium-legering under driftsforhold. Fra venstre, forskere O -
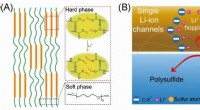 Stabiliserende svovlkatode med enkelt Li-ion-kanal polymerbindemiddel(A) Skematiske diagrammer over dannelsen af polymeren med enkeltionkanalerne. (B) Skematiske diagrammer af enkelt-ion-kanaler regulerer polysulfid- og Li-ion-hopningen. Kredit:©Science China Press
Stabiliserende svovlkatode med enkelt Li-ion-kanal polymerbindemiddel(A) Skematiske diagrammer over dannelsen af polymeren med enkeltionkanalerne. (B) Skematiske diagrammer af enkelt-ion-kanaler regulerer polysulfid- og Li-ion-hopningen. Kredit:©Science China Press
- Forskere finder ud af, at bygning af gangbroer påvirker landdistrikternes økonomier i oversvømmel…
- Støtten stiger til kontroversielle Canada -rørledning
- EU begynder blodig hård kamp for at nå grønne ambitioner
- Ny katalysator imødekommer udfordringen med at rense udstødningen fra moderne motorer
- Klasseværelsesaktiviteter på luftvejene
- Hvordan forbereder man sig på ugunstige vejrhændelser? Afhænger af dine tidligere erfaringer


