Kemikere udvikler en ny metode til at identificere proteiner

Kredit:University of Michigan
Proteiner er arbejderbierne i cellen, hovedsageligt slå sig sammen for at danne makromolekylære, multikomponentkomplekser til at udføre indviklede cellulære opgaver.
At forsøge at karakterisere sådanne proteinkomplekser og alle deres funktioner inden for organismer er en disciplin kaldet "proteomik." Historisk set forskere har undersøgt proteiners form og funktion ved at opløse dem med enzymer og sekvensere de resulterende små brudte stykker, kaldes peptider. Men at studere proteiner på denne måde medfører tab af en masse væsentlig information, siger Brandon Ruotolo, lektor i kemi ved University of Michigan.
Ruotolo og hans team, herunder forskere fra UCLA, University of Leeds og University of Antwerp, har udviklet en ny måde at studere proteinkomplekser, der ikke involverer ødelæggelse af de intakte samlinger i processen. Deres metode er blevet offentliggjort i tidsskriftet Analytisk kemi .
"Proteiner er hoveddrivere til stort set alle kritiske cellulære processer - alt fra celledeling til celledød, "Ruotolo sagde." De dominerer også narkotikamål på grund af deres centrale betydning i forbindelse med livet, som vi kender det. Forståelse for hvordan disse proteiner virker, sammensat, på et meget grundlæggende niveau, er meget vigtig for at forstå, hvordan sygdomme virker. "
Både den traditionelle tilgang og Ruotolos metode bruger en enhed kaldet et massespektrometer, som måler vægten, eller masse, af ioniserede molekyler ved at trække molekylerne ind i et vakuum. I den traditionelle tilgang, efter at forskere har brudt proteinkomplekser ned i peptider, de bruger en teknologi kaldet elektrosprayionisering for at give disse peptider en elektrisk ladning. Massespektrometeret måler derefter massen af disse ladede peptider, og nedbryder dem yderligere ved hjælp af en baggrundsgas.
Men brug af enzymer til at fordøje proteiner gør det svært at forstå rollen som mindre kemiske enheder, der akkumuleres på proteiner, kaldet post-translationelle ændringer. Hver gang din celle udtrykker et protein, det kan også producere hundredvis af disse individuelle post-translationelle modifikationstilstande, samlet betegnet proteoformer, Siger Ruotolo.
Det er arrangementet af disse proteoformer i proteinkomplekser, der ofte bestemmer deres funktion. I traditionelle metoder til at studere proteinkomplekser, disse proteoformer går tabt.
"De vigtigste bevægere og rystere i cellen er ikke individuelle proteiner, der løber rundt og gør job-det er faktisk snesevis af proteiner, der samles for at danne supermolekylære komplekser, der udfører meget komplicerede job i cellen, "Sagde Ruotolo." Nu, den virkelige opgave er at forstå, hvordan disse store maskiner fungerer. "
Ruotolos team bruger elektrosprayionisering til at ionisere intakte proteinkomplekser. Ligesom de peptider, der typisk analyseres i proteomikundersøgelser, disse multiproteinkomplekser kan sekventeres med et massespektrometer, men det er ofte ikke muligt at sekvensere noget mere end en lille brøkdel af samlingens struktur. Ruotolos team har udviklet en kemisk modifikationsstrategi, der væsentligt forbedrer evnen til at sekvensere store, multiproteinkomplekser direkte ved massespektrometri.
"Dette er vigtigt, fordi i massespektrometeret, du har forbindelse mellem startproteoform og sekvensioner, "Sagde Ruotolo." I den enzymatiske fordøjelse, at forbindelsen er brudt. "
I sit studie, Ruotolos team udviklede deres metode ved hjælp af tre forskellige proteinkomplekser. De håber at tilpasse deres metode til at kunne studere større proteinkomplekser, mange gange størrelsen af dem i deres undersøgelse.
"Jeg tror, at vi hver dag lærer, at selv en klasse af kræftformer, som leukæmi, faktisk består af mange forskellige sygdomme, "Ruotolo sagde." De typer målinger, vi udvikler, vil kun øge vores evne til at observere denne detaljerighed og give information, der forhåbentlig kan informere evnen til at opdage nye terapier. "
Gruppen har også udgivet et papir i Analytisk kemi detaljeringssoftware, som blev udviklet i samarbejde med forskere på Institut for Computational Medicine og Bioinformatik ved U-M. Softwaren er i stand til hurtigt at fange sekvensinformation fra proteinkomplekser.
 Varme artikler
Varme artikler
-
 Sådan regnes ud af den ioniske procentdel, når du har fået forskellen mellem elektronegativitetVed ionisk binding mellem atomer tager det ene atom en elektron fra det andet og bliver negativ, mens dets partner bliver positiv. De to atomer holdes derefter sammen af deres modsatte afgifter.
Sådan regnes ud af den ioniske procentdel, når du har fået forskellen mellem elektronegativitetVed ionisk binding mellem atomer tager det ene atom en elektron fra det andet og bliver negativ, mens dets partner bliver positiv. De to atomer holdes derefter sammen af deres modsatte afgifter. -
 Fra fladskærms -tv til din smartphone - elementet bor fortjener mere opmærksomhedBor ignoreres ofte, men det har mange vigtige kvaliteter. Kredit:David Ellis/Flickr, CC BY-NC-ND Hver gang du ser sport på et fladskærms -tv, eller send en besked ved at røre ved din smartphones s
Fra fladskærms -tv til din smartphone - elementet bor fortjener mere opmærksomhedBor ignoreres ofte, men det har mange vigtige kvaliteter. Kredit:David Ellis/Flickr, CC BY-NC-ND Hver gang du ser sport på et fladskærms -tv, eller send en besked ved at røre ved din smartphones s -
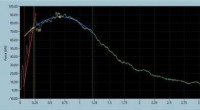 NIST-software hjælper med analysen af instrumenterede charpy-testsEt skærmbillede fra NICAS, viser en instrumenteret kraft/forskydningskurve for en fuldt duktilt prøve og dens tilsvarende karakteristiske kraftparametre (generelt udbytte, maksimal kraft, slutningen a
NIST-software hjælper med analysen af instrumenterede charpy-testsEt skærmbillede fra NICAS, viser en instrumenteret kraft/forskydningskurve for en fuldt duktilt prøve og dens tilsvarende karakteristiske kraftparametre (generelt udbytte, maksimal kraft, slutningen a -
 Undersøgelse bruger neutroner til at skinne lys på lukning af kræftcellerSt. Jude-forsker Eric Gibbs klargør prøver til neutroneksperimenter for at studere forskellige sammensætninger af en proteintumorsuppressor for bedre at forstå dens rolle i at afbøde kræftaktivitet i
Undersøgelse bruger neutroner til at skinne lys på lukning af kræftcellerSt. Jude-forsker Eric Gibbs klargør prøver til neutroneksperimenter for at studere forskellige sammensætninger af en proteintumorsuppressor for bedre at forstå dens rolle i at afbøde kræftaktivitet i
- Programmerbar dråbe manipulation med en magnetisk aktiverings robot
- Afsløring af kvantestatistik med et par fjerne atomer
- Merkurs gamle magnetfelt har sandsynligvis udviklet sig over tid
- Forskere udnytter metanforbrugende mikrober til brug i industrielle applikationer
- Arkæologer finder gammelt massebørn i Peru
- Ny undersøgelse tyder på, at der findes titusindvis af sorte huller i Mælkevejenes centrum


