Fremskridt hen imod tilslutning af en antibiotikapumpe

Susan Rempe står foran Center for Integrated Nanotechnologies, hvor noget af hendes forskning om bakteriepumper blev lavet. Hendes team fra Sandia National Laboratories og University of Illinois i Urbana-Champaign studerer pumperne for at forstå mekanismerne bag antibiotikaresistens i bakterier. Kredit:Sandia National Laboratories
Hvert år i USA mindst 23, 000 mennesker dør af infektioner forårsaget af antibiotikaresistente bakterier, ifølge Centers for Disease Control and Prevention.
Ved hjælp af computermodellering, forskere fra Sandia National Laboratories og University of Illinois i Urbana-Champaign hjælper med at udvikle midlerne til at forhindre nogle af disse dødsfald.
En måde, hvorpå bakterier udvikler resistens over for mange forskellige antibiotika, er ved at producere pumper, der spytter ukendte små molekyler ud, såsom antibiotika, før de kan gøre skade. Forskerne drillede detaljerne om, hvordan en antibiotikapumpe virker.
Det endelige mål er at udvikle nye lægemidler til at stoppe pumpen, så den ikke kan spytte antibiotika ud, måske genoprette deres effektivitet, sagde Susan Rempe, Sandia beregningsbiofysiker. Hun tilføjede, "Nu hvor vi har pumpens struktur og ved, hvordan den fungerer, forskere kan designe et molekyle, der klæber tæt til transportøren. Jeg tror, det kan lade sig gøre på kort sigt, måske fem år."
Denne forskning blev for nylig offentliggjort i Proceedings of the National Academy of Sciences .
Forfining af data for at bestemme pumpens detaljerede struktur
De specifikke pumpeforskere undersøgte, kaldet EmrE, kommer fra E coli , almindelige bakterier, der lejlighedsvis forårsager madforgiftning. Pumpen genkender og fjerner moderat olieagtig, positivt ladede små molekyler, sagde Josh Vermaas, en tidligere kandidatstuderende i Illinois, hvis arbejde med Rempe blev støttet gennem Sandias Campus Executive Program. Mange almindelige antibiotika inklusive streptomycin, doxycyclin og chloramphenicol er olieagtige og positivt ladede.
Deres første skridt var at bestemme en detaljeret struktur af pumpen. Startstrukturen af pumpen var meget ru, mangler mange af de væsentlige kemiske detaljer, og misskabe, sagde Vermaas. Rempe tilføjede, at det kan være særligt udfordrende at få gode strukturelle data fra lægemiddeltransportører som EmrE, fordi de er fleksible. Forestil dig at skulle tage et billede af et slingrende lille barn med et trægt kamera:Det resulterende billede er mere en sløring end en nøjagtig lighed.
De kombinerede eksperimentelle data fra en række almindelige biofysiske metoder såsom røntgenkrystallografi, kryo-elektronmikroskopi og elektronparamagnetisk resonansspektroskopi samt årtiers viden om de mest sandsynlige interne arrangementer af aminosyrer, byggestenene i proteiner, at producere en højopløsningsstruktur af pumpen.
"Det store gennembrud var i, hvordan vi kunne tage dårlige strukturelle data fra eksperimenter og massere dem for at komme med en forbedret struktur, vi kunne arbejde med for at forstå pumpens mekanisme, sagde Rempe.
Molekylær modellering afslører pumpens "lås" og mekanisme
Når de havde den detaljerede struktur af pumpen, det rigtige arbejde begyndte.
Først, holdet tilføjede en lipidmembran til at modellere pumpens virkelige miljø. Derefter, de kørte computersimuleringer for at se, hvordan pumpen ser ud med nul, en eller to protoner. At lukke to protoner ind er det batteri, der driver denne pumpe. De kørte simuleringer for at se overgangen fra proteinet vendt inde i bakterien til udenfor for at finde den "nemmeste" vej og dermed se, hvordan pumpen fungerer. Modellering af denne "flip" tog over 80, 000 timers computerbehandling.
De kørte også simuleringer for at se, hvordan pumpen ser ud med et eksempel på lægemiddel i den lægemiddelbindende lomme. Rempe sagde, at de fandt masser af fleksibilitet i lommen, hvor antibiotika ville binde, hvilket giver mening, da pumpen kan genkende en lang række forskellige lægemidler. De identificerede også nogle få kritiske aminosyrer, der fungerer som en lås for at sikre, at pumpen ikke slipper protonerne frit.
"Antibiotikaresistens er et vigtigt problem. 'Låsen' på pumpen er det, der får denne transportør til at tikke. Med denne viden, i fremtiden kan vi udvikle nye antibiotika, der ikke pumpes ud eller på anden måde bryder låsen i EmrE, " sagde Vermaas. "Hvis vi finder ud af at bryde pumpen, så den er ureguleret og lækker protoner, det ville være en ny måde at dræbe bakterier på."
Yderligere forskning i modvirkning af antimikrobiel resistens
Ud over hendes arbejde med antibiotikapumper, Rempe har også modelleret et protein, der transporterer miltbrandtoksiner ind i værtsceller, hvor de forårsager kaos, lader Bacillus anthracis-bakterierne trives og forårsage miltbrand. Rempe og hendes samarbejdspartnere, herunder Sandia postdoc Mangesh Chaudhari, bestemte de molekylære mekanismer for, hvordan disse toksiner trænger ind i værtscellen og udviklede en prop til at blokere denne proces. Sandia bioingeniør Anson Hatch ledede et team, der lavede og testede stikket.
I et treårigt projekt finansieret af Sandias Laboratory Directed Research &Development (LDRD) program, Rempe leder også undersøgelser af et nyt antimikrobielt middel kaldet teixobactin. Teixobactin blokerer produktionen af bakteriecellevæggen på en unik måde, som er svær for bakterier at udvikle resistens over for. Hun og hendes samarbejdspartnere fra Sandia og Illinois bruger computersimuleringer og -eksperimenter til at forstå, hvordan det antimikrobielle middel fungerer for at gøre det mere potent og bredt virkende. De offentliggjorde deres første resultater i Kemisk Videnskab , afslører to måder, hvorpå lægemidlet binder sig til specialiserede fedtmolekyler i bakterielle membraner. Bindingen hindrer opbygningen af bakteriers beskyttende cellevægge.
Selvom det er udfordrende for gram-positive bakterier, såsom Staphylococcus aureus, at ændre, hvordan de laver deres cellevæg, for at udvikle resistens over for teixobactin, det antimikrobielle stof kan stadig pumpes ud af bakterier, før det gør sin skade, gør Rempe og Vermaas' forskning for at forstå mekanismen for antibiotikapumper relevant.
Rempe sagde, "Molekylær dynamik modellering har meget høj opløsning i rum og tid, som du ikke får fra andre eksperimenter. Vi kan se dynamikken over tid i trin på en milliontedel af en milliardtedel af et sekund. Vi kan også se stumper og stykker af en proces, der ikke løses i eksperimenter, og afgøre, hvilke kemiske strukturer der bidrager til det involverede arbejde. Dette giver os en fordel i at lære, hvordan patogener fungerer, og den information kan føre til nye terapier til at imødegå disse patogener."
Sidste artikelNæste generations insektmidler til at bekæmpe myggebårne sygdomme
Næste artikelSpyt kan påvirke smagspræferencer
 Varme artikler
Varme artikler
-
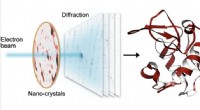 Sniger op på små krystaller med elektrondiffraktionVed seriel elektrondiffraktion, en lille elektronstråle rammer sekventielt tusindvis af små krystaller lavet af proteiner, som spreder strålen i diffraktionsmønstre. Fra disse mønstre, strukturen af
Sniger op på små krystaller med elektrondiffraktionVed seriel elektrondiffraktion, en lille elektronstråle rammer sekventielt tusindvis af små krystaller lavet af proteiner, som spreder strålen i diffraktionsmønstre. Fra disse mønstre, strukturen af -
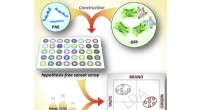 Forskere bruger en syntetisk tunge til at sortere whisky fraDette visuelle abstrakt viser et sensorsystem med tre elementer, der kan skelne alder, blandingsstatus, oprindelsesland, og smagselementer i whisky. Kredit:Jinsong Han et al./ Chem 2017 Whisky k
Forskere bruger en syntetisk tunge til at sortere whisky fraDette visuelle abstrakt viser et sensorsystem med tre elementer, der kan skelne alder, blandingsstatus, oprindelsesland, og smagselementer i whisky. Kredit:Jinsong Han et al./ Chem 2017 Whisky k -
 Gennembrud i molekylære maskinerKunstnerfortolkning af en molekylær maskine. Kredit:Mikkel Larris, Syddansk Universitet Molekyler er nogle af livets mest basale byggesten. Når de arbejder sammen på den rigtige måde, de bliver mo
Gennembrud i molekylære maskinerKunstnerfortolkning af en molekylær maskine. Kredit:Mikkel Larris, Syddansk Universitet Molekyler er nogle af livets mest basale byggesten. Når de arbejder sammen på den rigtige måde, de bliver mo -
 En ende på huller for folk med følsomme tænder?Kredit:American Chemical Society En iskold drink er forfriskende om sommeren, men for mennesker med følsomme tænder, det kan forårsage en smertefuld stød i munden. Denne tilstand kan behandles, me
En ende på huller for folk med følsomme tænder?Kredit:American Chemical Society En iskold drink er forfriskende om sommeren, men for mennesker med følsomme tænder, det kan forårsage en smertefuld stød i munden. Denne tilstand kan behandles, me
- Ny proces kan reducere energibehovet fra gødning, nitrogenbaserede kemikalier
- Ethanopbevaring ses som nøglen til revitalisering af Appalachia
- Gravityens virkninger i solsystemet
- Bevis for indrammet grafen-membranoverbygning åbner en membranspecifik lægemiddeltilførselstilsta…
- 3-D katalysatorer til forbedrede hydrazinfrie drivmidler
- Nyt videospil lærer teenagere om elektricitet


