En platform til at forberede fluorescensmærkede proteiner og simulere deres native miljø
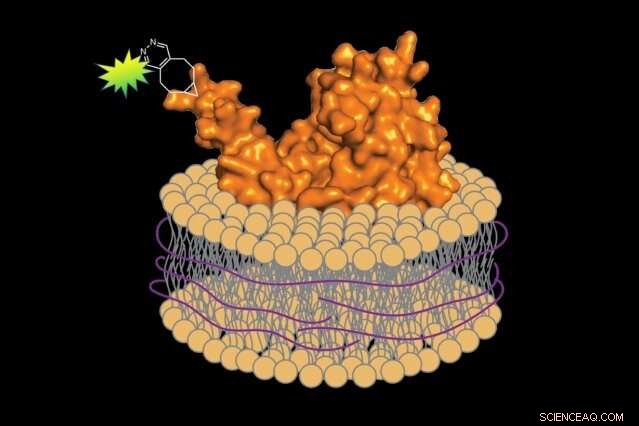
Ved at bruge en vaskemiddelfri metode, biologer kan fremstille fluorescens-mærkede proteiner sammen med et lille segment af den associerede cellemembran, bevare proteinets native miljø. Kredit:Jean-Marie Swiecicki
Alle celler har en lipidmembran, der omkranser deres indre komponenter - danner en beskyttende barriere for at kontrollere, hvad der kommer ind, og hvad der bliver ude. Proteinerne indlejret i disse membraner er essentielle for livet; de hjælper med at lette transporten af næringsstoffer, energiomdannelse og lagring, og mobilkommunikation. De er også vigtige ved sygdomme hos mennesker, og repræsenterer omkring 60 procent af godkendte lægemiddelmål. For at studere disse membranproteiner uden for cellens kompleksitet, forskere skal bruge vaskemiddel til at fjerne membranen og trække dem ud. Imidlertid, at bestemme det bedste vaskemiddel for hvert protein kan involvere omfattende forsøg og fejl. Og, fjernelse af et protein fra dets naturlige miljø risikerer at destabilisere den foldede struktur og forstyrre funktionen.
I en undersøgelse offentliggjort den 9. december i Cellekemisk biologi , forskere fra MIT udtænkte en hurtig og generaliserbar måde at udvinde, rense, og mærke membranproteiner til billeddannelse uden detergent overhovedet - medbringer en del af den omgivende membran for at beskytte proteinet og simulere dets naturlige miljø. Deres tilgang kombinerer veletablerede kemiske og biokemiske teknikker på en ny måde, effektivt at isolere proteinet, så det kan fluorescerende mærkes og undersøges under et mikroskop.
"Jeg spøger altid med, at det ikke er særlig livagtigt at studere proteiner i sæbe, " siger seniorforfatter Barbara Imperiali, professor i biologi og kemi. "Vi har skabt en arbejdsgang, der gør det muligt at afbilde membranproteiner, mens de bevarer deres oprindelige identiteter og interaktioner. Forhåbentlig vil færre mennesker nu vige tilbage fra at studere membranproteiner, givet deres betydning i mange fysiologiske processer."
Som medlem af Imperiali-laboratoriet, tidligere postdoc og hovedforfatter Jean-Marie Swiecicki undersøgte membranproteiner fra det fødevarebårne patogen Campylobacter jejuni. I dette studie, Swiecicki fokuserede på PglC og PglA, to membranproteiner, der spiller en rolle i at gøre det muligt for bakterierne at inficere menneskeceller. Hans eksperimenter krævede mærkning af PglC og PglA med fluorescerende tags for at spore dem. Imidlertid, han var ikke tilfreds med eksisterende metoder til at gøre det.
I nogle tilfælde, de fluorescerende tags, der skal inkorporeres i proteinet for at visualisere det, er for store til at blive placeret i definerede positioner. I andre tilfælde, disse tags lyser ikke klart nok, eller forstyrre proteinets struktur og funktion.
For at undgå sådanne problemer, Swiecicki besluttede at bruge en metode kendt som "unaturlig aminosyremutagenese." Aminosyrer er de enheder, der udgør proteinet, og unaturlig aminosyremutagenese involverer tilføjelse af en ny aminosyre indeholdende en konstrueret kemisk gruppe i proteinsekvensen. Denne kemiske gruppe kan derefter mærkes med et stærkt glødende mærke.
Swiecicki indsatte den genetiske kode for C. jejuni membranproteinerne i en anden bakterie, Escherichia coli. Inde i E. coli, han kunne inkorporere den unaturlige aminosyre, som kunne modificeres kemisk for at tilføje det fluorescerende mærke.
Da det blev tid til at fjerne proteinerne fra membranen, han erstattede vaskemidlet med et andet stof:en polymer af styren-maleinsyre (SMA). I modsætning til vaskemiddel, SMA pakker det ekstraherede protein og et lille segment af den tilhørende membran ind i en beskyttende skal, bevare sit oprindelige miljø. Imperiali forklarer, "Det er som et tørklæde, der beskytter din hals mod kulden."
Swiecicki kunne derefter overvåge de glødende proteiner under et mikroskop for at bekræfte, at hans teknik var selektiv nok til at isolere individuelle membranproteiner. Hele processen, han siger, tager kun et par dage, og er generelt meget hurtigere og mere pålidelig end vaskemiddelbaserede ekstraktionsmetoder, som kan tage måneder og kræver ekspertise fra højtuddannede biokemikere for at optimere.
"Jeg vil ikke sige, at det er en magisk kugle, der vil virke for hvert enkelt protein, " siger han. "Men det er et yderst effektivt værktøj, der kunne gøre det lettere at studere mange forskellige slags membranproteiner." han siger, det kan endda hjælpe med at lette lægemiddelscreeninger med høj gennemstrømning.
"Som en, der arbejder på membranproteinkomplekser, Jeg kan bekræfte det store behov for bedre metoder til at studere dem, " siger Suzanne Walker, en professor i mikrobiologi ved Harvard Medical School, som ikke var involveret i undersøgelsen. Hun håber at udvide den tilgang, der er skitseret i papiret, til de proteinkomplekser, hun undersøger i sit eget laboratorium. "Jeg satte pris på de omfattende detaljer inkluderet i teksten om, hvordan man anvender strategien med succes, " tilføjer hun.
De næste trin vil være at teste teknikken på pattedyrsproteiner, og isolering af flere proteiner på én gang i SMA-skallen for at observere deres interaktioner. Og, selvfølgelig, hver ny teknik fortjener et navn. "Vi arbejder stadig på et fængende akronym, " siger Imperiali. "Noen idéer?"
Denne historie er genudgivet med tilladelse fra MIT News (web.mit.edu/newsoffice/), et populært websted, der dækker nyheder om MIT-forskning, innovation og undervisning.
 Varme artikler
Varme artikler
-
 Sådan beregnes polymerisationsgradenEn polymerisationsgrad er en nøglekarakteristik for polymerer, der bestemmer fysiske egenskaber for polymermaterialer. Polymerer er store molekyler, der består af gentagne strukturelle (monomere) enhe
Sådan beregnes polymerisationsgradenEn polymerisationsgrad er en nøglekarakteristik for polymerer, der bestemmer fysiske egenskaber for polymermaterialer. Polymerer er store molekyler, der består af gentagne strukturelle (monomere) enhe -
 Ny forskning afslører hemmelighederne bag fotosyntesen, der kan hjælpe med at udvikle computertekn…Illustrerer, hvordan destinationen for en elektron (repræsenteret af toget) kan dirigeres efter påføring af en ultrahurtig mid-infrarød puls (repræsenteret af ilden). Kredit:Helen Towrie ved CLF F
Ny forskning afslører hemmelighederne bag fotosyntesen, der kan hjælpe med at udvikle computertekn…Illustrerer, hvordan destinationen for en elektron (repræsenteret af toget) kan dirigeres efter påføring af en ultrahurtig mid-infrarød puls (repræsenteret af ilden). Kredit:Helen Towrie ved CLF F -
 Usædvanlig halvmetal viser tegn på unikke overfladeledningstilstandeKredit:Masaki Uchida Forskere ved Tokyo Institute of Technology kontrollerer eksperimentelt eksistensen af eksotiske overfladeledningstilstande i topologiske semimetaller (TSMer), materialer, de
Usædvanlig halvmetal viser tegn på unikke overfladeledningstilstandeKredit:Masaki Uchida Forskere ved Tokyo Institute of Technology kontrollerer eksperimentelt eksistensen af eksotiske overfladeledningstilstande i topologiske semimetaller (TSMer), materialer, de -
 At bevare smagen, at reducere saltetMens mennesker har brug for saltet i snacks som kartoffelchips, Amerikanere indtager betydeligt mere salt end nødvendigt eller endda sundt. Kredit:Public Domain Forskere fra Washington State Unive
At bevare smagen, at reducere saltetMens mennesker har brug for saltet i snacks som kartoffelchips, Amerikanere indtager betydeligt mere salt end nødvendigt eller endda sundt. Kredit:Public Domain Forskere fra Washington State Unive
- Kraftfuld ny teknik bestemmer samtidig nanomaterialers kemiske makeup, topografi
- Undersøgelse viser, at det sydlige Arizona engang lignede Tibet
- Neutronstjerner kan være vores GPS til dybe rumrejser
- Rapporter:Facebook risikerer en bøde på $5B FTC, største nogensinde inden for tech
- Cryo-EM afslører kritisk protein-modificerende kompleks og potentielt lægemiddelmål
- Velhavende bliver anti-immigrant, når deres rigdomme er truet


