Maskinindlæring afslører nye kandidatmaterialer til biokompatibel elektronik
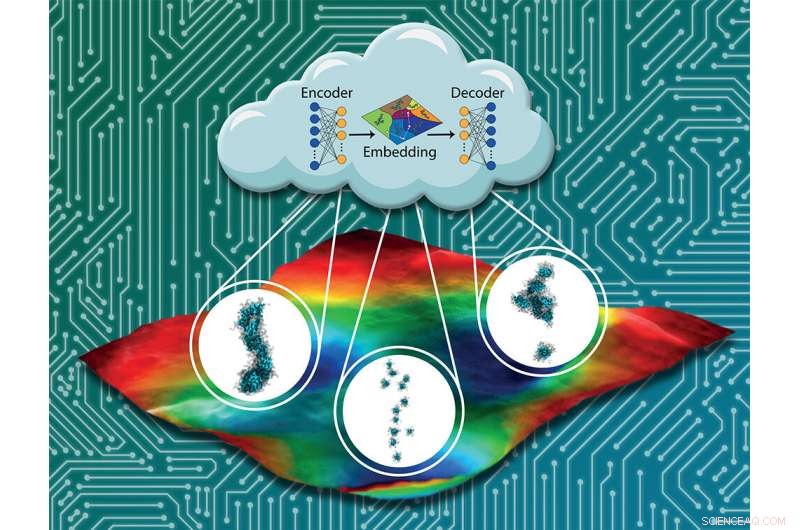
Maskinlæringsværktøjer udviklet af Assoc. Professor Andrew Ferguson og hans samarbejdspartnere er i stand til at screene selvsamlende peptider for at finde de bedste kandidater til elektronisk, biokompatible materialer. Kredit:Kirill Shmilovich et al.
Forskere og ingeniører er på jagt efter at udvikle elektroniske enheder, der er kompatible med vores kroppe:tænk på materialer, der kan hjælpe ledning af neuroner sammen igen efter hjerneskader, eller diagnostiske værktøjer, der let kan optages i kroppen.
En familie af selvsamlende peptider, kaldet π-konjugerede oligopeptider, har vist løfte om at blive grundlaget for den næste generation af disse elektroniske, biokompatible materialer. Men at identificere de rigtige molekylære sekvenser til at skabe de optimale selvsamlede nanostrukturer ville kræve afprøvning af tusindvis af muligheder, som hver især tager cirka en måned at teste i laboratoriet.
Assoc. Prof. Andrew Ferguson og hans samarbejdspartnere har fremskyndet den proces ved at udvikle maskinlæringsværktøjer, der kan screene for de bedste kandidater. Ved screening 8, 000 kandidater til selvsamlede peptider, holdet var i stand til at rangere hvert design. Det baner vejen for eksperimentalister til at teste de mest lovende kandidater.
Resultaterne blev offentliggjort i Journal of Physical Chemistry B . Papiret blev også valgt som ACS Editors 'Choice, som giver gratis offentlig adgang til ny forskning af betydning for det globale videnskabelige samfund, og blive vist på journalforsiden.
"Ved at forstå datavidenskab, materialevidenskab, og molekylær videnskab, vi var i stand til at finde en innovativ måde at screene for nye mulige kandidater, "Ferguson sagde." Det faktum, at dette papir blev valgt som et ACS Editors 'Choice, viser, at der er stor interesse i at koble kunstig intelligens til domænevidenskab. Det er et vigtigt problem, der er af bred interesse for det fysisk-kemisamfund."
Ranking af peptider for eksperimentelle
For at hjælpe med at finde de bedste kandidater, Ferguson og kandidatstuderende Kirill Shmilovich screenede en familie af π-konjugerede oligopeptider ved hjælp af maskinlæring og molekylær simulering. Sættet inkluderede 8, 000 potentielle peptider, hvis forskere beholdt den samme kerne og bare ændrede de tre aminosyrer på hver side af molekylet. (Aminosyrerne på siderne er symmetriske - hvis du skifter en på den ene side, det ændrer sig på den anden side, såvel.)
Brug af en form for maskinlæring kendt som aktiv læring eller Bayesiansk optimering til at guide molekylære simuleringer, de var i stand til at konstruere pålidelige datadrevne modeller af, hvordan sekvensen af peptidet påvirkede dets egenskaber efter kun at have overvejet 186 peptider.
Modelforudsigelserne kunne derefter ekstrapoleres pålideligt for at forudsige egenskaberne for resten af peptidfamilien. Processen fjernede også menneskelig bias fra ligningen, lade kunstig intelligens finde træk ved peptiddesign, som forskere ikke havde overvejet før, der faktisk gjorde dem til bedre kandidater.
De rangerede derefter hvert peptid og afleverede deres resultater til deres eksperimentelle samarbejdspartnere, som så tester topkandidaterne i laboratoriet. Næste, de håber at udvide deres system til at omfatte afprøvning af forskellige π-konjugerede kerner, mens de fodrer nye eksperimentelle data tilbage i sløjfen for yderligere at styrke deres modeller.
De håber også at kunne bruge dette machine learning -system til at designe proteiner, optimering af selvsamlende kolloider til fremstilling af atomkrystaller, og selv for en dag at inkorporere disse værktøjer i et selvkørende laboratorium, hvor kunstig intelligens ville tage data, skabe forudsigelser, køre eksperimenter, feed derefter disse data tilbage til modellen – alt sammen uden menneskelig indgriben.
"Dette er en metode, der kan være nyttig i mange forskellige domæner, " sagde Ferguson.
 Varme artikler
Varme artikler
-
 Syntese af UV-absorbere fra cashewnøddeskalvæskeForskere fra Mainz og Johannesburg med en prøve af udgangsmaterialet til UV-absorber fra cashewnøddeskalvæske. Kredit:Opatz Lab Forskere ved Johannes Gutenberg University Mainz (JGU) i Tyskland, i
Syntese af UV-absorbere fra cashewnøddeskalvæskeForskere fra Mainz og Johannesburg med en prøve af udgangsmaterialet til UV-absorber fra cashewnøddeskalvæske. Kredit:Opatz Lab Forskere ved Johannes Gutenberg University Mainz (JGU) i Tyskland, i -
 Sandia identificerer usædvanlige polycykliske aromatiske carbonhydrider ved hjælp af tandem masses…Sandia National Laboratories forskere Scott Skeen, venstre, Nils Hansen, centrum, og Brian Adamson diskuterer tandem -massespektrometri, som blev brugt til påvisning af alifatisk forbundne polycyklisk
Sandia identificerer usædvanlige polycykliske aromatiske carbonhydrider ved hjælp af tandem masses…Sandia National Laboratories forskere Scott Skeen, venstre, Nils Hansen, centrum, og Brian Adamson diskuterer tandem -massespektrometri, som blev brugt til påvisning af alifatisk forbundne polycyklisk -
 Det er sejt! Flash-frosne billeder afslører molekylær verdenLille er smuk:Den schweiziske videnskabsmand Jacques Dubochet giver tommelfingeren op efter at være blevet udpeget som en af tre vindere af dette års Nobelpris i kemi, præmieret for deres arbejde me
Det er sejt! Flash-frosne billeder afslører molekylær verdenLille er smuk:Den schweiziske videnskabsmand Jacques Dubochet giver tommelfingeren op efter at være blevet udpeget som en af tre vindere af dette års Nobelpris i kemi, præmieret for deres arbejde me -
 Hvad er en forbindelse?Ved du, at du bruger forbindelser hver dag uden engang at tænke over det? Alle forbindelser inkluderer en vare lavet af to eller flere genstande, der er bundet sammen for at oprette den nye vare. D
Hvad er en forbindelse?Ved du, at du bruger forbindelser hver dag uden engang at tænke over det? Alle forbindelser inkluderer en vare lavet af to eller flere genstande, der er bundet sammen for at oprette den nye vare. D
- Nanorør, der helbreder:Ingeniør bedre ortopædiske implantater
- Lovende lægemiddelkandidater til prostatacancer identificeret
- Forskere bruger diamant i verdens første kontinuerlige rumtemperatur solid-state maser
- Australiens første elaphrosaur opdaget i Victoria
- Et teoretisk boost til enheder i nano-skala
- Plastikmyten og den misforståede trekant


