Ny tilgang til metabolomics-forskning kan vise sig at ændre spillet
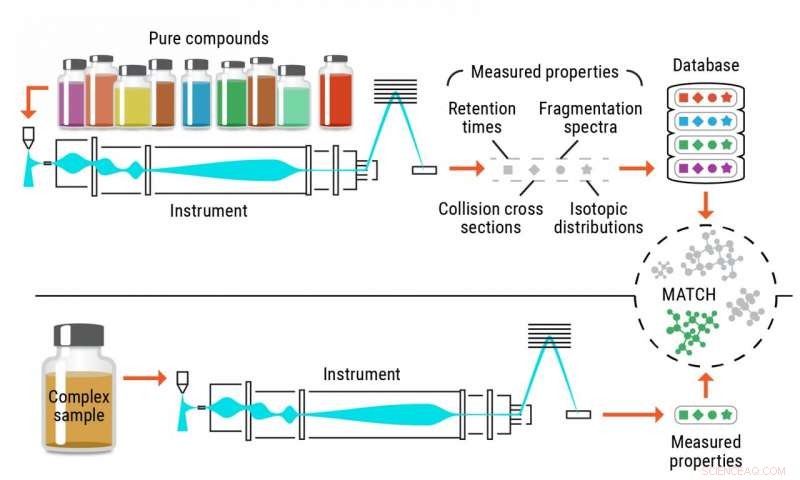
Illustration af den konventionelle identifikationsproces af metabolitter. Kredit:Pacific Northwest National Laboratory
Nøjagtig identifikation af metabolitter, og andre små kemikalier, i biologiske og miljømæssige prøver er historisk set kommet til kort ved brug af traditionelle metoder. Konventionel taktik er afhængig af rene referenceforbindelser, kaldet standarder, at genkende de samme molekyler i komplekse prøver. Disse tilgange er begrænset af tilgængeligheden af de rene kemikalier, der bruges som standarder.
"Vi ønskede virkelig at omgå det nuværende paradigme om, hvordan et metabolomisk eksperiment udføres, og hvordan molekyler med sikkerhed identificeres, " sagde Tom Metz, biomedicinsk videnskabsmand ved Pacific Northwest National Laboratory (PNNL) og direktør for Pacific Northwest Advanced Compound Identification Core.
Et problem med den nuværende metode er, at der kun er så mange rene forbindelser, forskere kan købe fra leverandører; de fleste leverandører har adgang til omkring 3, 000-4, 000 forbindelser.
"Hvis man tænker på, hvad der forventes at forekomme i naturen, du kigger på> 1030 forbindelser eller mere, der kunne være mulige, sagde Metz. når du sammenligner de få tusinde standardkemikalier, du har adgang til, med det store antal potentielle forbindelser, du er ikke engang tæt på."
Standardfri identifikationstilgang
For at løse dette problem konceptualiserede Metz og hans team hos PNNL en tilgang - standardfri metabolomics - hvormed de beregner eller forudsiger information om flere egenskaber for molekyler af interesse for at generere omfattende referencebiblioteker og derefter matche eksperimentelle data, der indeholder de samme egenskaber til disse biblioteker, muliggør sammensatte identifikation.
Ved at bruge denne nye tilgang, forskere sender kemiske strukturer gennem maskinlæring eller kvantekemi-programmer for præcist at forudsige metabolitternes eksperimentelle egenskaber.
"Hvis vi er nøjagtige nok på disse forudsigelser, så ville vi teoretisk set aldrig behøve at analysere en ren forbindelse igen, " sagde Metz. "Denne samling af værktøjer vil ændre det nuværende paradigme inden for metabolomics, og i den nærmeste fremtid vil der være nogle rigtig gode ansøgninger til at vise forskersamfundet fordelene ved denne nye tilgang."
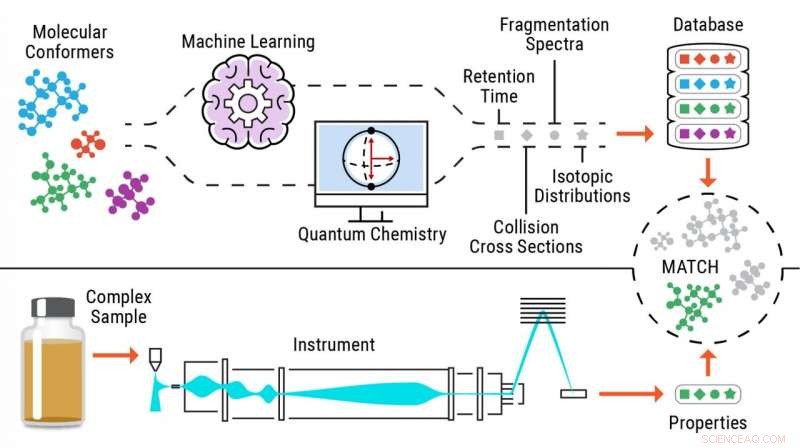
Illustration af standardfri identifikationsproces af metabolitter. Kredit:Pacific Northwest National Laboratory
Ved ikke at skulle stole på data fra analyser af rene standarder for at identificere små molekyler, den standardfri tilgang giver mulighed for identifikation af op til 90 procent flere kemikalier i prøver og gør disse beregningsværktøjer yderst nyttige i flere anvendelsesområder, herunder opdagelse af nye lægemidler, kemisk retsmedicin, og miljø- og biomedicinsk forskning.
"For eksempel, i nyt lægemiddeldesign ville en bruger være i stand til at sige, 'Jeg har et vist antal egenskaber med disse bestemte stoffer, men de er tilfældigvis giftige. Kan vi forudsige en forbindelse, der ville have lignende egenskaber, men måske ikke er giftig?'" sagde Metz. "Hvis de rigtige træningsdata kunne gives til DarkChem-programmet, DarkChem kunne derefter udføre den forudsigelse."
Tilpasset programpakke
Den nye tilgang til standardfri metabolomics-identifikation bruger fire nøgleværktøjer til at generere omfattende, i silico-afledte metabolitreferencebiblioteker, og at udtrække og matche eksperimentelle data for at give forbindelsesidentifikationer:
- I Silico Chemical Library Engine (ISiCLE), en højtydende-computervenlig, kvantekemi tilgang til generering af forudsagte kemiske egenskaber.
- DarkChem, en variationsautoencoder, der lærer en kontinuerlig numerisk eller latent repræsentation af molekylær struktur, som kan karakterisere og udvide referencebiblioteker.
- Dataekstraktion til integreret multidimensionel spektrometri (DEIMOS), et modulært softwareværktøj, der kan udtrække funktioner fra data indsamlet på multidimensionelle analytiske platforme.
- Multi Attribute Matching Engine (MAME), som matcher eksperimentelle data til referencebiblioteker baseret på forskellige kemiske egenskaber.
Værktøjerne er designet til at arbejde sammen, men de kan også bruges separat. Forskere kan tilpasse de forskellige applikationer baseret på en klients behov eller forskningsområder, skabe en fuldstændig modulær tilgang.
Fremme et forskningsfelt
Lige nu, i metabolomics samfund, alle forskere identificerer det samme sæt molekyler i hver prøve. Grunden til det er, at de alle har de samme rene forbindelser, som de købte for at bygge deres referencebiblioteker ud.
"Vores vision er, at du ved at bruge den standardfrie tilgang aldrig vil blive begrænset af de små molekyler, der kan identificeres i en prøve, " sagde Metz. "Det er virkelig en game changer for metabolomics. Og det er meget spændende at se, hvad det næste år eller deromkring har i vente."
 Varme artikler
Varme artikler
-
 Forskere opdager proces til bæredygtig fremstilling af psilocybin, en lægemiddelkandidat, der kunn…Junior Alexandra (Lexie) Adams er hovedforfatter i en publiceret artikel af deres resultater i et videnskabeligt tidsskrift. Kredit:Miami University Andrew Jones ved Miami University og hans hold
Forskere opdager proces til bæredygtig fremstilling af psilocybin, en lægemiddelkandidat, der kunn…Junior Alexandra (Lexie) Adams er hovedforfatter i en publiceret artikel af deres resultater i et videnskabeligt tidsskrift. Kredit:Miami University Andrew Jones ved Miami University og hans hold -
 Materialeforskere studerer årsagerne til slid - permanente molekylære modifikationer opstår ved f…Når safirkuglen bevæger sig hen over kobberprøven, materialet ændres permanent. Kredit:Paul Schreiber, SÆT/IAM Slid har stor indflydelse på økonomisk effektivitet og sundhed. Alle bevægelige dele
Materialeforskere studerer årsagerne til slid - permanente molekylære modifikationer opstår ved f…Når safirkuglen bevæger sig hen over kobberprøven, materialet ændres permanent. Kredit:Paul Schreiber, SÆT/IAM Slid har stor indflydelse på økonomisk effektivitet og sundhed. Alle bevægelige dele -
 Forskere afslører en køreplan for at hjælpe med udvikling af osteoporosebehandlingFra venstre mod højre:forskningsassistent Ruben Garcia-Ordonez, Forskningsassistent Jie Zheng, Professor Pat Griffin, Forskningsassistent Scott Novick, Stabsforsker Mi Ra Chang. Kredit:Scripps Researc
Forskere afslører en køreplan for at hjælpe med udvikling af osteoporosebehandlingFra venstre mod højre:forskningsassistent Ruben Garcia-Ordonez, Forskningsassistent Jie Zheng, Professor Pat Griffin, Forskningsassistent Scott Novick, Stabsforsker Mi Ra Chang. Kredit:Scripps Researc -
 Hydrofobe molekylers rolle i katalytiske reaktionerKredit:CC0 Public Domain Elektrokemiske processer kan bruges til at omdanne CO 2 til nyttige udgangsmaterialer til industrien. For at optimere processerne, kemikere forsøger i detaljer at beregn
Hydrofobe molekylers rolle i katalytiske reaktionerKredit:CC0 Public Domain Elektrokemiske processer kan bruges til at omdanne CO 2 til nyttige udgangsmaterialer til industrien. For at optimere processerne, kemikere forsøger i detaljer at beregn
- Øget global dødelighed forbundet med arseneksponering i risbaserede kostvaner
- Asteroid 2018 VP₁ kan være på vej mod Jorden. Men der er ingen grund til bekymring
- Nanomateriale forbedrer væsentligt potentiel COVID-19-terapi
- Politikker, der menes at stabilisere det finansielle system, kan faktisk gøre det modsatte, undersø…
- Venskaber har betydning for opstartssucces (og fiasko)
- Undersøgelse gør tvivl om det traditionelle syn på pterosaurflyvning


