Ny teknik udpeger placeringer af individuelle molekyler i deres cellulære kvarterer
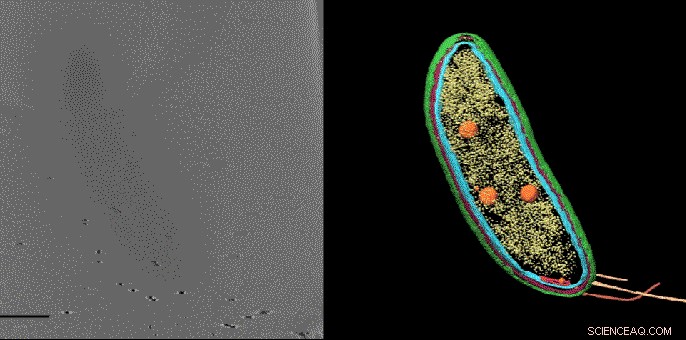
Forskere fra Stanford University troede, at et tilsyneladende tomt område i den ene ende af Caulobacter-bakterien kunne indeholde to proteiner involveret i celledeling. Ved at mærke proteinerne med fluorescerende tags og derefter afbilde de samme prøver med kryoelektrontomografi, de var i stand til at bekræfte denne placering og vise præcis, hvordan proteinerne var arrangeret. Kredit:Peter Dahlberg et al., PNAS , 8. juni 2020
Forskere har giftet sig med to af nutidens mest kraftfulde mikroskopiteknikker for at lave billeder, der præciserer, for første gang, identiteter og præcise placeringer af individuelle proteiner inden for den detaljerede kontekst af bakterieceller. Denne information er afgørende for at lære, hvordan proteinmolekyler arbejder sammen for at organisere celledeling og udføre andre vigtige opgaver, som at sætte mikrober i stand til at opsnuse mad og farer.
Den nye metode har allerede fundet ny information om bakterielle proteiner og deres nærliggende cellulære kvarterer. Forskere siger, at det også har potentiale til at besvare grundlæggende spørgsmål om virussers molekylære maskineri, parasitter, og processer som fotosyntese.
"Dette er et stort spring for biologi, og jeg tror der er mange, mange systemer, der vil drage fordel af denne form for billedbehandling, " sagde Stanford Professor Lucy Shapiro, hvis forskergruppe deltog i undersøgelsen.
Den nye hybridmetode, kaldet korreleret billeddannelse ved annotering med enkelte molekyler, eller CIASM (udtales "kløft"), blev udviklet af Peter Dahlberg, en postdoc-forsker i laboratoriet hos professor W. E. Moerner ved Stanford University.
Det er en variation af en teknik kaldet lavtemperatur enkeltmolekylemikroskopi, opfundet af Moerner for tre årtier siden, som sætter glødende mærker til molekyler, så de kan identificeres individuelt. Denne metode ligger til grund for superopløsningsfluorescensmikroskopi, emnet for Moerners Nobelpris i kemi i 2014.
Det, Dahlberg gjorde, var at finde en måde at få denne type fluorescensbilleddannelse til at fungere ved temperaturer under frysepunktet, så de samme prøver også kunne undersøges med kryogen elektrontomografi (CET). CET bruger strømme af elektroner til at lave 3D-billeder af flash-frosne celler og deres komponenter ved næsten atomær opløsning. Ved at kombinere CET med fluorescerende billeddannelse tillader videnskabsmænd at se de mærkede molekyler i sammenhæng med den omgivende celle, et afgørende perspektiv for at forstå deres rolle i det cellulære maskineri.
"Vi kan mærke specifikke molekyler af interesse, så det lys, vi ser, kun kommer fra disse molekyler, og så finder vi, hvor de er inden for omkring 10 nanometer, eller milliardtedele af en meter. Dette giver os et meget mere præcist billede af, hvad der foregår, " sagde Dahlberg. "Vi har taget de ultrapræcise snapshots leveret af CET og tilføjet en lille smule farve."
Han tilføjede, "Det er spændende at udvikle nye billeddannelsesmetoder. Når du er færdig, du kommer til at tage et skridt tilbage og se på alle de nye spørgsmål, du kan angribe."
med CIASM, forskerholdet var i stand til at udpege placeringen af tre typer proteiner i højopløselige CET-billeder af bakterier taget ved Department of Energy's SLAC National Accelerator Laboratory. Resultaterne blev rapporteret i Proceedings of the National Academy of Sciences i dag.
"Enhver metode har sine fordele og ulemper, " sagde Mørner, "og det er en god situation, hvor vi kan kombinere to metoder for at lære mere."
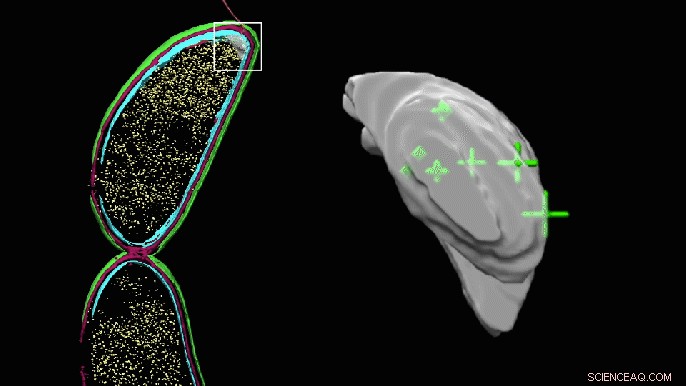
Et roterende 3D-billede af den tilsyneladende tomme lomme i den ene ende af en Caulobacter-celle viser nu de præcise placeringer af PopZ-molekyler. Lommen ser klumpet ud, fordi den er blevet manuelt farvet for at fremhæve det område, hvor forskerne troede, at molekylerne kunne være, men kunne ikke identificere direkte i kryoelektrontomografi. Kredit:Peter Dahlberg et al., PNAS , 8. juni 2020
At finde orden i en cellulær suppe
Selv i relativt simple bakterieceller, beliggenhed er alt, sagde Saumya Saurabh, en postdoc-forsker i Shapiros laboratorium, der spillede en ledende rolle i forskningen.
"Folk har en tendens til at tænke på bakterier som sække med proteiner uden organisation, " sagde han. "Men det viser sig, at det ikke er sandt, og faktisk er mange af molekylerne i bakterier netop placeret i både rum og tid. Hvis de ikke er i den rigtige position, cellen dør. Det, som Petes arbejde endelig giver os mulighed for, er at se ind med molekylær opløsning og finde ud af, hvornår og hvor disse molekyler er placeret i forhold til hinanden."
Caulobacter crescentus, for eksempel, en velundersøgt art af ferskvandsbakterier, er kendt for at dele sig i to meget forskellige typer datterceller:Den ene svømmer frit, mens den anden danner en stilk og fæstner sig til en overflade. Hvordan hver dattercelle får det, den skal bruge for at følge sin unikke vej, har været et langvarigt mysterium.
Forskere havde tidligere identificeret små områder i hver ende af den delende celle, som kunne indeholde proteiner, der spiller nøgleroller i denne skæve celledeling. Et af proteinerne, PopZ, findes i begge ender af den delende celle, mens den anden, SpmX ("Spam-X") findes kun i den halvdel, der vil udvikle en stilk.
Til denne undersøgelse, Saurabh og kandidatstuderende Jiarui Wang mærkede proteiner i Caulobacter med fluorescerende tags. Så frøs Dahlberg disse prøver ned, udførte enkeltmolekyle fluorescensbilleddannelse på dem med hjælp fra kandidatstuderende Annina Sartor, og tog dem til Stanford-SLAC Cryo-EM faciliteterne til CET-billeddannelse instrueret af Wah Chiu, en professor ved Stanford og SLAC.
Kortlægning af et protein-hangout
De kombinerede billeder bekræftede ikke kun, at begge proteiner var i de områder, forskerne havde mistænkt, men afslørede også præcis, hvordan de var arrangeret:SpmX var indlejret i cellens indre membran og stak ud i cellens indre, hvor den kom i direkte kontakt med PopZ.
"Den nøjagtige orientering af dette proteinkompleks er blevet diskuteret i løbet af de sidste 12 år, " sagde Saurabh. "Vi var i stand til at observere proteinpartnerne med udsøgt opløsning. Nu har vi et meget præcist billede af, hvordan disse proteiner taler med hinanden i cellen."
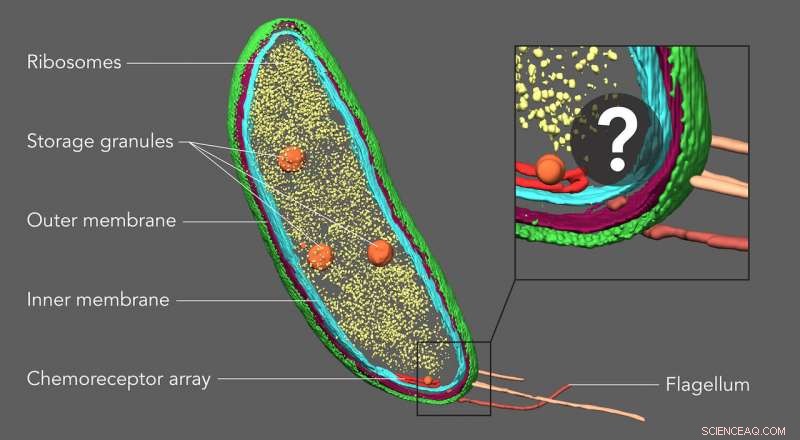
Med en teknik kaldet kryoelektrontomografi, videnskabsmænd kan skabe detaljerede 3D-billeder af celler, såsom denne Caulobacter-bakterie, og fremhæve deres komponenter - i dette tilfælde, cellemembranerne (røde og blå), proteinskal (grøn), proteinfabrikker kendt som ribosomer (gul) og opbevaringsgranulat (orange). Men indtil nu, mindre strukturer og individuelle molekyler kunne ikke identificeres og lokaliseres præcist i disse billeder. En ny billedbehandlingsteknik udviklet på Stanford udfylder dette hul, afslører små molekyler, der ikke er synlige her. Kredit:Peter Dahlberg et al., PNAS , 8. juni 2020
Holdet testede nøjagtigheden af CIASM ved at bruge det til at bekræfte placeringen af et protein kaldet McpA, der var kendt for at være en del af et kemoreceptorarray i bakterierne. "Udsøgt følsomme proteiner i dette array tjener som Caulobacters næse, Saurabh sagde, "fornemmer kemien i det omgivende miljø, så de kan bevæge sig væk fra ubehagelige ting og bevæge sig mod den glukose, de spiser."
Arrayet vises som parallelle sorte linjer i CET-billeder, og fluorescerende tagging af de samme billeder udpegede placeringerne af individuelle McpA-proteiner inden for omkring 10 nanometer.
Et detaljeret kig på kvanteprikker
I en separat, parallel undersøgelse, udgivet 24. april i Angewandte Chemie , forskerne brugte en lignende teknik til at se på enkelte kvanteprikker, med nogle overraskende resultater.
Kvanteprikker er krystaller i nanoskala af halvledermateriale, der naturligt fluorescerer i farver bestemt af deres størrelse, form og sammensætning. Disse prikker bruges i forskning til at mærke og spore proteiner og andre biologiske materialer, og har potentielle anvendelser i fremtidens elektronik, belysning, kvanteberegning, medicinsk billeddiagnostik og andre områder.
I dette studie, Målet var at se, hvordan de finere strukturelle detaljer af individuelle prikker var relateret til specifikke detaljer om deres optiske egenskaber, sagde Davis Perez, en ph.d. elev i Mørners laboratorium.
"Vi var i stand til at se nogle overraskende adfærd af de individuelle kvanteprikker - for eksempel, i deres reaktion på excitation med laserlys, " sagde han. "Men det mest spændende aspekt for mig er, at den metode, vi udviklede til at studere kvanteprikker, også kan bruges til at studere biologiske systemer såsom fotosyntetiske proteiner, hvor energi overføres mellem grupper af proteiner, og se, hvordan fotosyntesemaskineriet fungerer."
Moerner sagde, at hans laboratorium arbejder sammen med Chiu for at forfølge disse udfordringer.
"Det er de tidlige dage med at kombinere de to metoder, og vi er spændte på at udforske flere samarbejder, der forbinder lys og elektroner, " sagde Chiu. "Denne hybride billeddannelsestilgang har potentialet til at afdække strukturer af molekylære komponenter involveret i vigtige biologiske processer i celler, der spænder over alle livets domæner."
Sidste artikelCOVID-19 antistoftest:Hvor pålidelige er de?
Næste artikelForholdet mellem sart suturstruktur og dæmpning af biomaterialer
 Varme artikler
Varme artikler
-
 Sådan konverteres grader Brix til sukkerGrader Brix er et vildledende udtryk, da grader i en videnskabelig sammenhæng normalt refererer til enten temperaturniveauer eller geometriske vinkler. Grad i denne forstand beskriver massefraktionen
Sådan konverteres grader Brix til sukkerGrader Brix er et vildledende udtryk, da grader i en videnskabelig sammenhæng normalt refererer til enten temperaturniveauer eller geometriske vinkler. Grad i denne forstand beskriver massefraktionen -
 Forskere løser et videnskabeligt mysterium om fordampningHadi Ghasemi, Cullen lektor i maskinteknik ved University of Houston, ledet forskning, der eliminerer flaskehalsen, der har komplicerede forudsigelser og simuleringer af processer, der involverer ford
Forskere løser et videnskabeligt mysterium om fordampningHadi Ghasemi, Cullen lektor i maskinteknik ved University of Houston, ledet forskning, der eliminerer flaskehalsen, der har komplicerede forudsigelser og simuleringer af processer, der involverer ford -
 Forstå sprøde revner for at designe stærkere materialerKredit:CC0 Public Domain I et papir udgivet i Naturfysik , Northeastern University Department of Physics Arts and Sciences Distinguished Professor Alain Karma, i samarbejde med hans postdoktoral
Forstå sprøde revner for at designe stærkere materialerKredit:CC0 Public Domain I et papir udgivet i Naturfysik , Northeastern University Department of Physics Arts and Sciences Distinguished Professor Alain Karma, i samarbejde med hans postdoktoral -
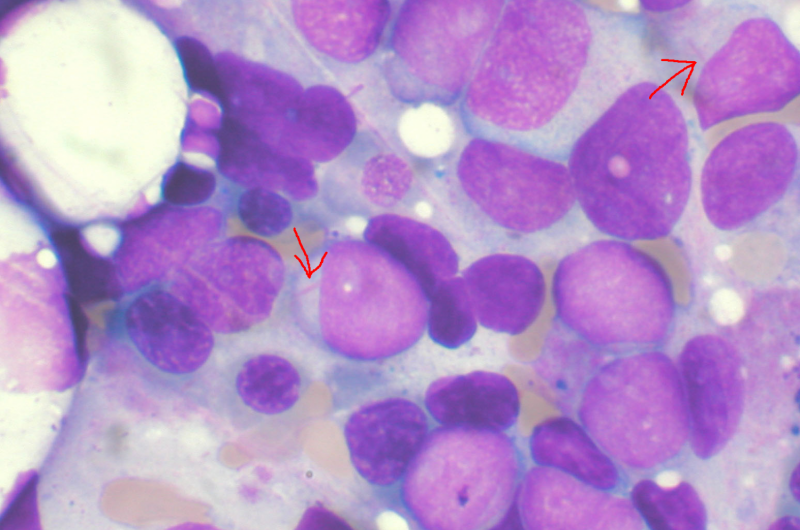 Forskning baner vej for en ny kilde til leukæmimedicinKnoglemarvsaspirat, der viser akut myeloid leukæmi. Flere blaster har Auer -stænger. Kredit:Wikipedia Kemiforskere ved Oregon State University har patenteret en metode til fremstilling af antilukæ
Forskning baner vej for en ny kilde til leukæmimedicinKnoglemarvsaspirat, der viser akut myeloid leukæmi. Flere blaster har Auer -stænger. Kredit:Wikipedia Kemiforskere ved Oregon State University har patenteret en metode til fremstilling af antilukæ
- Intensiv forvaltning af afgrøder og husdyr ansporede La Bastidas økonomiske udvikling
- Forskere opretter genopladelige svømmemikrorobotter ved hjælp af olie og vand
- Hvordan fungerer et speedometer i et fly?
- Fleksible batterier driver fremtiden for bærbar teknologi
- Tønder med gammel antarktisk luft sigter mod at spore historien om sjælden gas
- Mad på Mars, mad på Jorden:NASA tapper USU -forskere til rumsøgning


