Forskere præsenterer en ny tilgang til overvågning af ferskvands sundhed

MinION er en bærbar enhed, der kan sekvensere genetisk materiale, der er taget fra miljøet - her, fra River Cam i Cambridge, Storbritannien. Kredit:Team PuntSeq (CC BY 4.0)
Forskere har brugt verdens mindste, smartphone-størrelse DNA-sekventeringsenhed til at overvåge hundredvis af forskellige bakterier i et flodøkosystem.
Skriver i journalen eLife , det tværfaglige team fra University of Cambridge, Storbritannien, give praktiske og analytiske retningslinjer for brug af enheden, kaldet MinION (fra Oxford Nanopore Technologies), at overvåge ferskvands sundhed. Deres retningslinjer lover en betydeligt mere omkostningseffektiv og enkel tilgang til dette arbejde uden for laboratoriet, sammenlignet med eksisterende metoder.
Roere og svømmere i Cambridge påvirkes regelmæssigt af vandbårne infektioner som Weils sygdom, nogle gange fører til offentlig lukning af byens ikoniske vandveje. Overvågning af de mikrobielle arter i ferskvand kan hjælpe med at indikere tilstedeværelsen af sygdomsfremkaldende mikroorganismer og endda vandforurening. Men traditionelle test for ferskvandsbakterier kræver ofte veludstyrede laboratorier og komplekse metoder til dyrkning af kolonier af individuelle bakteriearter.
"Den direkte måling af alle bakterielle DNA -spor i ferskvand, en metode kendt som metagenomics, er et værdifuldt alternativ, men kræver stadig store, dyrt udstyr, der kan være svært at betjene, "siger Andre Holzer, medforfatter og ph.d. studerende ved Institut for Plantevidenskab, University of Cambridge. "Vi havde til formål at beskrive bakteriearterne i River Cam ved hjælp af den nye bærbare DNA -sekventeringsteknologi."
Teamet brugte MinION -enheden til at sekvensere DNA'et for hele grupper af mikroorganismer fundet i vandprøver fra River Cam. Men før de kunne bruge sekvensdataene, de havde brug for at optimere deres eksperimentelle metoder og analysesoftware. "Det var vigtigt at redegøre for kvaliteten af denne nye type bakteriel DNA -sekvensinformation, "Holzer forklarer." Vi testede mange forskellige algoritmer til behandling af dataene for at finde de mest nøjagtige metoder. "
Forskerne brugte derefter deres optimerede retningslinjer til at analysere dataene og med succes måle andelen af hundredvis af forskellige bakteriearter til stede i vandet. De tog prøver fra ni forskellige steder langs floden, prøver ofte steder på steder på tre forskellige tidspunkter, så de kan sammenligne andelen af arter forskellige steder og årstider.
Holdet var også i stand til at skelne nært beslægtede, skadelige mikrobielle arter fra ikke-skadelige. Ved at sammenligne prøverne fra forskellige steder, de fandt ud af, at der var flere potentielt skadelige bakterier og dem, der er forbundet med spildevand nedstrøms for de mest bebyggede, byområder ved floden. Kemiske opfølgningsanalyser af vandprøverne fra de samme byområder afslørede en matchende tendens til stigende spildevandsforurening i disse områder.
"Vores arbejde viser, hvordan MinION og den tilhørende DNA -sekventeringsteknologi kan bruges til effektiv overvågning af ferskvands sundhed, "siger Lara Urban, medlederforsker af arbejdet og nu en Alexander von Humboldt Research Fellow ved University of Otago, New Zealand. "Dette udvider teknologiens eksisterende applikationer, som omfatter nøjagtig sporing af virale transmissioner mellem patienter under den seneste ebola, Zika og SARS-CoV-19 virusudbrud. "
"Vi håber, at vores resultater vil tilskynde andre uafhængige forskere og kollektiver til at deltage i forenklet ferskvandsforvaltning og biodiversitetstest rundt om i verden, "slutter seniorforfatter Maximilian Stammnitz, en ph.d. studerende på Institut for Veterinærmedicin, University of Cambridge.
 Varme artikler
Varme artikler
-
 Synkrotronundersøgelse afslører oxygens indflydelse på kemien af atmosfærisk forureningEt repræsentativt brændstofmolekyle, der viser iltmolekyler (højre, rød) og 2-methylheptan (højre, grå og hvid) mødes i en jet-omrørt reaktor. De stærkt oxygenerede molekyler, der resulterer (til vens
Synkrotronundersøgelse afslører oxygens indflydelse på kemien af atmosfærisk forureningEt repræsentativt brændstofmolekyle, der viser iltmolekyler (højre, rød) og 2-methylheptan (højre, grå og hvid) mødes i en jet-omrørt reaktor. De stærkt oxygenerede molekyler, der resulterer (til vens -
 Sådan konverteres ledningsevne til koncentrationKonduktivitet måler hvor godt elektrisk strøm passerer gennem en opløsning og er direkte relateret til ionkoncentration. Jo højere ionkoncentration i din opløsning, jo bedre leder den elektricitet. Br
Sådan konverteres ledningsevne til koncentrationKonduktivitet måler hvor godt elektrisk strøm passerer gennem en opløsning og er direkte relateret til ionkoncentration. Jo højere ionkoncentration i din opløsning, jo bedre leder den elektricitet. Br -
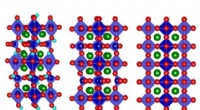 Elektrisk varmeventil:Strontium cobaltoxid tynd film ændrer termiske egenskaber med påført spænd…Forskere fandt ud af, at selen cobaltoxid (SCO) naturligt forekommer i en atomart konfiguration kaldet brownmillerite (midten), men når oxygenioner tilsættes (til højre), det bliver mere velordnet og
Elektrisk varmeventil:Strontium cobaltoxid tynd film ændrer termiske egenskaber med påført spænd…Forskere fandt ud af, at selen cobaltoxid (SCO) naturligt forekommer i en atomart konfiguration kaldet brownmillerite (midten), men når oxygenioner tilsættes (til højre), det bliver mere velordnet og -
 Raffineret finish til fin fiskeolieProfessor Colin Raston ved Flinders University. Kredit:Flinders University Ikke alle fiskeolier er olier af høj kvalitet, så videnskabsmænd har udviklet en overlegen metode til at hjælpe med at pr
Raffineret finish til fin fiskeolieProfessor Colin Raston ved Flinders University. Kredit:Flinders University Ikke alle fiskeolier er olier af høj kvalitet, så videnskabsmænd har udviklet en overlegen metode til at hjælpe med at pr
- On-chip lægemiddelscreening til identifikation af antibiotikainteraktioner på otte timer
- Undersøgelse viser seks årtiers forandring i planktonsamfund
- Verdens hurtigste kamera fryser tiden med 10 billioner billeder i sekundet
- Hvilken enhed opfandt Douglas Engelbart?
- Google Home Hub vs. Amazon vs. Facebook:Sådan sammenlignes videoenhederne
- Indsatsen for virksomhedernes sociale ansvar kan give bagslag for nye brands


