Supercomputersimuleringer afslører detaljerne om coronavirusfusion
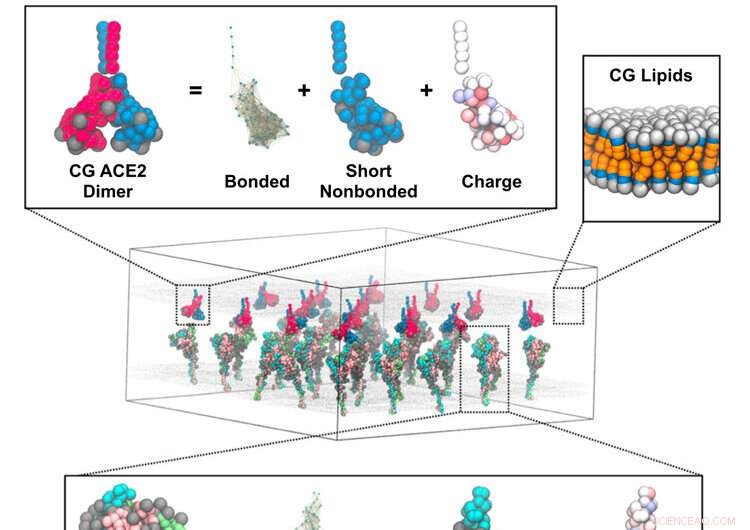
Mekanismen, hvorved coronavirus smelter sammen med værtsceller, er blevet foreslået gennem simuleringer af forskere fra University of Chicago, der bruger Frontera-supercomputeren ved TACC. Repræsentativ afbildning af en kurs-kornet (CG) simulering af spidstrimere i membran, der interagerer med en tilstødende membran med ACE2-dimerer. Indsætningerne viser CG-modelkomponenterne for spidstrimeren (nederst), ACE2-dimeren (øverst til venstre) og lipidmembranen (øverst til højre). Kredit:Pak, A.J., Yu, A., Ke, Z. et al.
Mysteriet om præcis, hvordan SARS-CoV-2-virus inficerer menneskelige lungeceller, forbliver stort set skjult for eksperimentelle videnskabsmænd. Men nu er de djævelske detaljer om mekanismen, hvorved coronavirus smelter sammen med værtsceller, blevet foreslået gennem simuleringer af forskere fra University of Chicago, der bruger Frontera-supercomputeren ved Texas Advanced Computing Center (TACC).
Computermodellerne viser værtscellereceptorproteinernes samarbejdsadfærd, der fører til deres egen infektion. Arbejdet kan bruges til at hjælpe med at forstå den øgede virulens af coronavirus-varianter såsom delta, omicron og mere.
"Vi opdagede, at spidsproteinet interagerer med to ACE2-receptorer på en meget samarbejdsvillig måde," sagde Gregory Voth, en fremtrædende professor i kemi ved University of Chicago. "Dette er en grundlæggende biofysisk indsigt."
Voth er seniorforfatter på undersøgelsen, der modellerede coronavirus og receptorcelleinteraktioner med computersimuleringer offentliggjort i tidsskriftet Nature Communications i februar 2022.
Som en fodbold med pigge pryder piggeproteinerne overfladen af coronavirus. Piggene opsøger og smelter sammen med de angiotensin-konverterende enzym 2 (ACE2) proteinreceptorer i humane lungeceller. Spikeproteinet er sammensat af to hoveddele. S1-domænet indeholder det receptorbindingsdomæne, der genkender ACE2-proteiner. Og S2-domænet indeholder fusionsmaskineriet, som er beskyttet og dækket som et husk af S1-domænet.
Simuleringerne afslører, hvordan et ACE2-receptorprotein holder på coronavirus-spidsen og svækker det, mens det andet begynder at trække det fra hinanden. S1-domænet falder derefter fra hinanden og afslører fusionsmaskineriet. Denne "en-to" punch primer virussen til fusion og indtræden i humane lungeværtsceller.
"Det ser ud til, at varianter som delta og omicron kan accentuere denne adfærd endnu mere - det er et nøgletrin. I sidste ende bør fremtidige antistoffer og muligvis molekylære lægemidler være i stand til at forstyrre denne proces," sagde Voth.
Voth og kolleger udviklede, hvad de kalder "bottom-up grovkornede modeller", der tog data fra kryo-elektrontomografi fra laboratoriet af undersøgelsens medforfatter John Briggs fra Max Planck Institute of Biochemistry. De kombinerede det med simuleringer af atomistiske molekylære dynamik. De genererede data ledte ind i en teoretisk ramme, der udviklede de grovkornede modeller.
"De grovkornede modeller er op til 1.000 gange hurtigere end simple simuleringer af atomistisk molekylær dynamik, men de bevarer de væsentlige fysiske egenskaber," sagde Voth. Denne metode giver en enorm besparelse i tid og penge på beregningerne.
Videnskabsholdet blev tildelt supercomputerressourcer og -tjenester af COVID-19 HPC Consortium, en offentlig-privat indsats til støtte for COVID-19-forskning. Gennem konsortiet brugte de det National Science Foundation-finansierede Frontera-system hos TACC; Witherspoon computerklyngen hos IBM Research; og ressourcer fra Oak Ridge Leadership Computing Facility på Oak Ridge National Laboratory.
"Vi beregnede all-atom molekylær dynamik data på Frontera og brugte analyseværktøjer tilgængelige fra TACC - begge var meget værdifulde," sagde Voth.
Voths hold indsendte deres papir, før delta- og omicron-varianterne var kendt, og forudsagde derfor ikke mutationerne. Men de gik tilbage og reviderede modellerne for at undersøge varianterne.
"Delta har noget som en åbning i spidsen, der sker lettere end i tidligere coronavirus-mutationer," sagde Voth. "Det føltes spændende fra et videnskabeligt synspunkt at se adfærd, der ikke var set før."
Voth henviste til laboratoriedata fra kryo-elektronmikroskopi, der viser strukturen af et opløseligt spidsprotein med to ACE2-receptorer bundet til det. Men han adskilte dette krystalliserede eksempel fra det, han undersøgte ved hjælp af simuleringer i det mere realistiske miljø af mange proteiner, der interagerer med hinanden på membranplader.
"Supercomputere, hvis de bruges godt og er baseret på god fysik, kan give en helt ny måde at se disse processer på. Gennem computersimulering kan man studere ting, som i øjeblikket ikke kan lade sig gøre med eksperimenter. Simulering og eksperimenter fungerer meget godt sammen, hånd i hånd," sagde Voth. + Udforsk yderligere
Første komplette coronavirus-model viser samarbejde
 Varme artikler
Varme artikler
-
 Celleadfærd, engang indhyllet i mystik, afsløres i nyt lysGavin King og et team af forskere ved University of Missouri er et skridt tættere på at forstå celleadfærd ved hjælp af et specialiseret mikroskop. Kredit:University of Missouri-Columbia En celles
Celleadfærd, engang indhyllet i mystik, afsløres i nyt lysGavin King og et team af forskere ved University of Missouri er et skridt tættere på at forstå celleadfærd ved hjælp af et specialiseret mikroskop. Kredit:University of Missouri-Columbia En celles -
 Materialebehandlingstricks gør det muligt for ingeniører at skabe nyt lasermaterialeVed at dope aluminiumoxidkrystaller med neodymioner, ingeniører ved University of California San Diego har udviklet et nyt lasermateriale, der er i stand til at udsende ultrakort, højeffektimpulser-en
Materialebehandlingstricks gør det muligt for ingeniører at skabe nyt lasermaterialeVed at dope aluminiumoxidkrystaller med neodymioner, ingeniører ved University of California San Diego har udviklet et nyt lasermateriale, der er i stand til at udsende ultrakort, højeffektimpulser-en -
 Pandemi giver en unik mulighed for atmosfæriske kemikereKredit:CC0 Public Domain Da COVID-19-pandemien bremsede rejser og forretninger rundt om i verden, forureningsemissionsraterne faldt som reaktion. Med færre biler på vejen og klarere himmel, atmosf
Pandemi giver en unik mulighed for atmosfæriske kemikereKredit:CC0 Public Domain Da COVID-19-pandemien bremsede rejser og forretninger rundt om i verden, forureningsemissionsraterne faldt som reaktion. Med færre biler på vejen og klarere himmel, atmosf -
 Sådan sammenlignes størrelsen på et AtomNår man sammenligner atomer med større objekter - med en stor forskel i størrelse - viser størrelsesordrer, hvordan man kvantificerer størrelsesforskellen. Størrelsesordrer giver dig mulighed for at s
Sådan sammenlignes størrelsen på et AtomNår man sammenligner atomer med større objekter - med en stor forskel i størrelse - viser størrelsesordrer, hvordan man kvantificerer størrelsesforskellen. Størrelsesordrer giver dig mulighed for at s
- Små højdeændringer kan reducere indflydelse på flyvninger med op til 59 procent
- Studiet sætter de første germanium-baserede begrænsninger på mørkt stof
- 3 enkle videnskabshacks til at gøre din Thanksgiving-måltid Tastier
- Verdens mindste propel kunne bruges til mikroskopisk medicin
- Hvordan kontrollerer jeg lysniveauet for en LED?
- Hvad er forskellen mellem en løsning og en suspension?


